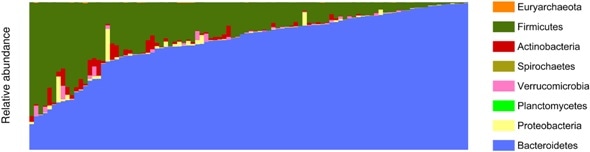
 Stablet stregdiagram over fylogenetisk sammensætning af alle mikrobiometaxaer i denne undersøgelse kollapsede på phyla -niveau i fækale prøver. Grønne søjler repræsenterer Firmicutes og de blå repræsenterer Bacteroidetes, de to mest udbredte bakteriefamilier. Af æstetiske formål er prøverne (n =98, bund) blev sorteret efter deres sammensætning af Bacteroidetes og Firmicutes for at demonstrere, hvordan baseline -tarmmikrobiomresultaterne fra denne undersøgelse kunne bruges i forbindelse med resultater fra tidligere undersøgelser. Kredit:King et al, 2019
Stablet stregdiagram over fylogenetisk sammensætning af alle mikrobiometaxaer i denne undersøgelse kollapsede på phyla -niveau i fækale prøver. Grønne søjler repræsenterer Firmicutes og de blå repræsenterer Bacteroidetes, de to mest udbredte bakteriefamilier. Af æstetiske formål er prøverne (n =98, bund) blev sorteret efter deres sammensætning af Bacteroidetes og Firmicutes for at demonstrere, hvordan baseline -tarmmikrobiomresultaterne fra denne undersøgelse kunne bruges i forbindelse med resultater fra tidligere undersøgelser. Kredit:King et al, 2019 Selvom forskningsinteressen for tarmmikrobiomets betydning for den generelle sundhed fortsat vokser, der er i øjeblikket ingen omfattende tarmmikrobiom -referenceliste tilgængelig for forskere og patienter. I dette studie, King og kolleger begynder at katalogisere den organismeriske sammensætning af sunde menneskelige tarmmikrobiomer, og udviklede en prototype rapporteringsskabelon til klinikere til at videresende resultater til patienter.
For at kompilere deres database, forfatterne genetisk sekventerede 48 fækale prøver fra seksten raske deltagere rekrutteret fra George Washington University campus i Washington, D.C., ud over at bruge 50 fækale metagenomiske prøver downloadet fra Human Microbiome Project fra personer screenet som "sunde".
Efter at have analyseret alle prøvernes genomiske og metagenomiske sekvenser ved hjælp af en ny software-baseret arbejdsgang, King og kolleger udarbejdede en indledende database med bekræftede mikrober og deres relative mængde på tværs af alle prøver, GutFeelingKB, ved hjælp af NCBIs omfattende og offentligt tilgængelige genetiske oplysninger til at levere metadata om de beskrevne organismer.
GutFeelingKB beskriver 157 organismer (155 bakterier og to arkealiske organismer) på tværs af 60 forskellige slægter. Det største antal bakterier repræsenteret var Firmicutes (40 procent af alle organismer på listen), som igen bestod af 20 procent Clostridia, 19 procent Bakterioidia, 17 procent Bifidobacteriales, 14 procent Enterobacterales, og 14 procent Lactobacillales -bakterier - bakterieklasser, der også findes i yoghurt og andre probiotiske fødevarer. Forfatterne bemærker også, at 84 organismer var fælles for alle prøverne, muligvis angiver, at disse kan være kernearter for den menneskelige tarm.
Denne undersøgelse rekrutterede kun seksten deltagere, en lille prøve. Men mens yderligere undersøgelser fortsat kan identificere yderligere organismer, der findes i sunde tarm fra hele verden, GutFeelingKB er et vigtigt første skridt. Databasen kunne fungere som udgangspunkt for komparativ analyse af prøver og udvikling af fremtidige patientmikrobiombehandlinger.
Forfatterne tilføjer:
Vores mål er at kortlægge det sunde tarmmikrobiom, så vi og andre forskere kan bruge vores data til at udvikle sygdomsspecifikke forudsigelsesmodeller. "
 Menneskelige coronavirus har brug for organiske materialer for effektivt at overføre mellem overflader
Menneskelige coronavirus har brug for organiske materialer for effektivt at overføre mellem overflader
 Hvad kan gammel afføring fortælle os om udviklingen af det menneskelige tarmmikrobiom?
Hvad kan gammel afføring fortælle os om udviklingen af det menneskelige tarmmikrobiom?
 Forståelse af halsbrand
Forståelse af halsbrand
 Pankreatitis
Pankreatitis
 Probiotika kan hjælpe med at bremse underernæring i løbet af de næste to årtier,
Probiotika kan hjælpe med at bremse underernæring i løbet af de næste to årtier,
 Tarmmikrobiom forbundet med adfærdsproblemer hos børn
Tarmmikrobiom forbundet med adfærdsproblemer hos børn
 Oral sex kan udløse bakteriel vaginose
En ny undersøgelse offentliggjort i tidsskriftet PLOS Biologi har afsløret forskellige mikrobielle populationer kan lette vaginal kolonisering af patogener, som igen kan forlænge egenskaberne ved ba
Oral sex kan udløse bakteriel vaginose
En ny undersøgelse offentliggjort i tidsskriftet PLOS Biologi har afsløret forskellige mikrobielle populationer kan lette vaginal kolonisering af patogener, som igen kan forlænge egenskaberne ved ba
 Undersøgelse tyder på en forbindelse mellem probiotisk brug og "hjernetåge"
En undersøgelse foretaget ved Medical College of Georgia ved Augusta University har vist, at probiotisk indtagelse kan resultere i en betydelig ophobning af tyndtarmbakterier, der fører til tåget i hj
Undersøgelse tyder på en forbindelse mellem probiotisk brug og "hjernetåge"
En undersøgelse foretaget ved Medical College of Georgia ved Augusta University har vist, at probiotisk indtagelse kan resultere i en betydelig ophobning af tyndtarmbakterier, der fører til tåget i hj
 Kronisk hoste kunne lindres med nyt lægemiddel
Et par undersøgelser har vist, at kronisk hoste og dens plagsomme symptomer kunne lindres ved hjælp af et nyt lægemiddel. Også, stoffet ikke har bivirkninger, der kan begrænse dets anvendelse, forklar
Kronisk hoste kunne lindres med nyt lægemiddel
Et par undersøgelser har vist, at kronisk hoste og dens plagsomme symptomer kunne lindres ved hjælp af et nyt lægemiddel. Også, stoffet ikke har bivirkninger, der kan begrænse dets anvendelse, forklar