Magenkrebs (GC) bleibt eine der Hauptursachen für Morbidität und Mortalität weltweit, und es ist daher ein klarer Bedarf für empfindlichere Früherkennungs Biomarkern zu suchen. Wir führten eine systematische Überprüfung der acht veröffentlichten Studien miRNA Profilierungs, die GC-Gewebe mit benachbarten noncancerous Gewebe verglichen. Ein miRNA-Ranking-System wurde verwendet, dass die Häufigkeit der Vergleiche nahm, die Richtung der differentiellen Expression und Gesamtstichprobengröße in Betracht. Wir identifizierten fünf miRNAs, die waren die meisten konsequent werden upregulated berichtet (miR-21, miR-106b, miR-17, miR-18a und miR-20a) und zwei miRNAs, die nach unten reguliert wurden (miR-378 und miR-638). Sechs davon wurden in 32 gepaarte Sätze von GC und benachbarten noncancerous Gewebeproben unter Verwendung von Echtzeit-PCR weiter validiert. MiR-21, miR-106b, miR-17, miR-18a und miR-20a wurden bestätigt upregulatedin GC Gewebe zu sein, während die Expression von miR-378 verringert wurde. Außerdem fanden wir eine signifikante Assoziation zwischen der Expression von miR-21, miR-106b, miR-17, miR-18a und miR-20a und klinisch-pathologischen Eigenschaften von GC. Diese miRNAs können für diagnostische und /oder prognostischer Biomarker für die GC und damit rechtfertigen weitere Untersuchungen verwendet werden
Citation:. Wang J-L, Hu Y, Kong X, Wang Z-H, Chen H-Y, Xu J, et al. (2013) Kandidaten microRNA Biomarkers in menschlichen Magenkrebs: Eine systematische Übersicht und Validierungsstudie. PLoS ONE 8 (9): e73683. doi: 10.1371 /journal.pone.0073683
Editor: William CS. Cho, Queen Elizabeth Hospital, Hong Kong
Empfangen: 16. Mai 2013; Akzeptiert: 19. Juli 2013 beginnen; Veröffentlicht: 9. September 2013
Copyright: © 2013 Wang et al. Dies ist eine Open-Access-Artikel unter den Bedingungen der Lizenz Creative Commons, die uneingeschränkte Nutzung erlaubt, die Verteilung und Vervielfältigung in jedem Medium, vorausgesetzt, der ursprüngliche Autor und Quelle genannt werden
Finanzierung:. Diese Arbeit durch Zuschüsse aus dem National Natural Science Foundation of Key-Programm (Nr 30830055), das Ministerium für öffentliche Gesundheit, China (Nr 200802094) unterstützt wurde, das Ministerium für Bildung (Nr 20090073110077) zu Fang JY, und der Doktor Innovation Foundation von Shanghai Jiao-Tong University School of Medicine (Nr BXJ201219) an Wang JL. Die Geldgeber hatten keine Rolle in Studiendesign, Datenerfassung und Analyse, Entscheidung oder Vorbereitung des Manuskripts zur Veröffentlichung
Konkurrierende Interessen:.. Die Autoren haben erklärt, dass keine Interessenkonflikte bestehen
Einführung
Trotz einer jüngsten Abnahme der Inzidenz von Magenkrebs (GC) [1], bleibt es eine Ursache der großen Morbidität und Mortalität weltweit, vor allem in Ostasien. Insgesamt eine Million neue Fälle von GC erfolgte im Jahr 2008 mit 738.000 Todesfälle [2]. Dies entspricht 8% der Gesamtzahl der Fälle von Krebs und 10% der gesamten Todesfälle. Obwohl Endoskopie die frühen Stadien von GC erkennen kann, sind den meisten Fällen noch in einem fortgeschrittenen Stadium diagnostiziert, die in einer schlechten Prognose führt [3]. Die 5-Jahres-Überlebensrate für die GC-Fälle mit Stadium II im Bereich von 30% bis 50%, fällt aber auf einen Wert zwischen 10% und 25% für Patienten mit Stadium III [4]. Obwohl endoskopische Techniken schnell entwickeln, deren Wert für die Früherkennung von GC ist begrenzt aufgrund eines Mangels an Empfindlichkeit, hohe Kosten und Unannehmlichkeiten. Neue diagnostische und prognostische Biomarker für GC sind daher dringend erforderlich.
MicroRNAs (miRNAs) sind kurze nichtkodierende RNA-Moleküle von 19-25 nt. Sie regulieren die Genexpression in der post-translationalen Ebene durch die RNA-induzierten Silencing-Komplex zu miRNA Zielstellen in der 3'-untranslatierten Region der mRNA führen, was zu einer mRNA-Abbau oder die Hemmung der Translation [5]. Frühere Studien haben gezeigt, dass zahlreiche miRNAs sind aberrantly in vielen Arten von Krebsarten, und miRNA-Expressionsprofilerstellung hat bestimmte miRNAs gezeigt mit Tumorentwicklung, Verlauf und das Ansprechen auf die Therapie in Verbindung gebracht werden. Sie sind daher gute Kandidaten für die Verwendung als diagnostische, prognostische und prädiktive Biomarker [6].
Mehrere Studien wurden durchgeführt, durch die Identifizierung der differentiellen Expression von miRNAs zwischen GC Gewebeproben und entsprechenden nicht-Tumor Magen nach Biomarkern zu suchen Gewebe aus dem gleichen Patienten [7] - [14]. Diese Untersuchungen haben zur Identifizierung von Hunderten von differentiell exprimierten miRNAs geführt. Allerdings sind viele von ihnen wahrscheinlich falsche Positive zu sein, und nur ein kleiner Anteil könnte als diagnostische oder prognostischen Biomarkern verwendet werden. Ein logischer Ansatz wichtig miRNAs aus einer großen Anzahl von Kandidaten-miRNA-Listen zu unterscheiden ist der Schnittpunkt der miRNAs in mehreren unabhängigen Studien [15] identifiziert zu suchen. Obwohl diese Methode hat [15] populär geworden zunimmt, [16], [17], nicht veröffentlichte Studie, die die Kreuzungen von GC-bezogenen miRNAs basierend auf einer Vielzahl von miRNA-Expressionsprofilerstellung Studien identifiziert hat.
Wir haben diese systematische Überprüfung der wichtigsten differentiell exprimierte miRNAs zu identifizieren, die in einer Reihe von unabhängigen miRNA-Expressionsprofilerstellung Studien in GC Patienten konsequent berichtet. Darüber hinaus haben wir weiter einige der miRNAs validiert, dass die meisten Up- waren oder nach unten reguliert mit Echtzeit-PCR in 32 Paare von GC und abgestimmt benachbarten Nicht-Tumorgewebeproben.
Ethikerklärung
die Studie wurde von der Ethikkommission der Shanghai Jiaotong University School of Medicine genehmigt wurde, und eine schriftliche Einverständniserklärung wurde von allen Patienten zu Beginn der Studie erhalten.
Suchstrategie
Potenzialstudien in englischer Sprache veröffentlicht wurden von Medline mit den folgenden Suchbegriffen gesammelt: 'miRNA' OR 'microRNA' OR 'miR', 'Magen' OR 'Magen', 'Profilierungs' OR 'Microarray ". Referenzlisten von Übersichtsartikeln und Originalartikel wurden manuell für weitere Publikationen gesucht.
Für eine Studie in dieser systematischen Überprüfung einbezogen werden, mehrere Kriterien, die erfüllt werden mussten : 1) hatte Studien miRNA-Studien in GC-Patienten zu sein; 2) Studien hatten GC Gewebe und deren entsprechenden benachbarten Nicht-Tumorgewebe zum Vergleich zu verwenden; 3) Verfahren hatte miRNA Microarray-Techniken umfassen. Des Weiteren wurden nur Volltext-Publikationen in englischer Sprache enthalten. Die Profilierung Studien, die GC-Zelllinien oder Serumproben von GC Patienten verwendet, die mit verschiedenen Krankheitsstadien verglichen jene GC Biopsien von Tumoren, und jene, die unterschiedliche miRNA-Technologien verwendet wurden nicht berücksichtigt. Übersichtsartikel wurden auch nicht in dieser systemischen Überprüfung einbezogen.
Differentiell exprimierte miRNAs identifiziert wurden aus jeder Profilierungs Studie eingeschlossen. Relevante Informationen bestimmt wurde (dh chromosomalen Lage, pre-miRNA Länge, reifen miRNA-Sequenz und mögliche Ziele der miRNAs), und fehlende Informationen wurde aus der miRBase Datenbank (www. Mirbase.org/) und Pubmed identifiziert.
Ranking
Jede inklusive Profilierung Studie [7] - [14], um eine Liste von differentiell exprimierten miRNAs (Tabelle S1) zur Verfügung gestellt. Griffith und Chan entwickelte eine Methode potenzielle molekulare Biomarker für die Vergleichsgruppen auf Rang [16], [17], die für die miRNA-Studien verwendet wurde. Beispielsweise Ma et al. [15] identifiziert die Schnittpunkte von kolorektalen miRNAs krebsbedingte basierend auf einer großen Anzahl von miRNA Profilierungsstudien. So wurden auf diejenigen, die in ihren Berichten basieren die Kriterien für die Literatur in diesem aktuellen systematischen Überprüfung einbezogen [15]. MiRNAs wurden in der folgenden Reihenfolge der Wichtigkeit der Kriterien gewählt: (i) die miRNA wurde konsequent als differentiell in einer konsistenten Richtung der Veränderung ausgedrückt berichtet; (Ii) die Frequenz des miRNA wurde in der Microarray-Studien berichtet; (Iii) die gesamte Stichprobengröße für jede konsistente miRNAs berichtet.
Die Profilierung Ergebnisse zu validieren, 32 frische GC Gewebe und deren gepaart Nicht-Tumor Magen Gewebe wurden aus dem Renji Krankenhaus, angeschlossen an die Shanghai Jiaotong University School of Medicine erhalten. Gesamt-RNA wurde aus 32 Paaren von angepassten menschlichen GC Proben (einschließlich Krebs und benachbarten Geweben noncancerous) unter Verwendung von TRIzol-Reagens (Invitrogen) extrahiert. Die RNA-Konzentration und die Reinheit wurde Nanodrop ND-2000 gemessen, und das Messverfahren ultraviolette Absorption wurde die Reinheit der RNA zu detektieren angewendet, werden nur die A260 /A280 liegt zwischen 1,80-2,00 und A260 /A230 > 1,7, wurden verwendet für werden muss, der letzte Versuch, sonst wird die RNA Wieder extrahiert. Reverse Transkription von 3 &mgr; g RNA wurde durchgeführt usingAll-in-OneTM Erststrang-cDNA Synthesis Kit (Genecopoeia, Guangzhou, China), entsprechend dem Protokoll des Herstellers. Kurz gesagt, wurde die hergestellte RNA reversen Transkriptionsreaktionslösung bei 37 ° C für 60 Minuten inkubiert und für 5 Minuten bei 85 ° C beendet wird, und dann bei -20 ° C für die weitere Analyse gespeichert. Quantitative PCR (qPCR) wurde unter Verwendung eines ABI Prism 7900HT Sequence Detection System (Applied Biosystems, USA) mit SYBR Premix Ex Taq II (Takara) durchgeführt. Die Primer (miR-21-5p, miR-106b-5P, miR-17-5p, miR-18a-5P, miR-20a-5P und miR-378-5p) mit U6 wurden aus Genecopoeia (Guangzhou, China) erhalten . Die Quantifizierung wurde mit der 2 -ΔΔCT Methode berechnet und als normalisierte Muster dargestellt.
Die Ergebnisse wurden mit Hilfe analysiert SAS 9.2-Software (SAS Institute Inc. USA). Die Daten werden als Mittelwert ± SD dargestellt. Student-t-Test wurde verwendet, um Werte zwischen zwei unabhängigen Gruppen zu vergleichen.
 Genetik kann die Zusammensetzung des Mikrobioms stärker beeinflussen als Umweltfaktoren
Genetik kann die Zusammensetzung des Mikrobioms stärker beeinflussen als Umweltfaktoren
 Colitis ulcerosa
Colitis ulcerosa
 Obere Endoskopie
Obere Endoskopie
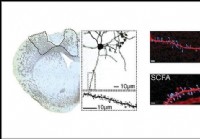 Die Nahrungsergänzung mit kurzkettigen Fettsäuren verbessert die Erholung von Schlaganfällen,
Die Nahrungsergänzung mit kurzkettigen Fettsäuren verbessert die Erholung von Schlaganfällen,
 Dass Pepto Ihrem Geschwür wahrscheinlich nicht hilft
Dass Pepto Ihrem Geschwür wahrscheinlich nicht hilft
 SARS-CoV-2-Infektion verlängert die Virusausscheidung und den Lymphozytenverlust bei Krebspatienten
SARS-CoV-2-Infektion verlängert die Virusausscheidung und den Lymphozytenverlust bei Krebspatienten
 Ganzkörpervibration hilft Entzündungen zu reduzieren,
dank des Darmmikrobioms Ganzkörpervibrationen scheinen viele Symptome von Diabetes mellitus Typ II zu verbessern. wobei Glukose und destruktive Entzündungen in die Höhe schießen. Das Verfahren hilft d
Ganzkörpervibration hilft Entzündungen zu reduzieren,
dank des Darmmikrobioms Ganzkörpervibrationen scheinen viele Symptome von Diabetes mellitus Typ II zu verbessern. wobei Glukose und destruktive Entzündungen in die Höhe schießen. Das Verfahren hilft d
 Bio-Äpfel haben probiotische Eigenschaften
„Ein Apfel am Tag…“, sagt ein altes Sprichwort. Es scheint, dass alle zustimmen, Gemessen an der Nummer 1 Position, die Äpfel unter den Früchten auf der ganzen Welt einnehmen, mit 83 Millionen Äpfeln,
Bio-Äpfel haben probiotische Eigenschaften
„Ein Apfel am Tag…“, sagt ein altes Sprichwort. Es scheint, dass alle zustimmen, Gemessen an der Nummer 1 Position, die Äpfel unter den Früchten auf der ganzen Welt einnehmen, mit 83 Millionen Äpfeln,
 Colitis ulcerosa und eine fehlende Mikrobe im Darm
Colitis ulcerosa ist eine stark schwächende entzündliche Erkrankung des Darms, die zu lähmenden Symptomen führt, die die Lebensqualität stark beeinträchtigen können. Forscher der Stanford University S
Colitis ulcerosa und eine fehlende Mikrobe im Darm
Colitis ulcerosa ist eine stark schwächende entzündliche Erkrankung des Darms, die zu lähmenden Symptomen führt, die die Lebensqualität stark beeinträchtigen können. Forscher der Stanford University S