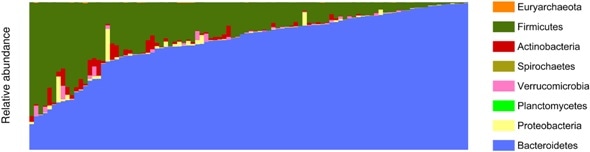
 Das gestapelte Balkendiagramm der phylogenetischen Zusammensetzung aller Mikrobiom-Taxa in dieser Studie kollabierte auf Phyla-Ebene in Kotproben. Grüne Balken stehen für Firmicutes und die blauen für Bacteroidetes, die beiden häufigsten Bakterienfamilien. Aus ästhetischen Gründen sind die Stichproben (n =98, unten) wurden nach ihrer Zusammensetzung von Bacteroidetes und Firmicutes sortiert, um zu zeigen, wie die Ausgangsergebnisse des Darmmikrobioms aus dieser Studie in Verbindung mit Ergebnissen aus früheren Studien verwendet werden können. Bildnachweis:King et al., 2019
Das gestapelte Balkendiagramm der phylogenetischen Zusammensetzung aller Mikrobiom-Taxa in dieser Studie kollabierte auf Phyla-Ebene in Kotproben. Grüne Balken stehen für Firmicutes und die blauen für Bacteroidetes, die beiden häufigsten Bakterienfamilien. Aus ästhetischen Gründen sind die Stichproben (n =98, unten) wurden nach ihrer Zusammensetzung von Bacteroidetes und Firmicutes sortiert, um zu zeigen, wie die Ausgangsergebnisse des Darmmikrobioms aus dieser Studie in Verbindung mit Ergebnissen aus früheren Studien verwendet werden können. Bildnachweis:King et al., 2019 Obwohl das Forschungsinteresse an der Bedeutung des menschlichen Darmmikrobioms für die allgemeine Gesundheit weiter zunimmt, Derzeit steht Forschern und Patienten keine umfassende Referenzliste für das Darmmikrobiom zur Verfügung. In dieser Studie, King und seine Kollegen beginnen mit der Katalogisierung des Organismus gesunder menschlicher Darmmikrobiome. und entwickelte eine Prototyp-Berichtsvorlage für Kliniker, um die Ergebnisse an die Patienten weiterzugeben.
Um ihre Datenbank zusammenzustellen, die Autoren haben 48 Stuhlproben von sechzehn gesunden Teilnehmern, die auf dem Campus der George Washington University in Washington rekrutiert wurden, genetisch sequenziert, DC, zusätzlich zur Verwendung von 50 fäkalen metagenomischen Proben, die aus dem Human Microbiome Project von als „gesund“ gescreenten Personen heruntergeladen wurden.
Nach dem Parsen aller genomischen und metagenomischen Sequenzen aller Proben mit einem neuartigen softwarebasierten Workflow King und Kollegen erstellten eine erste Datenbank mit bestätigten Mikroben und ihrer relativen Häufigkeit in allen Proben. die GutFeelingKB, Nutzung der umfassenden und öffentlich zugänglichen genetischen Informationen des NCBI, um Metadaten zu den beschriebenen Organismen bereitzustellen.
Die GutFeelingKB beschreibt 157 Organismen (155 Bakterien- und zwei Archaeen) aus 60 verschiedenen Gattungen. Der größte vertretene Bakterienstamm war Firmicutes (40 Prozent aller Organismen auf der Liste), die wiederum zu 20 Prozent aus Clostridien bestand, 19 Prozent Bakterioidien, 17 Prozent Bifidobakterien, 14 Prozent Enterobakterien, und 14 Prozent Lactobacillales-Bakterien – Bakterienklassen, die auch in Joghurt und anderen probiotischen Lebensmitteln vorkommen. Die Autoren stellen auch fest, dass allen Proben 84 Organismen gemeinsam waren, Dies deutet möglicherweise darauf hin, dass es sich um Kernarten für den menschlichen Darm handeln könnte.
Diese Studie rekrutierte nur sechzehn Teilnehmer, eine kleine Probe. Aber während weitere Studien möglicherweise weitere Organismen aus der ganzen Welt in gesunden Eingeweiden identifizieren könnten, GutFeelingKB ist ein wichtiger erster Schritt. Die Datenbank könnte als Ausgangspunkt für die vergleichende Analyse von Proben und die Entwicklung zukünftiger Behandlungen für das Mikrobiom von Patienten dienen.
Die Autoren fügen hinzu:
Unser Ziel ist es, das gesunde Darmmikrobiom abzubilden, damit wir und andere Forscher unsere Daten nutzen können, um krankheitsspezifische Vorhersagemodelle zu entwickeln."
 Typ-1-Diabetes im Zusammenhang mit Darmmikrobiom und genetischen Faktoren
Typ-1-Diabetes im Zusammenhang mit Darmmikrobiom und genetischen Faktoren
 Neugeborenes Mausmodell liefert Hinweise auf die Ursache einer verheerenden Darmerkrankung bei anämischen Frühchen
Neugeborenes Mausmodell liefert Hinweise auf die Ursache einer verheerenden Darmerkrankung bei anämischen Frühchen
 Bio-Äpfel haben probiotische Eigenschaften
Bio-Äpfel haben probiotische Eigenschaften
 Könnten antivirale Verbindungen aus Mikroalgen SARS-CoV-2 und andere Viren bekämpfen?
Könnten antivirale Verbindungen aus Mikroalgen SARS-CoV-2 und andere Viren bekämpfen?
 Obere Endoskopie
Obere Endoskopie
 CED weitaus häufiger als erwartet,
CED weitaus häufiger als erwartet,
 Morbus Crohn
Morbus Crohn verursacht eine Entzündung des Magen-Darm-Trakts. Es kann mit Colitis ulcerosa und Reizdarmsyndrom verwechselt werden. aber Morbus Crohn ist einzigartig. An der Ogden Clinic GI at McKay,
Morbus Crohn
Morbus Crohn verursacht eine Entzündung des Magen-Darm-Trakts. Es kann mit Colitis ulcerosa und Reizdarmsyndrom verwechselt werden. aber Morbus Crohn ist einzigartig. An der Ogden Clinic GI at McKay,
 Eine Art von Darmbakterien kann das Darmkrebsrisiko erhöhen
Neue Forschungsergebnisse, die auf der NWRI-Krebskonferenz 2019 vorgestellt wurden, haben gezeigt, dass Menschen mit einer bestimmten Art von Bakterien im Darm mit höherer Wahrscheinlichkeit an Darmkr
Eine Art von Darmbakterien kann das Darmkrebsrisiko erhöhen
Neue Forschungsergebnisse, die auf der NWRI-Krebskonferenz 2019 vorgestellt wurden, haben gezeigt, dass Menschen mit einer bestimmten Art von Bakterien im Darm mit höherer Wahrscheinlichkeit an Darmkr
 Ein Multi-Omics-Ansatz zur Entwicklung von Medikamenten gegen COVID-19
Eine neue Studie auf dem Preprint-Server veröffentlicht medRxiv* berichtet im Mai 2020 über einen Multi-Omics-Ansatz, der es erleichtern könnte, Medikamente zu entwickeln, die gegen COVID-19 wirksam
Ein Multi-Omics-Ansatz zur Entwicklung von Medikamenten gegen COVID-19
Eine neue Studie auf dem Preprint-Server veröffentlicht medRxiv* berichtet im Mai 2020 über einen Multi-Omics-Ansatz, der es erleichtern könnte, Medikamente zu entwickeln, die gegen COVID-19 wirksam