Identifizierung und Validierung von Genen im Magen tumorigenesis
Zusammenfassung
Hintergrund
Magenkrebs beteiligt ist eine der häufigsten in Süd-Indien gesehen Krebserkrankungen. Leider mehr als 90% werden von der Zeit, die sie im Land zu einem tertiären Zentrum berichten fortgeschritten. Es besteht ein dringender Bedarf, diese Krebsarten zu charakterisieren und zu versuchen, potentielle Biomarker und neue therapeutische Targets zu identifizieren.
Materialien und Methoden kaufen Wir 24 Magenkrebs verwendet, 20 Gepaart normal (PN) und 5 offensichtlich normal von Patienten erhalten Magengewebe mit nicht-Magen-Krebs (anscheinend normal - aN) für die Studie Microarray durch die Validierung der wesentlichen Gene gefolgt (n = 63) durch eine relative Quantifizierung Taqman Low Density Array Real Time PCR verwendet wird. Wir haben dann eine maßgeschneiderte Quantibody Protein-Array, die Expression von 15 Proteinen in Magengewebe (4 AN, 9 PN und 9 Magenkrebs) zu validieren. Das gleiche Array-Format wurde verwendet, um die Plasmaspiegel dieser Proteine, die in 58 Patienten mit Magenkrebs und 18 von Patienten mit normal /nicht-malignen Magenbedingungen zu studieren.
Ergebnisse
Siebzehn-Gene (ASPN, CCL15 /MIP-1δ , MMP3, SPON2, PRSS2, CCL3, TMEPAI /PMEPAI, SIX3, MFNG, SOSTDC1, SGNE1, SST, IGHA1, AKR1B10, FCGBP, ATP4B wurden NCAPH2) gezeigt unterschiedlich zum ersten zwischen den Tumoren und dem gekoppelten normal, ausgedrückt werden Zeit. EpCAM (p = 0,0001), IL8 (p = 0,0003), CCL4 /MIP-1β (p = 0,0026), CCL20 /MIP-3α (p = 0,039) und TIMP1 (p = 0,0017) Gewebe-Proteinspiegel waren signifikant unterschiedlich (Mann Whitney U-Test) zwischen Tumoren im Vergleich zu einem & PN. Darüber hinaus wurden mittlere Plasmaspiegel von IL8, CXCL9 /MIG, CCL3 /MIP-1α, CCL20 /MIP-3α, PDGFR-B und TIMP1 Proteine signifikant unterschiedlich zwischen der nicht-malignen Gruppe und der Magenkrebs-Gruppe. Die postoperativen Ebenen der EpCAM, IGFBP3, IL8, CXCL10 /IP10, CXCL9 /MIG, CCL3 /MIP-1α, CCL20 /MIP-3α, SPP1 /OPN und PDGFR-B zeigte eine gleichmäßige Tropfen in alle Proben untersucht.
Schlussfolgerungen
Unsere Studie mehrere Gene differentiell in Magenkrebs ausgedrückt, einige zum ersten Mal identifiziert hat. Einige von ihnen wurden auf Proteinebene bestätigt, wie gut. Einige dieser Proteine müssen weiter auf ihr Potenzial als diagnostische Biomarker in Magenkrebs evaluiert werden und einige als Follow-up-Marker bei Magenkrebs nützlich sein könnte.
Einführung
Magenkrebs ist eine der häufigsten Krebserkrankungen gesehen in Südindien, auf Platz 2
nd bei Männern und 5 th bei Frauen im Bereich Chennai Metropolitan [1]. Von den Patienten mit der Tertiär Institution präsentieren, werden mehr als 90% bei Präsentation vorgeschoben und nur palliative Management in diesen Patienten möglich ist [2]. In den Jahren 2005 und 2006 wurden am Institut insgesamt 1239 Magenkrebs-Patienten gesehen, von denen 211 Patienten, die zuvor an anderer Stelle behandelt worden war. Unter den nicht vorbehandelten Patienten (n = 1028) wurden 61% lokal fortgeschrittenem und 39% waren mit Fernmetastasen. Im Hinblick auf die fortgeschrittene Art der Erkrankung und aufgrund der schlechten Performance-Status, kam nur 91 Patienten für eine Operation mit der Absicht, bis zu heilen. Dies unterstreicht das Problem des Mangels an Früherkennung für den Magenkrebs in Indien.
In Japan, die eine hohe Inzidenz von Magenkrebs hat, ist Schirmbild Screening als Screening-Programm für die Bevölkerung durchgeführt, bei der Früherkennung von Läsionen führt, einige nur an Mukosa beschränkt [3]. Ein solches Verfahren ist unwahrscheinlich, dass die Lösung in einem großen Land wie Indien zu sein. Im Hinblick auf die subtilen Symptome wie Verdauungsstörungen, allmählicher Gewichtsverlust in der Regel ignoriert werden, die meisten Patienten mit fortgeschrittener Erkrankung vorhanden. Es ist daher wichtig, zuverlässige Screeningtests zu entwickeln, die bei der Früherkennung der Krankheit helfen kann. Serum basierte Tests für Pepsinogen und H. pylori-Antikörper haben keine weitverbreitete Akzeptanz gewonnen und nicht für das Screening von Personen, die von der National Cancer Center, Tokyo, Japan [3] betrachtet worden. Der diagnostische Test sollte vorzugsweise Blut- oder Urinprobe basiert und muss spezifisch sein.
Zu den wichtigsten Risikofaktoren für Magenkrebs, Ernährung spielt eine wichtige Rolle. Der Konsum von gesalzenen Speisen (Fisch und Fleisch) und Tabak sind mit einem erhöhten Risiko verbunden [4-6]. Chronischer atrophischer Gastritis und intestinale Metaplasie wurden als prämalignen Veränderungen der Magenschleimhaut betrachtet. Das Rauchen von Tabak, H. pylori, Diäten mit hohem Gehalt an Salz, Nitrite und Nitrate und geringe Aufnahme von Obst und Gemüse sind bekannte Risikofaktoren für chronische atrophische Gastritis [7]. Die südindische Diät besteht traditionell aus hohen kühl Inhalt und tiefe Lebensmittel gebraten, mit dem Öl vor zum Braten läuft mehrere Zyklen der Nutzung verwendet verworfen werden und somit einen hohen Gehalt an Karzinogene enthalten [5, 8, 9].
Magenkrebs Adenokarzinome sind überwiegend und könnte aus drei Untertypen sein - Intestinale, Diffuse und Mixed [10]. Die intestinale Art von Magenkrebs ist in der Regel im distalen Teil des Magens zu sehen ist, hat Präkanzerosen und umfasst kohäsiver Krebszellen gland artigen Strukturen [11] bildet. Die meisten der Darmtypen sind gut mäßig differenzierten (WHO Klassifikation). Die Diffuse Typ besteht aus einzelnen Krebszellen infiltrieren und weit über die makroskopischen Grenzen zu verbreiten. Sie sind meist schlecht differenzierten zu undifferenzierten [12]. In Chennai, sind distale Tumoren häufiger als proximalen Krebserkrankungen.
Genexpression Studien in verschiedenen Krebsarten haben dazu beigetragen, bei der Identifizierung in den Prozess der Tumorentstehung beteiligten Gene [13]. Microarray-Studien, die die Genexpression Unterschiede zwischen normalen Magen und Magenkrebs [14], zwischen jungen und älteren Patienten mit Magenkrebs-Tumoren [15], zwischen Primärtumor und Metastasen zu vergleichen [16] berichtet. Unsere Studie vergleicht die Genexpression zwischen scheinbar normalen Magen von Patienten erhalten Gewebe mit nicht-Magen-Krebs (anscheinend normal - AN), Magen-Gewebeproben weit weg vom Tumor und bestätigt durch Gefrierschnitt nicht Tumorzellen zu haben (Gepaart normal - PN) und Magenkrebs (Geschwülste - T). Dies ist die erste Studie, in die Genexpressionsmuster von Magenkrebs in südindischen Patienten zu suchen, und einige dieser Gene auf Proteinebene für ihr Potential als Biomarker für Magenkrebs.
Materialien und Methoden
Fünf AN Proben validieren (2 von Patienten mit Hypopharynx Krebs, 1 von der oberen Speiseröhre Plattenepithelkarzinome, 1 von peri-ampullären Karzinom, 1 von Bauchspeicheldrüsenkrebs) von Patienten, die Magen-Resektion als Teil ihrer ersten Operation unterzogen wurden in die Studie eingeschlossen. Zudem 24 Magenkarzinome und 20 PN wurden in die Studie eingeschlossen. Alle Patienten zur Verfügung gestellt, ihre informierte Zustimmung für die Studie, die von der Institutional Ethikkommission genehmigt wurde.
Die operativen Proben wurden sofort bearbeitet und die Schnitte wurden für Gefrierschnitt genommen. Tumorproben mit mehr als 70% Krebszellen; laufen Oesophagogastroduodenoskopie (OGDscopy) für Dyspepsie PN und AN Proben ohne Anzeichen von Tumorzellen und scheinbar normale Morphologie in die Studie aufgenommen wurden.
Zusätzlich wurden Blutproben von Individuen gesammelt und Magen-Darm-Pathologie jede obere auszuschließen. Davon wurden 58 gefunden Magenkrebs zu sein, wurden 6 gutartigen Pathologie haben im Magen (Gastritis, gutartige Geschwür) und 12 hatte eine normale OGDscopy Bericht gefunden. In 8 Magenkrebs-Patienten, die radikale Operation unterzog, postoperative Blutprobe auch zwischen Tag 7 und Tag 15 mit Ausnahme von zwei Patienten, bei denen die Probe entnommen wurde zum Zeitpunkt ihres ersten Follow-up nach der Operation gesammelt wurde (Tage 55 und 64) .
RNA-Extraktion
die RNA aus den Gewebeproben extrahiert wurde die RNeasy RNA-Extraktions-Kits (Qiagen GmbH, Hilden, Kat-Nr: 74106) gemäß den Anweisungen des Herstellers. Die Qualität der RNA für die Mikroarray-Analyse eingesetzt wurde mit der Bioanalyzer und Proben mit einer RNA Integrity Number (RIN) von 7 oder mehr geprüft wurden in die Studie eingeschlossen. RNA wurde unter Verwendung von Nanodrop ™ ND1000 (Nanodrop Technologies, USA) Spektralphotometer quantitativ bestimmt.
Nachweis von Helicobacter-pylori-
Analyse für H pylori im Magen-Gewebeproben wurde eine PCR durchgeführt unter Verwendung von wie zuvor beschrieben [17]. PCR Primer entworfen S2 Region Vac-A-Gen in der H pylori Genom zu amplifizieren, wurden für den Nachweis verwendet. Die Reaktion verstärkt, um ein Amplicon von etwa 194 bp Länge
Mikroarray-Experiment
1 ug Gesamt-RNA aus dem Tumor /PN /AN Probe und universal RNA (Stratagene, Cat no: 740.000 bis 41). Wurden revers transkribiert unter Verwendung von Array Skript bei 42 ° C für 2 Stunden zu erhalten cDNA, die die Amino Allyl- MessageAmp II aRNA Verstärkung Kit (Ambion, Austin, TX, Bestellnummer: AM1797). Die cDNA wurde amplifiziert, markiert, hybridisiert und Folien, wie zuvor beschrieben gescannt [18]
Alle Rohdatendateien eingereicht wurden mit einer zugewiesenen GEO-Zugangsnummer zu GEO -.. GSE17154
Microarray Datenanalyse
Die Vorder- und Hintergrund Median Intensität für Cy3 und Cy5 wurden importiert in BRB-ArrayTools Software [19], um den Import-Assistenten-Funktion. Hintergrundkorrektur wurde nicht durchgeführt. Globale Normalisierung wurde die Log-Verhältnisse auf jedem Array um Medianzentrum genutzt, um Unterschiede bei der Kennzeichnung Intensitäten der Cy3 und Cy5 Farbstoffen einzustellen. Die Daten wurden mit dem Klassen-Vergleichsmodul analysiert [20] in der BRB-ArrayTools Software.
Klasse Vergleich in BRB-Array-Tools kaufen Wir Gene identifiziert, die unterschiedlich zu den drei Klassen ausgedrückt wurden (Tumor /PN /AN) mit der Klasse Vergleichsmodul. Univariate F-Test wurde verwendet, und die Gene wurden als statistisch signifikant angesehen, wenn die p-Wert war < 0,001. Zusätzlich wird ein zweifacher Unterschied zwischen den verschiedenen Klassen erforderlich
Quantitative Echtzeit-PCR-
Echtzeit-Validierung der Genexpression erfolgte die TLDA real time PCR (Applied Biosystems, Foster City, CA; Kat. Nr: 4.342.261). Triplicate cDNA-Templat-Proben wurden verstärkt und analysiert auf der ABI Prism 7900HT Sequenzdetektionssystem (Applied Biosystems, Foster City, CA), wie zuvor beschrieben [18]., Die Rohdaten aus der Prism 7900HT Sequenzdetektionssystem importiert wurde in Microsoft Excel Software für die statistische Analyse der Daten. Unter den endogenen Referenzgenen auf dem Array (18S ribosomale-Gen; ACTB) umfasste, wurde ACTB gewählt, nachdem die globale Ct Wertverteilung zu visualisieren, um die Daten zu normalisieren. Zusätzlich HSPE1 die basierend auf der differentiellen Expression auf Microarray gesehen aufgenommen worden war, wurde festgestellt, minimale Abweichung aufweisen und somit wurde als zusätzliche endogene Kontrolle eingeschlossen. Die TLDA Assays wurden bei Labindia Instruments Pvt Ltd Labors in Gurgaon laufen, Neu-Delhi.
Die AN Proben als Kalibratoren verwendet wurden, und die relativen Quantifizierung Werte wurden für alle Gene und die Proben berechnet.
Gene Ontology Analysis
die Gene für zu differentiell in Tumoren exprimiert und gepaart Normalen "auf der Basis der Microarray-Daten importiert wurden in das Fatigo Modul von Babelomics [21] und analysiert über~~POS=TRUNC der GO Begriffe im Vergleich zum Rest des Genoms gefunden, mit Fisher-Test für 2 × 2 Kreuztabellen. Der p-Wert wurde bei <gesetzt; 0,05.
Herstellung von Seiden Proteinlysaten und Sammlung von Plasma
Proteinlysate für die Analyse unter Verwendung von Antikörper-Arrays 60 bis 80 mg des gefrorenen Magengewebeproben hergestellt. Die Gewebeproben wurden in Gegenwart von flüssigem Stickstoff gemahlen Mörser und Pistill. Das pulverisierte Gewebe wurde in Gewebe Lysepuffer (Tris. HCl pH 7,5, 150 mM Natriumchlorid, 1% Natriumdesoxycholat, 1% NP40) resuspendiert. Vor der Verwendung wurde Lysepuffer mit Complete Mini ™ Protease-Inhibitor-Cocktail (Roche Diagnostics GmbH, Deutschland) ergänzt. Die Protein-Lysate wurden einer Beschallung unterworfen Vibra Zelle ™ (Sonics Inc., USA) Beschallungsgerät. Die Lysate wurden durch Zentrifugation geklärt und quantifiziert unter Verwendung von Coomassie Plus Bradford-Test ™ Reagenz (Pierce Inc., USA) gemäß Herstellerprotokoll. Die Qualität der Proteine wurde durch die Lösung 50 ug des Lysats auf 10% Natriumdodecylsulfat-Polyacrylamid (SDS) Gel analysiert und anschließend sichtbar gemacht durch Coomassie-Blau-Färbung verwendet.
Plasma wurde aus 5 ml Blut in Gegenwart gesammelt erhalten von 200 ul 10% Ethylendiamin-tetraessigsäure (EDTA). Das Plasma aus den Blutproben isoliert wurde bei 3000 g und gelagert bei -80 ° C in Aliquots zentrifugiert
Antikörper Arrays
 Mundwasser beeinflusst die Wirkung von Bewegung
Mundwasser beeinflusst die Wirkung von Bewegung
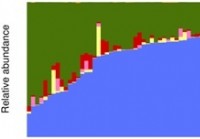 Die Studie beschreibt die erste Baseline-Datenbank des gesunden Darmmikrobioms und das Abundanzprofil
Die Studie beschreibt die erste Baseline-Datenbank des gesunden Darmmikrobioms und das Abundanzprofil
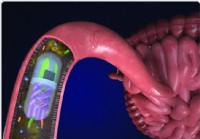 Wissenschaftler entwickeln eine 3D-gedruckte Pille, die Bakterien aus dem Darm untersucht
Wissenschaftler entwickeln eine 3D-gedruckte Pille, die Bakterien aus dem Darm untersucht
 Mundhygiene und Schweregrad von COVID-19 – die Verbindung
Mundhygiene und Schweregrad von COVID-19 – die Verbindung
 Fischschleim könnte eine potenzielle Quelle für Antibiotika sein
Fischschleim könnte eine potenzielle Quelle für Antibiotika sein
 Die Migration beeinflusst die Darmmikrobiota, was sich wiederum auf die Gesundheit auswirkt, finden Forscher
Die Migration beeinflusst die Darmmikrobiota, was sich wiederum auf die Gesundheit auswirkt, finden Forscher
 Ziegenmilchnahrung gut für die Darmgesundheit von Säuglingen
Forscher des RMIT-Labors analysierten Ziegenmilchnahrung und untersuchten die darin enthaltenen Oligosaccharide. Es wurde festgestellt, dass diese einfachen Zucker und die Präbiotika, die in der Säugl
Ziegenmilchnahrung gut für die Darmgesundheit von Säuglingen
Forscher des RMIT-Labors analysierten Ziegenmilchnahrung und untersuchten die darin enthaltenen Oligosaccharide. Es wurde festgestellt, dass diese einfachen Zucker und die Präbiotika, die in der Säugl
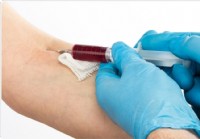 Bluttest auf mikrobielle DNA könnte vor Krebs warnen
Eine neue Studie in der Zeitschrift veröffentlicht Natur am 11. März 2020, könnte die derzeit vertretenen Ansichten über die Entstehung und Diagnose von Krebs verändern. Die Forscher haben eine neue
Bluttest auf mikrobielle DNA könnte vor Krebs warnen
Eine neue Studie in der Zeitschrift veröffentlicht Natur am 11. März 2020, könnte die derzeit vertretenen Ansichten über die Entstehung und Diagnose von Krebs verändern. Die Forscher haben eine neue
 Probiotika können biopolaren Patienten therapeutische Vorteile bieten
Eine Studie des Sheppard Pratt Health Systems in Baltimore hat gezeigt, dass Probiotika als potenzieller therapeutischer Ansatz für bipolare Störungen und andere psychiatrische Erkrankungen dienen kön
Probiotika können biopolaren Patienten therapeutische Vorteile bieten
Eine Studie des Sheppard Pratt Health Systems in Baltimore hat gezeigt, dass Probiotika als potenzieller therapeutischer Ansatz für bipolare Störungen und andere psychiatrische Erkrankungen dienen kön