 Estudio:La infección aguda por SARS-CoV-2 se asocia con una expansión de bacterias patógenas en la nariz, incluida Pseudomonas Aeruginosa. Haber de imagen:Christoph Burgstedt / Shutterstock
Estudio:La infección aguda por SARS-CoV-2 se asocia con una expansión de bacterias patógenas en la nariz, incluida Pseudomonas Aeruginosa. Haber de imagen:Christoph Burgstedt / Shutterstock El síndrome respiratorio agudo severo coronavirus 2 (SARS-CoV-2) se propaga principalmente por inhalación de partículas virales en el aire. Se cree que el sitio principal de la replicación viral inicial es la nariz, en lugar de la boca, debido a la mayor expresión del receptor de la enzima convertidora de angiotensina 2 (ACE2) en la nariz.
La replicación viral rápida en el tracto respiratorio superior puede provocar una infección en el tracto respiratorio inferior y una enfermedad grave. Sin embargo, muchos estudios no han demostrado ninguna correlación entre la gravedad de la enfermedad y la carga viral en la nariz, lo que sugiere que puede haber otros factores que contribuyan a la gravedad de la enfermedad.
Varios estudios han sugerido que el microbioma de la nariz y la garganta puede desempeñar un papel en las infecciones virales. La infección viral puede alterar la microbiota y provocar una coinfección. lo que puede empeorar la inflamación e interrumpir la respuesta inmunitaria. La infección grave también puede provocar una pérdida tanto del olfato como del gusto. Sin embargo, solo hay unos pocos estudios que han investigado el microbioma respiratorio en pacientes con COVID-19.
En un nuevo estudio publicado en el bioRxiv * servidor de preimpresión, Investigadores de la Universidad de California en Irvine investigaron el efecto de la infección por SARS-CoV-2 en el microbioma nasal.
El equipo de investigadores utilizó la secuenciación del amplicón del gen del ácido ribonucleico ribosómico (ARNr) 16S para determinar si la infección por SARS-CoV-2 provocaba un cambio en la composición del microbioma nasal. Este experimento encontró que el microbioma nasal de los pacientes con COVID-19 era, De hecho, diferente en comparación con aquellos que no tenían la enfermedad, así como la que estaba presente en las narices de los trabajadores de la salud. El microbioma nasal de los pacientes con COVID-19 y los trabajadores de la salud era más rico que el de los no pacientes; sin embargo, los microbiomas de los tres tipos de muestras estaban dominados por unos pocos microbios seleccionados.
El microbioma nasal de una persona sana se compone principalmente de Corynebacterium, Estafilococo, Estreptococo, Dolosigranulum, y Moraxella. Los autores encontraron que muchas bacterias patógenas, tal como Rothia , Acinetobacter , y Pseudomonas, fueron comunes en pacientes con COVID-19, con niveles particularmente altos de Pseudomonas aeruginosa informado en estos pacientes.
Los estudios han demostrado que las infecciones virales agudas pueden alterar el microbioma nasal y favorecer un aumento de bacterias patógenas. Un aumento en Pseudomonas también se ha reportado en casos de influenza, lo que sugiere que su presencia puede no ser específica de COVID-19, sino más bien una respuesta general a los procesos inflamatorios.
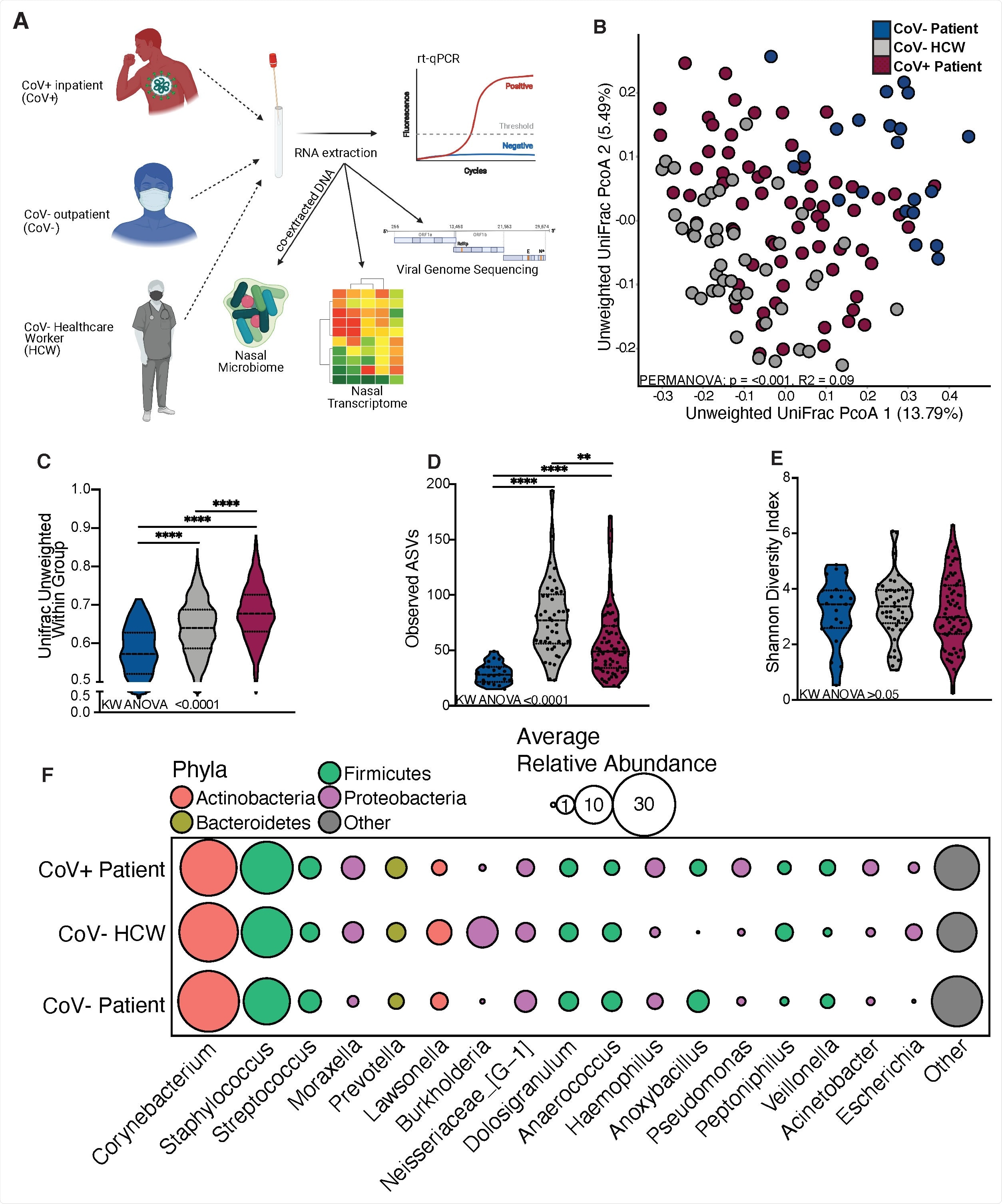 El microbioma nasal de los pacientes infectados con SARS-CoV-2 es distinto. (A) Esquema del diseño del estudio. (B) Análisis de coordenadas principales de la distancia UniFrac no ponderada de las comunidades microbianas nasales coloreadas según el estado del anfitrión. La contribución del estado del anfitrión a la varianza total en las matrices de disimilitud de UniFrac no ponderadas se midió utilizando PERMANOVA (Adonis con 10, 000 permutaciones). (C, D, E) Gráfico de violín que ilustra (D) distancias UniFrac no ponderadas promedio (C) número de variantes de secuenciación de amplicones observadas y (D) diversidad de Shannon (E) dividida por estado de anfitrión. La significancia para los paneles C-E se determinó utilizando ANOVA no paramétrico de Kruskal Wallis (valores de p insertados en la parte inferior de cada panel), con la comparación múltiple de Dunn * =p <0.05, ** =p <0,01, *** =p <0,001, **** =p <0,0001. (F) Gráficos de burbujas de géneros bacterianos que se encuentran en una abundancia promedio superior al 1% en toda la población de estudio, ordenados de izquierda a derecha en abundancia promedio descendente. El tamaño de cada círculo indica la abundancia relativa promedio para cada taxón en el grupo denotado y el color de cada círculo indica a qué Phyla bacteriano pertenece cada género.
El microbioma nasal de los pacientes infectados con SARS-CoV-2 es distinto. (A) Esquema del diseño del estudio. (B) Análisis de coordenadas principales de la distancia UniFrac no ponderada de las comunidades microbianas nasales coloreadas según el estado del anfitrión. La contribución del estado del anfitrión a la varianza total en las matrices de disimilitud de UniFrac no ponderadas se midió utilizando PERMANOVA (Adonis con 10, 000 permutaciones). (C, D, E) Gráfico de violín que ilustra (D) distancias UniFrac no ponderadas promedio (C) número de variantes de secuenciación de amplicones observadas y (D) diversidad de Shannon (E) dividida por estado de anfitrión. La significancia para los paneles C-E se determinó utilizando ANOVA no paramétrico de Kruskal Wallis (valores de p insertados en la parte inferior de cada panel), con la comparación múltiple de Dunn * =p <0.05, ** =p <0,01, *** =p <0,001, **** =p <0,0001. (F) Gráficos de burbujas de géneros bacterianos que se encuentran en una abundancia promedio superior al 1% en toda la población de estudio, ordenados de izquierda a derecha en abundancia promedio descendente. El tamaño de cada círculo indica la abundancia relativa promedio para cada taxón en el grupo denotado y el color de cada círculo indica a qué Phyla bacteriano pertenece cada género. Estos resultados corresponden a hallazgos de infecciones bacterianas secundarias en pacientes con COVID-19. Por lo tanto, la composición del microbioma nasal podría funcionar como el tipo de muestra principal para evaluar el riesgo de una persona de sufrir infecciones secundarias después de su recuperación de ciertas infecciones virales.
Adicionalmente, el grupo de investigación con sede en California también encontró que las cargas virales en la nariz no afectaron la diversidad del microbioma nasal. Se encontró que los pacientes con COVID-19 con cargas virales bajas tenían una cantidad más significativa de Estreptococo en su microbioma nasal , mientras que los pacientes con COVID-19 con cargas virales moderadas tenían una mayor cantidad de Corynebacterium . En tercer lugar, Se encontró que los pacientes con COVID-19 con altas cargas virales tenían mayores niveles de Cutibacterium, Neisseria, y Pseudomonas especies presentes dentro de sus microbiomas nasales.
Luego, los autores compararon los transcriptomas nasales de los sujetos de prueba y encontraron que 692 genes se expresaban de manera diferente entre los pacientes con COVID-19 y los trabajadores de la salud. Los genes regulados positivamente en individuos COVID-19 positivos se asociaron con vías de defensa del huésped. Los genes que están asociados con la muerte celular. así como los que codifican receptores inhibidores, también se regularon positivamente en pacientes con COVID-19.
Relativamente, Algunos de los genes en COVID-19 que se encontraron regulados a la baja incluyeron aquellos que están asociados con el mantenimiento de condiciones internas consistentes, organizacion celular, y procesamiento de tejido neuronal. Es más, los genes responsables de la producción de mucina en las fosas nasales, así como aquellos que influyen en los órganos sensoriales, También se encontró que estaban regulados a la baja. Tomados en conjunto, estos hallazgos podrían explicar la pérdida del olfato y el gusto que se ha informado ampliamente en pacientes con COVID-19.
Las infecciones intrahospitalarias siguen siendo un desafío importante en el ámbito clínico. Estas infecciones a menudo surgen debido a entornos hospitalarios y trabajadores sanitarios contaminados. Debido a su exposición prolongada en hospitales, los trabajadores sanitarios son portadores habituales de microbios patógenos.
Los autores encontraron niveles más altos de patógenos como Escerichia, Klebsiella, y Burkholderia en el microbioma nasal de los trabajadores sanitarios. La abundancia de Acinetobacter se encontró que era mayor tanto en pacientes con COVID-19 como en trabajadores de la salud, lo que puede deberse a una posible transferencia entre los dos.
Los resultados del presente estudio indican un cambio en el microbioma nasal de personas infectadas con SARS-CoV-2, con un aumento de patógenos como Pseudomonas aeruginosa . Una limitación de este estudio fue el hecho de que solo se evaluó el punto temporal; por lo tanto, estudios futuros que comparen la composición del microbioma nasal de pacientes con COVID-19, individuos sanos, y el hospital con el tiempo pueden mejorar la comprensión de cómo cambia el microbioma nasal.
bioRxiv publica informes científicos preliminares que no son revisados por pares y, por lo tanto, no debe considerarse concluyente, orientar la práctica clínica / comportamiento relacionado con la salud, o tratada como información establecida.
 Problemas de crecimiento en bebés prematuros asociados con bacterias intestinales alteradas
Problemas de crecimiento en bebés prematuros asociados con bacterias intestinales alteradas
 Grandes fagos recién descubiertos difuminan el límite entre vida y no vida
Grandes fagos recién descubiertos difuminan el límite entre vida y no vida
 Un gran estudio encuentra que la carga viral del SARS-CoV-2 es más baja en los niños
Un gran estudio encuentra que la carga viral del SARS-CoV-2 es más baja en los niños
 La infección por SARS-CoV-2 prolonga la diseminación viral y la pérdida de linfocitos en pacientes con cáncer
La infección por SARS-CoV-2 prolonga la diseminación viral y la pérdida de linfocitos en pacientes con cáncer
 Bacterias intestinales vinculadas a músculos más fuertes en personas mayores
Bacterias intestinales vinculadas a músculos más fuertes en personas mayores
 Complicaciones graves de COVID-19 relacionadas con la ruptura de la barrera intestinal
Complicaciones graves de COVID-19 relacionadas con la ruptura de la barrera intestinal
 La microbiota rural y urbana se diferencia de una edad temprana,
encuentra un nuevo estudio Un nuevo estudio ha demostrado que las diferencias en el microbioma intestinal humano entre las personas que viven en áreas rurales y urbanas de Nigeria comienzan a una eda
La microbiota rural y urbana se diferencia de una edad temprana,
encuentra un nuevo estudio Un nuevo estudio ha demostrado que las diferencias en el microbioma intestinal humano entre las personas que viven en áreas rurales y urbanas de Nigeria comienzan a una eda
 La dieta y la nutrición influyen en el microbioma de la mucosa del colon
La dieta es importante para mantener la salud humana, pero su mecanismo subyacente aún no se comprende completamente. Ahora, un equipo de investigadores arroja luz sobre la conexión entre la dieta y l
La dieta y la nutrición influyen en el microbioma de la mucosa del colon
La dieta es importante para mantener la salud humana, pero su mecanismo subyacente aún no se comprende completamente. Ahora, un equipo de investigadores arroja luz sobre la conexión entre la dieta y l
 La luz ultravioleta B es buena para el microbioma intestinal
Un pequeño estudio publicado en la revista Fronteras en inmunología el 24 de octubre 2019, muestra que la exposición a la luz ultravioleta B (UVB), que comúnmente se logra a través de la exposición
La luz ultravioleta B es buena para el microbioma intestinal
Un pequeño estudio publicado en la revista Fronteras en inmunología el 24 de octubre 2019, muestra que la exposición a la luz ultravioleta B (UVB), que comúnmente se logra a través de la exposición