Crédit d'image:Journal médical chinois
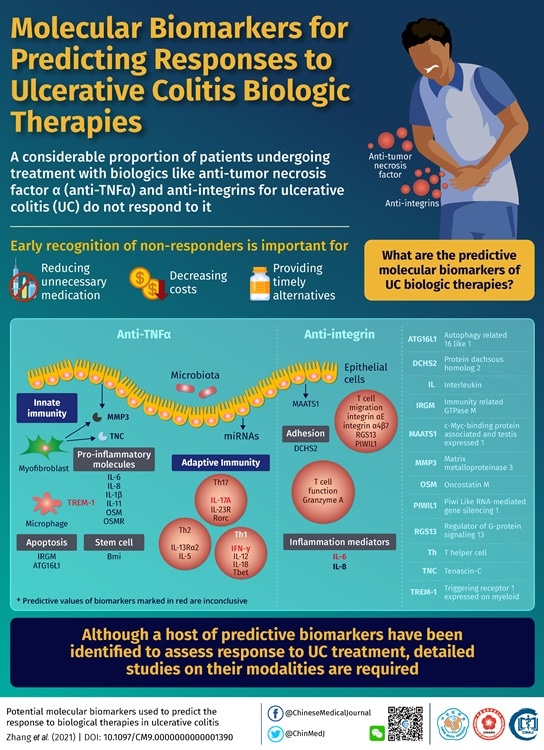
Des milliers de personnes dans le monde souffrent de maladies chroniques de l'intestin. Une de ces maladies courantes est la colite ulcéreuse (CU), qui provoque une inflammation et des plaies dans le tube digestif des personnes touchées. De façon intéressante, les symptômes de la CU sont causés par de nombreuses biomolécules de défense naturelles du corps devenues incontrôlables ! Quelques exemples incluent une élévation anormale des taux de facteur de nécrose tumorale (TNF)-a et d'intégrine. Pour traiter la CU, les médecins prescrivent plusieurs thérapies biologiques qui agissent pour faire baisser les niveaux élevés de ces biomolécules.
Bien que cela semble être une stratégie simple pour atténuer la CU, parfois, ces thérapies biologiques ne fonctionnent pas sur un sous-ensemble spécifique de patients, appelé à juste titre les non-répondants. Au grand désarroi des cliniciens et des non-répondeurs, ces situations conduisent à des médicaments inutiles, dépenses inutiles, et le retard dans le démarrage des alternatives. Des études placent ces non-répondeurs à un taux alarmant de 20 à 30 % pour le traitement anti-TNF, qui peut aller jusqu'à 15 à 30 % supplémentaires au fil du temps, et 43 % pour la thérapie anti-intégrine. De telles situations désastreuses appellent non seulement à de meilleures thérapies, mais aussi pour de meilleures mesures prédictives pour identifier ces non-répondants, tôt.
Plusieurs biomarqueurs moléculaires correspondant aux voies impliquées dans la réponse thérapeutique biologique spécifique contre la CU peuvent être considérés pour leur capacité à prédire la non-réponse. Un groupe de chercheurs de Chine et des États-Unis a envisagé cette possibilité et a cherché à identifier ces biomarqueurs ayant un potentiel prédictif de non-réponse thérapeutique. Leurs conclusions instructives ont été publiées sous la forme d'une revue complète dans Journal Médical Chinois .
Parlant de l'objectif de cet examen, Dr Ji Li du département de gastro-entérologie, Académie chinoise des sciences médicales et faculté de médecine de l'Union de Pékin, qui a dirigé l'étude, dit, " Nous résumons les prédicteurs moléculaires les plus potentiels de la réponse aux agents anti-TNF et anti-intégrine dans la CU, selon la physiopathologie ."
Par conséquent, Le Dr Li et son équipe ont identifié de larges catégories de biomarqueurs prédictifs, dont beaucoup permettent de contourner les effets thérapeutiques des produits biologiques utilisés. L'une de ces catégories importantes appartient au système immunitaire inné. Étant la première ligne de défense contre les entités envahissant le corps, ils sont susceptibles d'être actifs chez les non-répondeurs, pendant leur apparente « indifférence » à la thérapie biologique utilisée. Certains des biomarqueurs identifiés dans cette catégorie incluent le déclenchement du récepteur 1 exprimé sur myéloïde, autophagie liée 16 comme 1, gène GTPase M lié à l'immunité, ARNm, et les protéines.
De la même manière, l'autre catégorie importante appartient au système immunitaire adaptatif. Étant connu pour développer la "mémoire" des entités envahissantes, et aider à monter une défense appropriée, les biomarqueurs moléculaires prédictifs de cette catégorie sont susceptibles d'être efficaces pour identifier les non-répondeurs. De tels biomarqueurs ont déjà été rapportés dans plusieurs études, selon l'équipe du Dr Li, y compris les interleukines, Oncostatine M, etc.
Une autre catégorie importante est constituée par les biomolécules qui permettent la dégradation protéolytique des thérapies biologiques utilisées. Selon l'équipe, un biomarqueur moléculaire prédictif notable est la métalloprotéinase 3 de la matrice sérique, car il dégrade quelques agents anti-TNF classiquement utilisés in vitro , selon une étude récente qu'ils ont examinée.
Bien que l'utilisation de biomarqueurs moléculaires prédictifs uniques pour identifier les non-répondeurs semble idéale, les applications réelles justifient une approche plus pratique, qui combine plusieurs de ces biomarqueurs. L'équipe déclare que les modèles prédictifs ont été vantés par plusieurs études, comme voie à suivre. Ces modèles combinent plusieurs de ces biomarqueurs comprenant des gènes appropriés, protéines, polymorphismes mononucléotidiques, etc.
Bien que l'équipe d'examen propose différentes permutations et combinaisons de biomarqueurs moléculaires prédictifs pour identifier efficacement les non-répondeurs au traitement de la CU, aucun d'entre eux ne satisfait actuellement à l'applicabilité clinique en raison d'informations incomplètes. Bien que leur examen encourage la poursuite des recherches sur les biomarqueurs identifiés, l'équipe décrit également certaines des caractéristiques importantes à prendre en compte tout en visant un « combo efficace ».
Le biomarqueur prédictif idéal doit être facile à obtenir, montrer une grande précision, et avoir un retour rapide dans la pratique clinique ."
Dr Ji Li, Service de gastro-entérologie, Académie chinoise des sciences médicales et faculté de médecine de l'Union de Pékin
Globalement, cette revue est un tremplin pour résoudre le problème de « non-réponse » dans les thérapies biologiques utilisées pour traiter les patients atteints de CU, et peut certainement espérer inspirer des actions pour des problèmes similaires avec d'autres maladies.
 Comprendre les brûlures d'estomac
Comprendre les brûlures d'estomac
 La présence de certaines bactéries intestinales chez les mères pourrait protéger les bébés des allergies alimentaires
La présence de certaines bactéries intestinales chez les mères pourrait protéger les bébés des allergies alimentaires
 Bonne nouvelle pour les personnes souffrant du SII alors que les chercheurs identifient une « démangeaison intestinale »
Bonne nouvelle pour les personnes souffrant du SII alors que les chercheurs identifient une « démangeaison intestinale »
 Les personnes présentant des symptômes du SCI sont susceptibles d'avoir de faibles niveaux de vitamine D,
Les personnes présentant des symptômes du SCI sont susceptibles d'avoir de faibles niveaux de vitamine D,
 Des microbes sur la langue pourraient être utilisés pour diagnostiquer le cancer du pancréas
Des microbes sur la langue pourraient être utilisés pour diagnostiquer le cancer du pancréas
 Des scientifiques résolvent un mystérieux cas de syndrome de l'auto-brasserie
Des scientifiques résolvent un mystérieux cas de syndrome de l'auto-brasserie
 Ce Pepto n'aidera probablement pas votre ulcère
Un ulcère gastroduodénal est une plaie ou une lésion qui se développe dans la muqueuse de lœsophage, estomac, ou duodénum. Il ny a pas longtemps, on supposait que les ulcères étaient causés par une pe
Ce Pepto n'aidera probablement pas votre ulcère
Un ulcère gastroduodénal est une plaie ou une lésion qui se développe dans la muqueuse de lœsophage, estomac, ou duodénum. Il ny a pas longtemps, on supposait que les ulcères étaient causés par une pe
 La qualité du sommeil pourrait être un indicateur des découvertes ultérieures de la maladie d'Alzheimer
Une nouvelle étude des chercheurs de lUniversité de Californie, Berkeley a montré que la baisse progressive de la qualité du sommeil chez les personnes dans la cinquantaine et la soixantaine pourrait
La qualité du sommeil pourrait être un indicateur des découvertes ultérieures de la maladie d'Alzheimer
Une nouvelle étude des chercheurs de lUniversité de Californie, Berkeley a montré que la baisse progressive de la qualité du sommeil chez les personnes dans la cinquantaine et la soixantaine pourrait
 Le coronavirus se transmet-il par les selles ?
Une étude surprenante de Chine, publié dans la revue Gastroentérologie en mars 2020, rapporte que le nouveau coronavirus SARS-CoV-2 peut se propager par les matières fécales ainsi que par la voie re
Le coronavirus se transmet-il par les selles ?
Une étude surprenante de Chine, publié dans la revue Gastroentérologie en mars 2020, rapporte que le nouveau coronavirus SARS-CoV-2 peut se propager par les matières fécales ainsi que par la voie re