Le développement efficace de médicaments doit rationaliser le nombre de médicaments candidats qui entrent dans le cycle, pour réduire le coût et le temps du processus. Des études récentes recommandent d'intégrer une variété de techniques pour développer des pipelines pour la recherche et le développement (R&D) ainsi que d'utiliser des données génétiques pour identifier les nouveaux médicaments les plus susceptibles de réussir. La protéomique et la transcriptomique sont parmi les domaines les plus précieux à cette fin.
Maintenant, il existe plus de 150 essais cliniques testant des médicaments qui pourraient être efficaces pour augmenter la survie et améliorer la récupération des patients COVID-19. Il s'agit notamment de l'hydroxychloroquine, chloroquine, et baricitinib.
Une autre voie de collecte de preuves sur des médicaments potentiellement utiles contre COVID-19 consiste à trouver les protéines hôtes qui facilitent l'entrée et l'infection virales, et en examinant la possibilité de réaffecter des cibles médicamenteuses antérieures dans le SRAS-CoV pour lutter contre le virus actuel, coronavirus 2 du syndrome respiratoire aigu sévère (SARS-CoV-2).
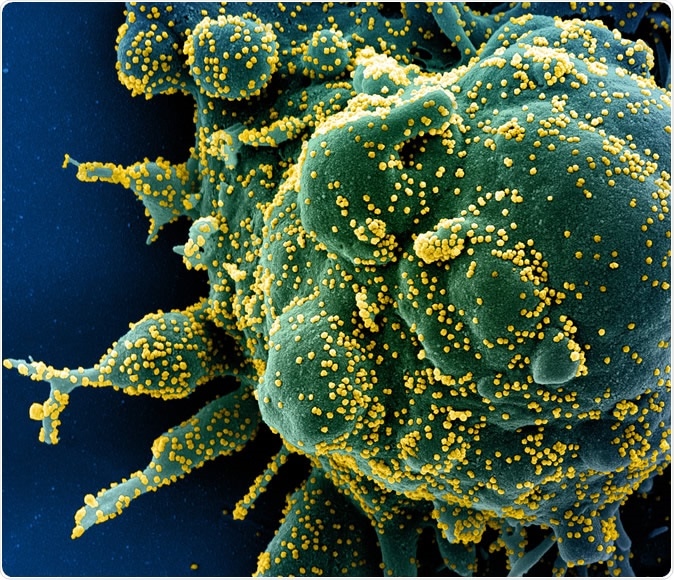 Nouveau coronavirus SARS-CoV-2 Micrographie électronique à balayage colorisée d'une cellule apoptotique (verte) fortement infectée par des particules du virus SARS-COV-2 (jaune), isolé à partir d'un échantillon de patient. Image capturée au NIAID Integrated Research Facility (IRF) à Fort Detrick, Maryland. Crédit :NIAID
Nouveau coronavirus SARS-CoV-2 Micrographie électronique à balayage colorisée d'une cellule apoptotique (verte) fortement infectée par des particules du virus SARS-COV-2 (jaune), isolé à partir d'un échantillon de patient. Image capturée au NIAID Integrated Research Facility (IRF) à Fort Detrick, Maryland. Crédit :NIAID Une étude récente a trouvé plus de 330 protéines hôtes humaines qui sont nécessaires pour que le virus infecte les humains. Ceux-ci interagissent avec 26 protéines virales. Cela pourrait aider à faire progresser la R&D le long de la première voie.
La deuxième méthode a été utilisée par quelques études, qui ont produit 59 gènes de souris liés à l'infection antérieure par le SRAS-CoV. Parmi ceux-ci, il y en a 44 qui ont des équivalents dans le génome humain. En bloquant les interactions virus-protéines humaines, il peut être possible de cibler plus efficacement les mécanismes d'infection virale avec un risque moindre de résistance aux médicaments, par rapport au ciblage direct du virus.
Un problème majeur avec cette approche est le danger de produire par inadvertance d'autres effets qui pourraient aggraver des maladies complexes - ou même leur être bénéfiques. La présente étude vise à évaluer comment ces cibles médicamenteuses pourraient affecter le fonctionnement du corps humain, basé sur une compréhension de la génétique sous-jacente.
Les chercheurs ont utilisé le protocole de priorisation des médicaments, qu'ils ont développé avec succès plus tôt, pour tester 353 cibles médicamenteuses susceptibles d'interagir avec le virus. Ils voulaient observer comment ces médicaments provoquaient d'autres effets visibles de l'infection, ainsi que la manière dont ils ont obtenu des effets à la fois intentionnels et imprévus sur des maladies complexes.
Ils ont d'abord construit un atlas des maladies présentant les protéines et les gènes humains qui participent à l'entrée virale. Cela s'est fait via des études de randomisation mendéliennes, fournissant plus de 372, 000 prédictions uniques de la façon dont le médicament affecte une maladie. Ceci était basé sur la protéomique plasmatique ainsi que sur la transcriptomique tissu-spécifique.
Par conséquent, ils ont pu évaluer comment ces 353 médicaments potentiels pourraient agir dans 49 phénotypes d'infection virale, comment ils pourraient affecter plus de 500 maladies complexes, et modifier 72 phénotypes de la maladie. Ces résultats ont été évalués par rapport aux données d'essais de médicaments, ainsi que le génome médicamenteux, identifier les principaux médicaments avec la plus grande possibilité de réutilisation, et le moins d'effets secondaires.
Ils ont créé une plateforme en ligne en libre accès qui contient les résultats de tous les tests, afin de permettre à quiconque d'examiner rapidement les résultats de l'un des médicaments.
L'atlas des interactions médicament-cible-maladie fournit plus de 370, 000 associations cible-maladie dans 11 tissus qui sont pertinentes dans le scénario COVID-19. D'eux, 833 présentaient des preuves solides de l'imagerie par résonance magnétique des 11 tissus. 726 d'entre eux ont également montré une colocalisation robuste, pour une probabilité de colocalisation supérieure à 70 %. Ce sont les résultats les plus solides de l'étude.
L'importance de détecter de telles associations est la capacité d'effectuer des analyses de la façon dont l'expression de certaines cibles affecte des maladies spécifiques, selon le tissu. Par exemple, les effets des cibles médicamenteuses sur la maladie de Crohn, hypertension, troubles atopiques, et le diabète pourraient être évalués. De 11 à 17 des gènes cibles avaient des associations avec ces quatre maladies, en fonction du tissu étudié.
Deuxièmement, les cibles médicamenteuses ont été analysées pour l'association avec 49 phénotypes d'infection virale. Il y avait deux associations fortes, à savoir, le gène NEU1 avec l'hépatite chronique et le gène DPY19L1 avec l'entérite virale. Il y avait aussi trois associations moins fortes mais suggestives, comme le gène JAK2 avec l'hépatite chronique.
Troisièmement, l'étude a montré 45 façons potentielles dont les protéines pourraient affecter les traits de la maladie et 430 associations similaires avec l'expression de l'ARNm. Parmi ceux-ci, il y avait 95 cibles associées à 105 maladies ou expressions de maladies. Le gène JAK2, par exemple, est associé à neuf phénotypes de maladie, allant de l'atopie à l'obésité, ce qui suggère des actions potentiellement pléiotropes pour ce gène.
Finalement, ils ont trouvé 249 associations où la cible du médicament affectait un phénotype de maladie différemment selon le tissu analysé. Parmi ceux-ci se trouvaient 52 gènes uniques exprimés dans 7 tissus pour affecter 47 maladies uniques.
Il y avait 29 effets tissu-dépendants fortement associés de la cible du médicament sur la maladie dans plus d'un seul tissu, parmi lesquels seulement deux n'ont pas montré la même direction d'effet lorsqu'ils sont exprimés dans le sang ainsi que dans une gamme d'autres tissus.
Par exemple, le gène DHODH affecte le cholestérol des lipoprotéines de basse densité (LDL). Un médicament commercialisé contre un LDL élevé, appelé Léflunomide, fonctionne en inhibant ce gène. Le léflunomide est également considéré comme une thérapie COVID-19.
L'analyse a montré que ce médicament s'exprime uniquement par rapport aux LDL sans beaucoup d'autres actions diverses, sur un large spectre de tissus, du poumon au côlon.
Les chercheurs ont noté les 726 associations trouvées par MR entre la cible et la maladie, en utilisant les données d'essais cliniques ainsi que les preuves putatives qu'ils ont causé différents effets phénotypiques dans une gamme de tissus, pour trouver les associations les plus droguées. 499 d'entre eux étaient des paires uniques de cible et de maladie, les autres étant observés dans plus d'un tissu.
Ils ont mis en place quatre classes de notation, comme le score omics, la note d'essai, le score de drogue, et le score d'infection. Chaque score était échelonné de 1 à 100. Il y avait des scores élevés pour 2 des 499 associations dans 3 des 4 classes, ce qui en a fait les cibles avec la plus haute priorité de développement. 77 ont obtenu un score élevé dans deux classes et 97 dans une. Les 323 autres avaient des scores universellement bas et sont donc considérés comme les cibles médicamenteuses les moins prioritaires.
Certaines cibles incluent le gène ITGB5 qui est ciblé par le médicament Cilengitide, qui était destiné aux glioblastomes et tumeurs similaires. Ses scores élevés et le profil génétique suggèrent qu'il a le potentiel de réduire l'hypertension artérielle. Parmi les 5 premiers, tous manquaient d'associations fortes avec des conditions telles que les arythmies cardiaques, ce qui pourrait affecter leur utilisation dans COVID-19.
D'autre part, le gène TLR9, qui est ciblé par le médicament hydroxychloroquine a des scores élevés dans deux catégories mais un score faible pour les deux autres. Aucune preuve clinique n'a encore été accumulée quant au bénéfice antiviral ou clinique d'une combinaison de ce médicament avec l'azithromycine. De plus, ce gène peut augmenter le risque d'accident vasculaire cérébral embolique, asthme, et certaines conditions immunitaires.
L'étude a pu promouvoir la hiérarchisation des cibles médicamenteuses de trois manières :en examinant les problèmes de sécurité pouvant découler de la réutilisation des médicaments pour le traitement COVID-19 ; l'identification de cibles médicamenteuses prometteuses ; et découvrir comment la cible du médicament affecte le phénomène humain dans différents tissus.
En utilisant cette approche, un médicament potentiel, baricitinib, est testé pour son efficacité dans COVID-19. On pense qu'il inhibe la protéine JAK2, et ainsi réduire l'inflammation systémique. Cependant, l'étude actuelle a proposé qu'il pourrait également provoquer une hépatite chronique. En réalité, la littérature suggère que l'hépatite B est réactivée après un traitement avec un inhibiteur de JAK 2 ruxolitinib.
Les trois médicaments à haut potentiel de cette étude comprennent le médicament immunosuppresseur Leflunomide, utilisé dans la polyarthrite rhumatoïde, entre autres conditions, et Cilengitide. Le premier a une activité antivirale contre plusieurs virus, et l'étude actuelle suggère également qu'il a des propriétés hypolipémiantes, tandis que ce dernier a une activité antihypertensive, peut-être.
Finalement, certaines de ces cibles sont exprimées dans plusieurs tissus pour produire le même phénotype, mais d'autres montrent un changement d'expression spécifique au tissu.
Bien que certaines limites existent, l'étude fournit un pipeline pour l'étude de génétique statistique et une plate-forme à accès ouvert pour organiser les cibles de médicaments contre COVID-19 par ordre de priorité. Les études d'association pangénomique (GWAS) pourraient encore améliorer la qualité des données, ce qui le rend encore plus utile pour trouver des cibles médicamenteuses prometteuses parmi les médicaments potentiellement réutilisés.
medRxiv publie des rapports scientifiques préliminaires qui ne sont pas évalués par des pairs et, donc, ne doit pas être considéré comme concluant, guider la pratique clinique/le comportement lié à la santé, ou traités comme des informations établies.
 L'infliximab peut entraver l'efficacité de certains vaccins COVID-19
L'infliximab peut entraver l'efficacité de certains vaccins COVID-19
 Des microbes sur la langue pourraient être utilisés pour diagnostiquer le cancer du pancréas
Des microbes sur la langue pourraient être utilisés pour diagnostiquer le cancer du pancréas
 Les personnes présentant des symptômes du SCI sont susceptibles d'avoir de faibles niveaux de vitamine D,
Les personnes présentant des symptômes du SCI sont susceptibles d'avoir de faibles niveaux de vitamine D,
 La césarienne est-elle bonne pour la santé de l'enfant ?
La césarienne est-elle bonne pour la santé de l'enfant ?
 Les vibrations du corps entier aident à réduire l'inflammation,
Les vibrations du corps entier aident à réduire l'inflammation,
 La greffe de liquide vaginal pourrait aider à traiter la vaginose bactérienne récurrente
La greffe de liquide vaginal pourrait aider à traiter la vaginose bactérienne récurrente
 Nouvel outil pour déchiffrer le microbiome intestinal
Les millions de bactéries résidant dans lintestin jouent un rôle très important dans la santé et la maladie. Cependant, un problème constant a été le manque de compréhension de la composition réelle d
Nouvel outil pour déchiffrer le microbiome intestinal
Les millions de bactéries résidant dans lintestin jouent un rôle très important dans la santé et la maladie. Cependant, un problème constant a été le manque de compréhension de la composition réelle d
 Les bactéries intestinales peuvent prédire le risque d'hypertension pulmonaire
Une équipe de chercheurs a découvert que la microflore des bactéries dans lintestin des humains pourrait contribuer au développement de lhypertension artérielle pulmonaire (HTAP). Létude intitulée, Pr
Les bactéries intestinales peuvent prédire le risque d'hypertension pulmonaire
Une équipe de chercheurs a découvert que la microflore des bactéries dans lintestin des humains pourrait contribuer au développement de lhypertension artérielle pulmonaire (HTAP). Létude intitulée, Pr
 La découverte de 100 nouveaux gènes pourrait aider la recherche sur les maladies de la pigmentation
Une nouvelle étude menée par des chercheurs du Kings College de Londres et du centre médical universitaire Erasmus MC de Rotterdam a découvert 124 gènes qui jouent un rôle clé dans la détermination de
La découverte de 100 nouveaux gènes pourrait aider la recherche sur les maladies de la pigmentation
Une nouvelle étude menée par des chercheurs du Kings College de Londres et du centre médical universitaire Erasmus MC de Rotterdam a découvert 124 gènes qui jouent un rôle clé dans la détermination de