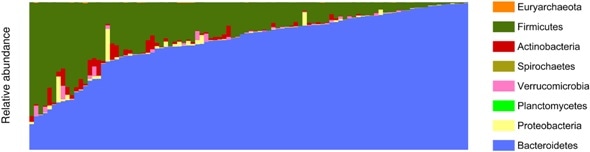
 Le graphique à barres empilées de la composition phylogénétique de tous les taxons du microbiome dans cette étude s'est effondré au niveau des phyla dans les échantillons fécaux. Les barres vertes représentent les Firmicutes et les bleues représentent les Bacteroidetes, les deux familles bactériennes les plus abondantes. A des fins esthétiques, les échantillons (n =98, bas) ont été triés en fonction de leur composition en Bacteroidetes et Firmicutes pour démontrer comment les résultats de base du microbiome intestinal de cette étude pourraient être utilisés en conjonction avec les résultats d'études antérieures. Crédit :King et al, 2019
Le graphique à barres empilées de la composition phylogénétique de tous les taxons du microbiome dans cette étude s'est effondré au niveau des phyla dans les échantillons fécaux. Les barres vertes représentent les Firmicutes et les bleues représentent les Bacteroidetes, les deux familles bactériennes les plus abondantes. A des fins esthétiques, les échantillons (n =98, bas) ont été triés en fonction de leur composition en Bacteroidetes et Firmicutes pour démontrer comment les résultats de base du microbiome intestinal de cette étude pourraient être utilisés en conjonction avec les résultats d'études antérieures. Crédit :King et al, 2019 Bien que l'intérêt de la recherche pour l'importance du microbiome intestinal humain pour la santé globale continue de croître, il n'y a actuellement aucune liste de référence complète du microbiome intestinal disponible pour les chercheurs et les patients. Dans cette étude, King et ses collègues commencent à cataloguer la composition organique des microbiomes intestinaux humains sains, et développé un prototype de modèle de rapport permettant aux cliniciens de transmettre les résultats aux patients.
Pour compiler leur base de données, les auteurs ont séquencé génétiquement 48 échantillons fécaux de seize participants en bonne santé recrutés sur le campus de l'Université George Washington à Washington, D.C., en plus d'utiliser 50 échantillons métagénomiques fécaux téléchargés à partir du Human Microbiome Project provenant d'individus sélectionnés comme « en bonne santé ».
Après avoir analysé les séquences génomiques et métagénomiques de tous les échantillons à l'aide d'un nouveau flux de travail logiciel, King et ses collègues ont compilé une base de données initiale de microbes confirmés et de leur abondance relative dans tous les échantillons, le GutFeelingKB, en utilisant les informations génétiques complètes et accessibles au public du NCBI pour fournir des métadonnées sur les organismes décrits.
Le GutFeelingKB décrit 157 organismes (155 organismes bactériens et deux organismes archéens) dans 60 genres distincts. Le plus grand phylum de bactéries représenté était Firmicutes (40 pour cent de tous les organismes de la liste), qui à son tour était composé de 20 pour cent de Clostridia, 19 pour cent de bactérioïdie, 17 pour cent de Bifidobacteriales, 14 pour cent d'entérobactéries, et 14% de bactéries Lactobacillales - des classes de bactéries également présentes dans le yaourt et d'autres aliments probiotiques. Les auteurs notent également que 84 organismes étaient communs à tous les échantillons, indiquant potentiellement qu'il pourrait s'agir d'espèces essentielles pour l'intestin humain.
Cette étude n'a recruté que seize participants, un petit échantillon. Mais alors que d'autres études pourraient continuer à identifier d'autres organismes présents dans des intestins sains du monde entier, GutFeelingKB est une première étape importante. La base de données pourrait servir de point de départ pour l'analyse comparative des échantillons et le développement de futurs traitements du microbiome des patients.
Les auteurs ajoutent :
Notre objectif est de cartographier le microbiome intestinal sain afin que nous et d'autres chercheurs puissions utiliser nos données pour développer des modèles de prédiction spécifiques à une maladie. »
 Les globules blancs et leur rôle dans le cerveau
Les globules blancs et leur rôle dans le cerveau
 Perte de la barrière épithéliale intestinale responsable du MIS-C lié au COVID-19 chez les enfants,
Perte de la barrière épithéliale intestinale responsable du MIS-C lié au COVID-19 chez les enfants,
 Le bain de bouche a un impact sur les effets de l'exercice
Le bain de bouche a un impact sur les effets de l'exercice
 Le régime méditerranéen favorise un vieillissement en bonne santé avec un microbiome intestinal plus sain
Le régime méditerranéen favorise un vieillissement en bonne santé avec un microbiome intestinal plus sain
 Ne craignez pas la coloscopie
Ne craignez pas la coloscopie
 Microbiome intestinal et MII - le lien peut-être dans l'alimentation selon une étude
Microbiome intestinal et MII - le lien peut-être dans l'alimentation selon une étude
 E. coli superbactérie se propageant par une mauvaise hygiène des toilettes,
pas par la nourriture Une nouvelle étude publiée dans Les maladies infectieuses du Lancet le 22 octobre 2019, dit quun superbactérie commun qui cause plus de 5, 000 cas dintoxication alimentaire en
E. coli superbactérie se propageant par une mauvaise hygiène des toilettes,
pas par la nourriture Une nouvelle étude publiée dans Les maladies infectieuses du Lancet le 22 octobre 2019, dit quun superbactérie commun qui cause plus de 5, 000 cas dintoxication alimentaire en
 Ne craignez pas la coloscopie
Les canaux radiculaires et les coloscopies ont quelque chose en commun :ils sont universellement craints par le public. Mais dans le domaine des procédures dentaires et des examens de routine nécessai
Ne craignez pas la coloscopie
Les canaux radiculaires et les coloscopies ont quelque chose en commun :ils sont universellement craints par le public. Mais dans le domaine des procédures dentaires et des examens de routine nécessai
 L'hypertension conduit-elle toujours à un COVID-19 sévère ?
Causée par lagent infectieux, syndrome respiratoire aigu sévère coronavirus 2 (SARS-CoV-2), la maladie à coronavirus 2019 (COVID-19) est souvent plus grave chez les personnes souffrant de problèmes mé
L'hypertension conduit-elle toujours à un COVID-19 sévère ?
Causée par lagent infectieux, syndrome respiratoire aigu sévère coronavirus 2 (SARS-CoV-2), la maladie à coronavirus 2019 (COVID-19) est souvent plus grave chez les personnes souffrant de problèmes mé