Abstract
I den æra av målrettet terapi, mutasjon profilering av kreft er en viktig del av å gjøre terapeutiske avgjørelser . For å karakterisere kreft på et molekylært nivå, er viktig anvendelse av formalinfiksert parafin-innleiret vev. Vi testet Ion AmpliSeq Cancer Hotspot Panel v2 og NCounter Kopier nummer Variasjon analysen i 89 formalinfiksert parafin-embedded magekreft prøver for å finne ut om de passer i arkiv kliniske prøver for personlig målrettet terapi. Vi validert resultatene med Sanger-sekvensering, sanntids kvantitativ PCR, fluorescens in situ hybridisering og immunhistokjemi. Ofte oppdages somatiske mutasjoner inkludert TP53 plakater (28,17%), APC plakater (10,1%), PIK3CA plakater (5,6%), KRAS plakater (4,5 %), SMO plakater (3,4%), STK11 plakater (3,4%), CDKN2A plakater (3,4%) og SMAD4 plakater (3,4%) . Utdypninger av HER2 Citation. Kim S, Lee J, Hong meg, IG, Kang SY, Ha SY, et al. (2014) høy gjennomstrømming sekvensering og Kopier nummer Variasjon Detection Bruke Formalin Fast Embedded Tissue i magekreft med spredning. PLoS ONE 9 (11): e111693. doi: 10,1371 /journal.pone.0111693 Redaktør: Hiromu Suzuki, Sapporo Medical University, Japan mottatt: 28 april 2014; Godkjent: 29 september 2014; Publisert: 05.11.2014 Copyright: © 2014 Kim et al. Dette er en åpen-tilgang artikkelen distribueres under betingelsene i Creative Commons Attribution License, som tillater ubegrenset bruk, distribusjon og reproduksjon i ethvert medium, forutsatt den opprinnelige forfatteren og kilden krediteres Data Tilgjengelighet:. Den forfatterne bekrefter at alle data som underbygger funnene er fullt tilgjengelig uten restriksjoner. Alle relevante data er i avisen og dens saksdokumenter filer Finansiering:. Denne studien ble støttet av en bevilgning fra Korea Healthcare Technology R & D Project, departementet for Health & Velferdsdepartementet, Republikken Korea (A101130) og en Samsung Biomedical Research Institute stipend (# SBRI-SP1B20112). Denne studien ble også støttet av en Samsung Medical Center utført stipend, 20 x 20 Project (# GFO1140111). Medforfattere Seokhwi Kim, Jeeyun Lee, Min Eui Hong, In-Gu Do, So Young Kang Sang Yun Ha, Seung Tae Kim, Se Hoon Park, Won Ki Kang, Min-Gew Choi jr Ho Lee, Tae Sung Sohn , Jae Moon Bae, Sung Kim og Kyoung-Mee Kim er ansatt i Samsung Medical Center. Medforfatter Duk-Hwan Kim er ansatt hos Samsung Biomedical Research Institute. Samsung Biomedical Research Institute og Samsung Medical Center gitt støtte i form av lønn for forfattere Seokhwi Kim, JL, meh, IGD, Syk, SYH, STK, SHP, WKK, MGC, JHL, TSS, JMB, Sung Kim, KMK og DHK , men ikke har noen ekstra rolle i studiedesign, datainnsamling og analyse, beslutning om å publisere, eller utarbeidelse av manuskriptet. De spesifikke roller disse forfatterne er formulert i § "forfatter bidrag ' Konkurrerende interesser:. Forfatterne har følgende interesser: Denne studien ble finansiert delvis av Samsung Biomedical Research Institute og Samsung Medical Center. Medforfattere Seokhwi Kim, Jeeyun Lee, Min Eui Hong, In-Gu Do, So Young Kang Sang Yun Ha, Seung Tae Kim, Se Hoon Park, Won Ki Kang, Min-Gew Choi jr Ho Lee, Tae Sung Sohn , Jae Moon Bae, Sung Kim og Kyoung-Mee Kim er ansatt i Samsung Medical Center. Medforfatter Duk-Hwan Kim er ansatt hos Samsung Biomedical Research Institute. Det finnes ingen patenter, produkter under utvikling eller markedsført produkter å erklære. Dette endrer ikke forfatternes tilslutning til alle PLoS ONE politikk på deling av data og materialer. Mens magekreft er den fjerde vanligste kreftformen i verden, er det den andre dødsårsaken. [1] Det er forekomsten er betydelig høyere i asiatiske land, inkludert Korea, hvor det er den nest vanligste kreftformen. [2] Nylig flere målrettede therapeutics for magekreft har blitt oppdaget, noe som gir flere alternativer for leger og pasienter [3] - [5] I den æra av målrettet terapi, mutasjon profilering av den utløsende kreft. er avgjørende for terapeutiske avgjørelser. Forsøk på å profilere mutasjoner har blitt laget med tradisjonelle Sanger-sekvensering; Det er imidlertid ikke en optimal metode i kliniske omgivelser på grunn av kostnad, tid og arbeidskraft som kreves. Dessuten krever Sanger-sekvensering vesentlige mengder av DNA; evaluering av små mengder av prøven i flere gener på samme tid er ikke mulig. Innføring av neste generasjons sekvense (NGS) metoder har løst dette problemet ved multiplex, high-throughput sekvensering av mange eksempler for flere gener samtidig. [6], [7] En av NGS plattformer, Ion Torrent AmpliSeq Cancer Panel, er avhengig av ikke-optisk deteksjon av hydrogenioner i en halvleder-enhet, [8] og er i stand til å oppdage 2,855 onkogene mutasjoner i 50 ofte muterte gener (tabell S1). Det er bedre enn andre massespektroskopi-basert sekvenseringsmetoder, og gir sekvense resultater raskere og til en lavere kostnad. [8] Den kan anvendes i formalinfikserte parafininnstøpte (FFPE) vevsprøver med små mengder DNA. Fordi det sikrer høy følsomhet i screening kjent onkogene mutasjoner, [9], [10] Ion Torrent AmpliSeq Cancer Panel er valget av 5 store kreft sentre i USA for molekylær diagnostikk i målrettet terapi [11]. Amplifisering av onkogener er en viktig mekanisme for genet overekspresjon og bidrar til tumorutvikling. [12] Eksempler er forsterkning av HER2 bedre tilpasset kreftbehandling kan forbedre pasientens utfall. Pasienttumorprøver vil være nødvendig for å karakterisere kreft på et molekylært nivå og identifisere sykdoms undergruppene som skal motta forskjellige behandlinger. Bruken av FFPE vev er viktig for å muliggjøre slike studier. [16] Her testet vi AmpliSeq og NCounter tilpassede CNV paneler i FFPE- magekreftprøver for å fastslå om de er aktuelt i arkiv kliniske prøver for personlig målrettet terapi. Samples Tumor celle prosent med mer enn 75% ble dissekert etter mikroskopi fra 4 mm ufargede seksjoner ved sammenligning med en H & E farget lysbilde, og genomisk DNA ble ekstrahert ved hjelp av en Qiagen DNA FFPE Tissue Kit (Qiagen, Hilden) i henhold til produsentens instruksjoner fra 96 pasienter med avansert magekreft. Etter ekstraksjon, målte vi konsentrasjon samt 260/280 og 260/230 nm ratio med spektrofotometer (ND1000, Nanodrop Technologies, ThermoFisher Scientific, MA, USA). Hver prøve ble deretter kvantifisert med qubit fluorometer (Life Technologies, Carlsbad, California). Genomisk DNA med > 10 ng målt ved qubit fluorometer ble utsatt for bibliotek forberedelse og syv prøvene ikke klarte å bygge biblioteker og ble ekskludert fra denne studien. Til slutt, 89 saker ble til slutt analysert og inkludert 31 kvinnelige og 58 mannlige pasienter. Tabell 1 viser kliniske og patologiske trekk ved pasientene i denne studien. Tilbakefall eller metastaser utviklet hos 11 pasienter med median oppfølgingstid på 76 måneder (variasjon 5,5 til 149,3). Studien ble godkjent av Institutional Review Board (IRB) på Samsung Medical Center. All klinisk undersøkelse ble gjennomført i henhold til de prinsipper som er nedfelt i Helsinkideklarasjonen. Den skriftlige samtykke ble frafalt av IRB grunn retrospektiv analyse og anonyme data. Prøver ble samlet som en del av en rutinemessig medisinsk prosedyre og ble samlet opp ved forfatterne for denne studien. Prøver fra avdøde pasienter eller levende pasienter ble alle avidentifisert, inkludert fjerning av enhver og all demografisk informasjon, før analyse og informert samtykkeskjema ble frafalt av IRB. Vi brukte Ion AmpliSeq Cancer Panel v2 (Ion Torrent) for å oppdage hyppige somatiske mutasjoner som ble valgt basert på litteraturgjennomgang. Den undersøker 2855 mutasjoner i 50 ofte muterte onkogener og tumorsuppressorgener (Tabell S1). Først 10 ng DNA fra hver av 89 FFPE- tumorprøver gikk single-tube, multipleks PCR forsterkning ved hjelp av Ion AmpliSeqCancer Primer Pool og Ion AmpliSeqKit reagenser (Life Technologies). Behandling av de resulterende amplikonene med FUPA Reagens delvis fordøyd primerene og fosforyleres til amplikonene. De fosforylerte amplikonene ble ligert til Ion Adaptere og renset. For strek bibliotek forberedelse, byttet vi strekkode adaptere fra Ion Xpress Barcode Adaptere 1-96 Kit for den ikke-strek adapter mix leveres i Ion AmpliSeq Library Kit. Den ligert DNA gikk nick-oversettelse og forsterkning for å fullføre sammenhengen mellom adaptere og amplikonene og til å generere tilstrekkelig materiale for nedstrøms mal forberedelse. To runder med Agencourt AMPure XP Reagens bindende 0.6 og 1.2 perle-til-prøve volumforhold fjernet innspill DNA og kommunefritt primere fra amplikonene. De endelige bibliotek molekyler var 125~300 bp i størrelse. Vi deretter overført bibliotekene til Ion OneTouch System for automatisert mal forberedelse. Sekvensering ble utført på Ion PGM sequencer i henhold til produsentens instruksjoner. Vi brukte IonTorrent Programvare for automatisert dataanalyse. For å måle sensitivitet og spesifisitet av kreft panel Ion AmpliSeq, hele exome sekvense resultater fra 4 magekreft prøver med kjent mutasjon status ble brukt [17]. NCounter Kopier nummer Variasjon CodeSets for deteksjon av CNV, NCounter Kopier nummer Variasjon CodeSets ble brukt sammen med 300 ng renset genomisk DNA ekstrahert fra 2-3 deler av 4-mikrometer tykke FFPE- representant tumor blokker med QIAamp DNA FFPE Tissue Kit (Qiagen, Hilden, Tyskland). DNA ble fragmentert via Alul fordøyelse og denaturert ved 95 ° C. Fragmentert DNA ble hybridisert med codeset av 86 gener i NCounter Cancer CN Assay Kit (Nanostring Technologies) i 18 timer ved 65 ° C og behandles i henhold til produsentens instruksjoner. Den NCounter Digital Analyzer telles og ordnet signalene fra reporter prober og gjennomsnittlig telle antall > 3 ble kalt og bekreftet av IHC, FISH eller real-time PCR IHC for HER2, EGFR (HER1) og CCNE1. for validering av CNV resultatene fra NCounter, utførte vi IHC for HER2 i alle tilfeller, og EGFR og CCNE1 i utvalgte saker. Etter deparaffinization og rehydrering, ble 4 mm seksjoner på silane-belagt lysbilder immunostained for HER2. Den HercepTest (Dako, Glostrup, Danmark) ble brukt i henhold til produsentens retningslinjer som tidligere beskrevet. [18] For EGFR vi brukte anti-NCL-L-EGFR-384 mus monoklonalt primært antistoff (1:100 fortynning, Novocastra /Vision Biosystems, Newcastle, UK) og for CCNE1 vi brukte anti-CCNE1 /Cyclin E1 antistoff (klone HE12; 1:200 fortynning, Thermo Fisher Scientific, MA). Ventana BenchMark XT automatisert slide-systemet for behandling ble brukt i henhold til produsentens protokoll. En ekspert patologen (KMK) evaluert resultatene FISH for HER2 FISH ble utført ved hjelp av dual-farge DNA-spesifikke prober fra PathVision ™ (Abbott /Vysis. LSI HER2 SpectrumOrange ™ og CEP 17 SpectrumGreen ™) som tidligere beskrevet i tilfeller med tvetydige økt forekomst av HER2. [19] Vi telte hybridisering signaler i 20 kjerner per prøve under et fluorescerende mikroskop (Zeiss Axioskop) ved hjelp av filtersett som anbefales av Vysis (DAPI /Spectrum Orange dual båndpass, DAPI /Spectrum Grønn dual båndpass). Alle overlappende kjerner ble ekskludert, og bare kjerner med en distinkt atom grensen ble evaluert. HER2 Vi brukte DNA hentet fra FFPE- magekarsinom tumorvev. Reaksjonsblandingen inneholdt 2 pl genomisk DNA-templat, 10 ul av Taqman universell PCR Hoved blanding (Applied Biosystems Inc., Foster City, CA) og 0,2 uM av hver primer. For nøyaktig påvisning av CN endringer, analyserte vi tre ulike regioner av KRAS Vi målte kopitall vinning ved hjelp av følgende profil: 2 minutter ved 50 ° C, denaturering ved 95 ° C i 10 min, etterfulgt av 40 sykluser på 95 ° C i 15 sekunder og 60 ° C i 1 min. Vi er fast bestemt relativ kvantifisering ved hjelp av 7900 HT rask real-time PCR system i fire. En RNaseP analysesett (Applied Biosystems) ble anvendt som en kontroll. Etter amplifikasjon importert vi resultatene fra eksperimentet inneholdende terskel-syklus verdier for kopiantallet og referanse assay inn i CopyCaller programvare (Applied Biosystems) for post-PCR analyse av data som tidligere beskrevet. [22] Vi tildelt CN gevinst status og antall KRAS Vi ekskluderte alle synonymt endringer etter en automatisert mutasjon-ringer algoritme ble brukt til å oppdage antatte mutasjoner. Tilbakevendende samtaler i mer enn 10 av 89 prøver ble ansett som falske positive og ble ekskludert. Vi brukte cutoff-verdier på mer enn 6% variant frekvens og mer enn X100 dekning for å oppdage ekte mutasjonsendringer i samsvar med tidligere studier og vår egen erfaring. [9], [10] Vi filtrert ut enkelt-nukleotid polymorfismer etter manuell gjennomgang av hver polymorfisme i Katalog av somatiske mutasjoner i Cancer (COSMIC, http://cancer.sanger.ac.uk/cancergenome/projects/cosmic) ( Figur 1). For kjente gener mutert i mage karsinom ( TP53 Resultater Resultater av Ion AmpliSeq kreft panel konsentrasjonen av DNA, sin konsentrasjon fold, gjennomsnittlig dekning av prøvene, totale antallet baser, > Q20 baser, leser, betyr lese lengde, kartlagt leser, på target-rate (%), var gjennomsnittlig dybde og ensartetheten av resultatene er beskrevet i Tabell S2. I alt fikk vi 8178 variant samtaler fra 89 prøver, blant dem 3554 samtalene var ikke synonymt endringer. Etter å filtrere ut gjentatte samtaler, < 6% av variant-allelet frekvens, < 100X dekning og de i intron-regionen, ble 65 variant samtaler valgt. I tillegg har vi gjennomgått automatiserte samtaler i kjente mutasjoner som BRAF I fire magekreft prøver med kjent mutasjon frekvenser bestemmes av hele exome sekvensering og bekreftet av Sanger-sekvensering, vi identifisert somatiske mutasjoner i TP53 Amplification by NCounter og validering av IHC, FISH eller real-time PCR Amplifikasjoner av HER2 I real-time PCR for KRAS For MET Diskusjoner Ved å bruke Ion AmpliSeq v2 fant vi at 39 av 89 avanserte adenokarsinom prøvene inneholdt somatiske mutasjoner, viser at denne plattformen er lett anvendelig i arkiv FFPE- vevsprøver. TP53 mutasjonsfrekvenser i det kosmiske database avsløre vesentlige likheter med de dataene vi hentet fra denne studien:. TP53 product: (32%), PIK3CA product: (10%), KRAS product: (6%), APC product: (6%), CTNNB1 product: (5%), CDKN2A product: (5%), FBXW7 product: (5%), SMO product: (4%), ErbB2 plakater (2%) og STK11 Semiconductor-basert sekvensering har grunnleggende forskjeller i sensing og signaloverføring i forhold til massespektrometri basert sekvensering. I stedet for å bruke optiske metoder for å oppdage nucleotide endringer, oppfatter halvlederbasert teknikk pH-endringer etter utgivelsen av protoner (H +) når nukleotidene integreres i den voksende DNA-tråden. [8] Derfor er det en betydelig reduksjon i kostnadene og tiden som er nødvendig for databehandling i forhold til andre NGS plattformer. Gir rask og nøyaktig informasjon om mutasjoner ved lav pris er avgjørende for pasienter med svært aggressive kreftformer, blant annet magekreft. I denne studien har vi manuelt anmeldt automatiserte samtaler i kjente mutasjoner etter påføring cutoff verdier av frekvens og dekning, deretter legge til to samtaler med lav-variant dekning. Selv om deres dekningsverdiene nådde ikke våre første kriterier, deres frekvenser overskredet vår første innstilling (6%) og kvaliteten av dataene var god. Dette understreker viktigheten av manuell gjennomgang etter automatisert screening. Nylig publiserte resultater ved hjelp AmpliSeq som analyseplattform også understreke kompensasjon av screening data med manuell gjennomgang [9], [10]. Personlig målrettet terapi for avansert kreft er avhengig primært på konseptet "onkogen avhengighet" i hvilke multiple genetiske avvik er avhengige av en eller noen få gener for tumorcelle vedlikehold og overlevelse. [28] En åpen, internasjonal, fase 3, randomisert kontrollert toga (Trastuzumab for magekreft) prøve indikerte at trastuzumab i kombinasjon med kjemoterapi er en ny standard alternativ for pasienter med HER2-positiv avansert mage eller gastro-esophageal krysset kreft. [29] En preklinisk studie viste at en undergruppe av magekreft med EGFR I sammendraget, vi vellykket utført basert på halvledere, sekvensering og NCounter CNV analyser i FFPE- vevsprøver fra 89 gastric adenokarsinomer. Høy throughput sekvensering og CNV screening i arkiv kliniske prøver muliggjør raskere, mer nøyaktig og kostnadseffektiv påvisning av hotspot mutasjoner og CNV i gener. I den æra av personlige genom medisin, planlegger vi å bruke disse verktøyene til å screene for magekreft pasienter som kan ha nytte av målrettet terapi.
, CCNE1
, MYC
, KRAS Hotell og EGFR
gener ble observert hos 8 (8,9%) , 4 (4,5%) 2 (2,2%) 1 (1,1%) og 1 (1,1%) tilfeller, henholdsvis. I tilfellene med forsterkning, fluorescens in situ hybridisering for HER2
bekreftet genamplifisering og immunhistokjemi for HER2, EGFR og CCNE1 verifisert overekspresjon av proteiner i kreftceller. I konklusjonen, vi vellykket utført basert på halvledere, sekvensering og NCounter kopiantall variasjon analyser i formalinfiksert parafin-embedded magekreftprøver. Høy throughput screening i arkiv kliniske prøver muliggjør raskere, mer nøyaktig og kostnadseffektiv påvisning av hotspot mutasjoner eller forsterkning i gener
Innledning
, MET
, FGFR2 Hotell og KRAS
gener i mage kreft. [13], [14] I deteksjon av kopinummervariasjoner (CNVs) i kliniske prøver, fluorescens in situ hybridisering (FISH) og /eller immunhistokjemi (IHC) har blitt mye brukt. Men høye kostnader og små utvalg av biopsimateriale begrense anvendelsen av disse metodene, og det er fortsatt behov for ytterligere high-throughput teknologi med lett tilgjengelighet, høy følsomhet og lave kostnader. NCounter CNV CodeSets (Nanostring teknologi, biovitenskap, Seattle, WA) gir overlegen nøyaktighet og reproduserbarhet for studier av alle størrelser og produsere bedre, raskere resultater med vesentlig mindre innsats enn med sanntids kvantitativ polymerase chain reaction (qPCR) eller CNV arrays [ ,,,0],15].
Materialer og metoder
Ion AmpliSeq kreft panel v2
genet ble ansett forsterket når FISH signal forholdet mellom HER2 /CEP17
var større enn eller lik 2,0 [20].
Real-time PCR for KRAS og MET forsterkning
gen: en region i intron 1 (TaqMan Kopier nummer analyse Hs06943812_cn), en region i intron 2 (Hs002534878_cn), og en region i ekson 6 (Hs02739788_cn). . For MET
genet, vi brukte primerene som tidligere beskrevet [21]
kopier basert på samstemmighet av resultatene i minst to av de tre sonder.
Analytiske metoder
, APC
, PIK3CA
, STK11
, CDKN2A
, KRAS
, HRAS
, BRAF Hotell og CTNNB1
), ble en manuell gjennomgang av automatiserte ringer resultater utført for å fange skadelige mutasjoner med litt lav- variant frekvens.
, KRAS Hotell og PIK3CA Hotell og kan spare to variant samtaler, som ble ekskludert under filtrering prosesser. Trettini av de 89 prøver (43,8%) hadde minst én mutasjon (figur 1). To tilfeller viste 22 og 5 mutasjoner, henholdsvis. Sistnevnte tilfelle næret MLH1
somatisk mutasjon [missense mutasjon i ekson 20: c.1147A > G (p.M383 V)] og MLH1
arrangøren hypermethylation med MLH1 protein tap ved IHC ved bruk av tidligere beskrevne fremgangsmåter, [23] som tyder hypermutated tumor. Imidlertid, selv om mutasjoner som finnes i det førstnevnte tilfellet føres variant frekvens og dekning kuttet offs, disse mutasjoner ble ikke bekreftet av Sanger-sekvensering, noe som tyder på falske positive i dette tilfellet på grunn av dårlig kvalitet av DNA. Så ble dette tilfelle utelukkes fra slutt analyser av resultatene. Ofte oppdages somatiske mutasjoner inkludert TP53 plakater (24 tilfeller, 27,0%), APC plakater (9 tilfeller, 10,1%), PIK3CA plakater (5 tilfeller, 5,6%), %), KRAS
(3 tilfeller, 3,4%), SMO plakater (4 tilfeller, 4,5%), STK11 plakater (3 tilfeller, 3,4%), CDKN2A
(3 tilfeller, 3,4%), og SMAD4 plakater (3 tilfeller, 3,4%) som vist i Tabell 2. Tabell 2 også oppsummert aminosyre endringer i ofte muterte gener. Vi identifiserte 19 pasienter (21,3%) med to eller flere unike og ledsagende somatiske mutasjoner.
, ErbB4 Hotell og CTNNB1
uten falske positive samtaler i andre gener (Tabell S3).
, CCNE1
, MYC
, KRAS Hotell og EGFR
gener ble observert i 8 (8,9%) 4 (4,5%) 2 (2,2%) 1 (1,1%) og 1 (1,1%) tilfeller, henholdsvis (tabell 3). Vi observerte ikke forsterkning av MET
, FGFR2
, CDK4 Hotell og CDK6 i noen av tilfellene. I tilfeller med forsterkning, IHC for HER2, EGFR og CCNE1 viste overekspresjon av proteiner i kreftceller (figur 2A, B og C). I ett tilfelle med HER2 2+ av HercepTest, FISH viste heterogen forsterkning av HER2
gener (figur 2D).
, ett tilfelle med forsterkning viste økt kopiantall (36, 37 og 49); i saker som var negative for KRAS
forsterkning, var det ingen økning av kopiantall (0,9 til 2,4, betyr 1,4).
genet, finner vi ingen Ved positiv på grunn av sin sjeldenhet [21]. Derfor har vi brukt ekstra ti (fem hver forsterket og ikke-forsterket) magekreftprøver og MET
forsterket mage kreft cellelinjer (MKN45 og SNU5) med kjente kopiantall og mRNA beløp. CNVs oppdaget av NCounter korrelert godt med kopiantall påvist ved real-time PCR (tabell S4) og mRNA nivåer av MET
genet (Pearsons korrelasjon test; r = 0,874, p = 0,001). (Figur S2)
ble oftest funnet mutasjon, etterfulgt av APC
, PIK3CA Hotell og KRAS
. Videre vår skikk CNV panel hell oppdaget CN økning av HER2
, CCNE1
, MYC
, EGFR Hotell og KRAS
gener, som vi bekreftet av IHC og real-time PCR
(2%). Siste hele exome sekvense studier på adenokarsinom i ventrikkel viste noe høyere frekvenser av TP53 plakater (36% og 73%) og PIK3CA
(14% og 20%) mutasjoner i forhold til våre resultater. [24], [25] Selv om våre mutasjons frekvensene var lavere sammenlignet med exome sekvenseringsresultater, var det en betydelig økning i forhold til våre tidligere data for massespektrometri-baserte OncoMap v4. [26] Både AmpliSeq og OncoMap påvise mutasjoner i hotspot regioner, som forklarer funn av sjeldnere mutasjon i noen onkogener og tumorsuppressorgener. Figur S1 sammen AmpliSeq v2 og OncoMap v4 i påvisemutasjons profiler. Tidligere OncoMap tester i 237 mage adenokarsinomer avdekket at PIK3CA
mutasjoner var hyppig i fremskredne stadier av sykdommen (5,1% i Stage IV, 6,4% i stadium II /III, 2,4% i stadium IB). [26] I denne studien observerte vi tre PIK3CA
mutasjoner hos pasienter med stadium III og to i stadium II sykdom, som støtter deres biologiske rolle i tumorprogresjon. Vi har også observert HER2 product: ( ErbB2
) c.2524G > A (V842I) mutasjon i et tilfelle av magekreft. I prekliniske studier cellelinjer som bærer den V842I mutasjon var resistente mot trastuzumab, men var følsom for irreversibel HER2-hemmer, neratinib [27].
eller MET
forsterkning og overekspresjon reagerer på cetuximab eller MET reseptortyrosinkinasehemmer terapi. [30] I tillegg presiseringer av cellesyklus megler CCNE1
antyder at potensialet for terapeutisk hemming av cyclin-avhengige kinaser i mage kreft. [31] Screening amplifiserte gener for målrettet behandling med high-throughput-teknologi er meget viktig. Tradisjonelle metoder som fisk og utvalg komparativ genomisk hybridisering lider av lav oppløsning på genomiske regioner, høye kostnader og er arbeids-og tidkrevende. [32] I denne første studien på NCounter CNV analyser, fant vi at denne teknologien er aktuelt i FFPE- kliniske prøver, og vi validert resultatene av IHC, FISH og real-time PCR. Selv om vi ikke validere alle genene som brukes i de tilpassede primere, valideringsresultater i flere utvalgte gener var bemerkelsesverdig.
Hjelpemiddel Informasjon
Figur S1. Host Sammenligning av dekning av Ion AmpliSeq v2 kreft panel versus Oncomap v4
doi:. 10,1371 /journal.pone.0111693.s001 plakater (TIF)
Figur S2.
Tomter av korrelasjon mellom MET
CNVs oppdaget av NCounter og mRNA nivåer av MET
genet ved real-time PCR
doi:. 10,1371 /journal.pone.0111693.s002 product: (TIF)
Tabell S1.
Gene Liste for Ion Torrent AmpliSeq Cancer Panel
doi:. 10,1371 /journal.pone.0111693.s003 plakater (XLS)
Tabell S2.
Konsentrasjonene av DNA, deres konsentrasjon fold, gjennomsnittlig dekning av prøvene, totale antallet baser, > Q20 baser, leser, betyr lese lengde, kartlagt leser, on-target rate (%), var gjennomsnittlig dybde og ensartethet.
doi: 10,1371 /journal.pone.0111693.s004 plakater (XLS)
tabell S3.
Somatiske mutasjoner i TP53
, ErbB4 Hotell og CTNNB1
doi:. 10,1371 /journal.pone.0111693.s005 plakater (XLS)
Tabell S4.
CNVs oppdaget av NCounter korrelert godt med kopiantall påvist ved real-time PCR
doi:. 10,1371 /journal.pone.0111693.s006 plakater (XLSX)
 Forskere løser mystisk tilfelle av auto bryggerisyndrom
Forskere løser mystisk tilfelle av auto bryggerisyndrom
 Forskere henter ut et fullstendig menneskelig genom fra en tusenvis av år gammel "tyggegummi"
Forskere henter ut et fullstendig menneskelig genom fra en tusenvis av år gammel "tyggegummi"
 Hvorfor bør du inkludere naturlige fiberkilder i kostholdet ditt?
Hvorfor bør du inkludere naturlige fiberkilder i kostholdet ditt?
 Flagger potensielle molekylære prediktorer for respons på biologiske terapier ved ulcerøs kolitt
Flagger potensielle molekylære prediktorer for respons på biologiske terapier ved ulcerøs kolitt
 Ny forskning identifiserer en kobling mellom tarmmikrobiomet og slag
Ny forskning identifiserer en kobling mellom tarmmikrobiomet og slag
 Tiopuriner kan hjelpe til med å stoppe virusreplikasjon hos menneskelige koronavirus
Tiopuriner kan hjelpe til med å stoppe virusreplikasjon hos menneskelige koronavirus
 Halvparten av stoffene i bruk skader tarmbakterier,
sier ny studie En oppsiktsvekkende presentasjon på UEG Uke 2019, i Barcelona, avslører at tarmmikrobiomet har høy risiko for skader hver gang vi bruker et stoff fra en eller flere av 18 ofte brukte gr
Halvparten av stoffene i bruk skader tarmbakterier,
sier ny studie En oppsiktsvekkende presentasjon på UEG Uke 2019, i Barcelona, avslører at tarmmikrobiomet har høy risiko for skader hver gang vi bruker et stoff fra en eller flere av 18 ofte brukte gr
 Blodprøve for mikrobielt DNA kan advare om kreft
En ny studie publisert i tidsskriftet Natur 11. mars, 2020, kan transformere det nåværende syn på kreftårsak og diagnose. Forskerne har kommet med en ny teknikk for å oppdage tilstedeværelse og type
Blodprøve for mikrobielt DNA kan advare om kreft
En ny studie publisert i tidsskriftet Natur 11. mars, 2020, kan transformere det nåværende syn på kreftårsak og diagnose. Forskerne har kommet med en ny teknikk for å oppdage tilstedeværelse og type
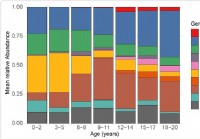 Endring av mikrobiom i øvre luftveier hos barn relatert til følsomhet for SARS-CoV-2
Forskere testet nasofaryngeal mikrobiom hos barn opp til 21 år og fant mikrobiomet endres med alderen. Spesifikke bakterier, hvis overflod også endres med alderen, er assosiert med alvorlig akutt resp
Endring av mikrobiom i øvre luftveier hos barn relatert til følsomhet for SARS-CoV-2
Forskere testet nasofaryngeal mikrobiom hos barn opp til 21 år og fant mikrobiomet endres med alderen. Spesifikke bakterier, hvis overflod også endres med alderen, er assosiert med alvorlig akutt resp