Этот метод «отпечатка пальца» микробиома помог выявить материнские источники микробов для микробиомов человеческого младенца или детеныша мыши, а также демонстрирует крайнюю стойкость штаммов кишечных микробов у взрослых близнецов, которые жили раздельно после сожительства в течение десятилетий.
Теперь Ку и Морроу направили свои исследования стабильности штаммов микробов на человеческих младенцев и детей, возраст вскоре после рождения (около 6 месяцев) до 6 лет. В основном, они обнаружили, что по мере развития микробиомов кишечника новорожденных существовали индивидуальные модели специфичности микробных штаммов.
Микробная экосистема кишечника младенца начинается с кратковременных изменений микробного состава, которые в конечном итоге превращаются в стабильный микробный состав. В центре внимания данного исследования. Эти стабильные взаимодействия микроб-хозяин необходимы для эффективного переваривания пищи. здоровое развитие иммунитета и устойчивость к колонизации болезнетворными микроорганизмами.
<цитата>В основном, в сообществе ранних кишечных микробов преобладают микробы, которые могут питаться углеводами, присутствующими в грудном молоке или смесях, Такие как Bifidobacterium adolescentis .
Когда младенец растет, переход на твердую пищу и физический рост приводят к изменению пространственной структуры кишечника, что способствует изменению физической и химической среды, что обеспечивает новые возможности экологической ниши для роста штаммов микробов. Этот переход экосистемы коррелирует с появлением Bacteroidetes, таких как Bacteroides vulgatus - в структуре микробного сообщества кишечника ".
Кейси Морроу, Кандидат наук., Университет Алабамы в Бирмингеме
Исследователи UAB применили свой метод отпечатков пальцев микробов к двум наборам данных секвенирования метагеномной ДНК:из ранее опубликованных исследований других авторов, образцов кала младенцев и детей младшего возраста, собранных в виде временного ряда. В первой группе из 31 ребенка образцы были собраны вскоре после рождения и через 1 год. 2-х и 3-х летнего возраста.
Четырнадцать из этих детей прошли несколько курсов лечения антибиотиками, которые могут нарушить микробиом кишечника; у остальных антибиотиков не было. Второй набор данных включал девять младенцев в возрасте от 6 месяцев до 6 лет; четверо из девяти получили несколько антибиотиков.
Из 17 младенцев в первом наборе данных, которые не получали антибиотики в течение трех лет после рождения, для стабильных и нестабильных штаммов микробов наблюдалась специфическая для младенцев картина. Только у одного младенца не было идентифицировано стабильных штаммов из 20 проанализированных видов бактерий. Для 14 младенцев, получавших несколько доз антибиотиков, 10 показал уникальный образец транзиторных штаммов, которые появлялись на короткое время после нескольких курсов лечения антибиотиками.
Для второго набора данных исследователи UAB проанализировали стабильность штаммов кишечных микробов Bacteroides vulgatus а также Bifidobacterium adolescentis до шести лет после рождения. Они обнаружили индивидуальные специфические паттерны различных доминирующих штаммов микробов, которые не зависели от воздействия антибиотиков и способа рождения.
Важно отметить, что не было очевидной связи между изменениями деформации в Б. vulgatus а также B. adolescentis . Например, у одного младенца, получавшего несколько антибиотиков, было ограниченное изменение Б. vulgatus напряжения как B. adolescentis штаммы сильно изменились, в то время как у другого ребенка, получавшего несколько антибиотиков, наблюдалась противоположная картина изменений деформации.
Исследователи также увидели несколько примеров кратковременного изменения штамма микробов, без лечения антибиотиками, с последующим восстановлением до доминирующего штамма. Хотя движущая сила этих изменений неизвестна, было несколько случаев, когда характерная для младенцев полная смена деформации на Б. vulgatus и для B. adolescentis произошел. Метаданные для этих младенцев не показали очевидной корреляции этих изменений с полом, страна происхождения, способ доставки или разовьется ли у младенца диабет.
«Результаты нашего анализа с использованием обоих наборов данных подчеркивают, что изменение штамма микробов является неотъемлемой частью развивающейся микробной экосистемы кишечника младенца, «Ку и Морроу сказали». результаты нашего исследования подтверждают использование метода отслеживания штаммов для мониторинга развития стабильного и здорового микробного сообщества ».
Завтра, Ку и его коллеги использовали свой инструмент для снятия отпечатков пальцев микробов в предыдущих исследованиях по отслеживанию деформации. В 2017 г. они обнаружили, что микробы доноров фекалий, используемые для лечения пациентов с рецидивирующими инфекциями Clostridium difficile, оставались у реципиентов в течение месяцев или лет после трансплантации фекалий.
В 2018 г. они показали, что изменения в верхних отделах желудочно-кишечного тракта в результате хирургии ожирения привели к появлению новых штаммов микробов. В 2019 г. они проанализировали стабильность новых штаммов у людей после лечения антибиотиками, и ранее в этом году они провели исследование взрослых близнецов, который показал, что близнецы разделяли определенное напряжение или напряжение между каждой парой в течение нескольких лет, и даже десятилетия, после того, как они начали жить отдельно друг от друга.
Также в этом году они показали, что индивидуализированная мозаика микробных штаммов была передана микробиому кишечника младенца от матери, рожающей через естественные родовые пути, как было проанализировано в парах мать-дитя, а также самки мышей и детенышей.
 Лечение целиакии
Лечение целиакии
 Некоторые виды бактерий могут увеличить риск заражения ВИЧ у женщин,
Некоторые виды бактерий могут увеличить риск заражения ВИЧ у женщин,
 Использование FLUOstar Omega для изучения новых кишечных бактерий, которые могут влиять на наше здоровье
Использование FLUOstar Omega для изучения новых кишечных бактерий, которые могут влиять на наше здоровье
 Нил Белл назначен директором по развитию Avacta Life Sciences
Нил Белл назначен директором по развитию Avacta Life Sciences
 Исследователи используют фаговую терапию для успешного лечения алкогольной болезни печени.
Исследователи используют фаговую терапию для успешного лечения алкогольной болезни печени.
 Панкреатит
Панкреатит
 Рыбная слизь может быть потенциальным источником антибиотиков, результаты исследования
С появлением бактерий с множественной лекарственной устойчивостью и отсутствием новых антибиотиков, исследователи ищут новые молекулы, которые могут бороться с этими патогенами. Группа исследователей
Рыбная слизь может быть потенциальным источником антибиотиков, результаты исследования
С появлением бактерий с множественной лекарственной устойчивостью и отсутствием новых антибиотиков, исследователи ищут новые молекулы, которые могут бороться с этими патогенами. Группа исследователей
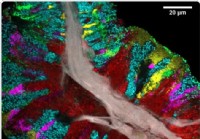 Подробная карта микробиома человеческого языка
Новое исследование, опубликованное в журнале Отчеты по ячейкам в марте 2020 года сообщает об использовании передовых и быстрых методов спектральной визуализации для разработки подробной карты микроб
Подробная карта микробиома человеческого языка
Новое исследование, опубликованное в журнале Отчеты по ячейкам в марте 2020 года сообщает об использовании передовых и быстрых методов спектральной визуализации для разработки подробной карты микроб
 Биологическая терапия может снизить риск тяжелой формы COVID-19
Недавнее обсервационное исследование испанских исследователей, в настоящее время доступно на medRxiv * сервер препринтов, предполагает, что пациенты с иммуноопосредованными заболеваниями, получающие
Биологическая терапия может снизить риск тяжелой формы COVID-19
Недавнее обсервационное исследование испанских исследователей, в настоящее время доступно на medRxiv * сервер препринтов, предполагает, что пациенты с иммуноопосредованными заболеваниями, получающие