DNA Kopie Zuel Profiler vun gastric Kriibs Virleefer lesions VerfÜgung méiglech VerfÜgung Background VerfÜgung Chromosomal Instabilitéit (Cin) ass déi verwandelt Typ vu Frankräich Onstabilitéit an gastric erhéijen, mä hir Roll an malignant Transformatioun vun der gastric mucosa nach obskur ass . Am Moment studéieren, setzen mir eraus ze studéieren, ob zwou morphologically verschidde Kategorien vu gastric Kriibs Virleefer lesions, dh intestinal-Typ an pyloric drüs adenomas, verschidden Musteren vun DNA Kopie Zuel Ännerungen droen hätt, méiglecherweis z'ënnerscheedde genetesch Curriculum vun gastric carcinogenesis an dëse Spigelteleskopen zwee adenoma Typen. VerfÜgung Resultater VerfÜgung engem 5K BAC vill CGH Plattform benotzt, mir weisen, dass déi gemeinsam geometrescher Figur vun der 11 intestinal-Typ gedeelt an 10 pyloric drüs adenomas Gewënn vun chromosomes 9 (29%) waren, 11q ( 29%) a 20 (33%), an Perte vun chromosomes 13q (48%), 6 (48%), 5 (43%) a 10 (33%). Déi heefegsten geometrescher Figur vun intestinal-Typ sech gastric adenoma Iwwerhand iwwert 11q, 9q an 8, a Verloschter op chromosomes 5Q, 6, 10 an 13, hierkommen an pyloric drüs gastric adenomas dës goufen Iwwerhand iwwert chromosome 20 a Verloschter op 5Q a 6. allerdéngs huet keng grouss Ënnerscheeder tëscht den zwee adenoma Zorte observéiert. VerfÜgung Conclusioun VerfÜgung d'Resultater hindeit, datt Iwwerhand iwwert chromosomes 8, 9q, 11q an 20, a Verloschter op chromosomes 5Q, 6, 10 an 13, wahrscheinlech bedeiten fréi Evenementer zu gastric carcinogenesis. D'phenotypical gouffen, intestinal-Typ an pyloric drüs adenomas Ee, ënnerscheeden do net vill (P = 0,8) um Niveau vun DNA Kopie Zuel Ännerungen. VerfÜgung Background VerfÜgung Gastric Kriibs ass déi zweet heefegsten malignancy weltwäit an der Iwwregens hätt vun dëser malignancy bleift ganz schlecht [1]. Gastric Kriibs Heefegkeet a veruerteelt Tariffer ënnerscheeden tëschent verschiddene Länner bannen der europäescher Unioun [2]. An Holland ass geklommen fënneften als Ursaach vum Kriibs Doud, mat ronn 2.200 nei Fäll all Joer [3]. Befënnt mat curative beduecht ass d'Behandlung vun presentéiert zu fortgeschratt Fäll vun gastric Kriibs hierkommen lokal Bild mucosectomy kann am Ufank gastric Kriibs curative ginn. Erkennen an Entféierung vun gastric neoplasias an eng fréi oder souguer premalignant Staat wäert droen Doud wéinst gastric Kriibs ze reduzéieren. Fir dëst Zil z'erreechen sinn, besser Tester fir fréi ze erkennen gastric Kriibs waren, an e bessere Verständnis vun der Biologie vun gastric Kriibs Werdegang ass wichteg an dat respektéieren. VerfÜgung Laut der Correa Modell, pathogenesis vun intestinal-Typ gastric adenocarcinoma follegt enger Passerelle vun chronescher aktiv gastritis wéinst Helicobacter pylori VerfÜgung Wonn, Spëtzekandidat vun intraepithelial neoplasia an endlech invasiv adenocarcinoma duerno atrophy, intestinal metaplasia zu mucosal [4]. Genetesch characterization vun Otemschwieregkeeten Echantillon vun intraepithelial neoplasia Etapp géif zu eisem Verständnis vun de molekulare pathogenesis vun gastric Kriibs méi cotiséieren. Allerdéngs sinn nëmmen dës lesions selten fonnt, méiglecherweis wéinst rapid Werdegang duerch dës Etapp Kriibs, a si meeschtens Moment nëmmen an Deeler vun Fro uplanzen, Frankräich Analys vun dësen lesions hampering. Analyse vun alternative Virleefer lesions dofir kéint, op d'mannst zum Deel, eng Bänk. Entwécklung vun gastric Kriibs duerch eng adenoma Etapp, obwuel manner heefeg, ass esou alternative Wee. Dës adenomas sinn heiansdo während gastroscopy a presentéieren wéi grouss lesions festgestallt dass histologically weisen Zollformalitéiten-epithelial neoplasia, déi se gëeegent fir Frankräich Analys mécht. Gastric adenomas hunn eng direkt malignant Potential an Kont fir ongeféier 20% vun all epithelial polyps [5, 6]. Gastric adenomas kann eng klassesch Bléien hunn, tubulovillous, oder villous Wirklechkeet mat engem Kannerbetreiung intestinal-Typ epithelium, mee kann och als pyloric drüs adenomas schéngen [6]. Pyloric drüs adenomas entstoen déif mucoid Glands am Mo a si betraff positiv fir mucin 6 [7, 8]. A grousser Zuel vun gastric adenomas weisen schon Werdegang ze adenocarcinoma. Um éischte Diagnos ronn 30-40% vun all pyloric drüs adenomas weisen schon e Schwéierpunkt vun carcinoma [9, 10]. Fir intestinal-Typ adenomas ass dës Zuel méi déif an schwankt vun 28,5% fir villous adenomas an 29,4% fir tubulovillous Typ adenomas ze nëmmen 5,4% an d'Bléien adenomas [11]. Béid adenocarcinomas, ex intestinal-Typ adenomas an ex pyloric drüs adenomas, glandular Strukturen weisen, am Géigesaz Typ gastric Kriibs ze diffusen. Eng wichteg Neierung an der pathogenesis vun stäerkste gastric Cancers VerfÜgung, wéi a villen anere staark Cancers, ass chromosomal Onstabilitéit , an Dumouriez a Verloschter vun Deeler oder souguer ganz chromosomes doraus [12]. Dës chromosomal Ännerunge kënnt vun Komparativ Frankräich hybridization (CGH) analyséiert ginn. Puer Joer virdrun Studien hunn genetesch hire Restaurant zu gastric adenomas mat dëser Technik fonnt, Iwwerhand iwwert chromosome 7q Wiesen, 8q, 13q, 20q, a Verloschter op chromosome 4P, 5Q, 9p 17p an 18q [13-16]. Obwuel Verschiddenheet an nëmmen zu adenomas mat Héichwäertege intraepithelial neoplasia observéiert, héije Niveau amplifications goufen op chromosomes 7q fonnt, 8p, 13q, 17q an 20q [13-16]. An gastric adenocarcinomas, konsequent beschriwwen chromosomal geometrescher Figur sinn Iwwerhand iwwert chromosome 3q, 7P, 7q, 8q, 13q, 17q an 20q a Verloschter op chromosome 4q, 5Q, 6q, 9p, 17p an 18q. Haut Niveau amplifications hunn op 7q ërem fonnt goufen, 8p, 8q, 17q, 19q an 20q [14, 17-23]. Nach, chromosomal geometrescher Figur, oder DNA Kopie Rei Ännerungen, sinn net eenheetlech an gastric Kriibs [24]. Subgroups mat verschiddene Musteren vun DNA Kopie Zuel hire Restaurant kann unerkannt ginn, deen och mat de Medeziner Resultat gin verbonne gewise goufen [25]. Am Moment studéieren VerfÜgung, setzen mir eraus ob zwee morphologically verschidde Kategorien vu gastric Kriibs ze studéieren Virleefer lesions, dh intestinal-Typ an pyloric drüs adenomas, géif verschidden Musteren vun DNA Kopie Zuel Ännerungen droen, méiglecherweis z'ënnerscheedde genetesch Curriculum vun gastric carcinogenesis am zwou adenoma Zorte bidden. VerfÜgung Resultater VerfÜgung DNA Kopie Zuel Ännerungen décidéiert observéiert an 10 aus 11 intestinal-Typ adenomas an 9 aus 10 pyloric drüs adenomas. Déi heeschen Zuel vun chromosomal Evenementer, definéiert als Dumouriez a Verloschter, pro entholl war 6.0 (Rei 0-18), dorënner 2,9 (Rei 0-14) Dumouriez a 3.0 (Rei 0-7) Verloschter. An intestinal-Typ adenomas, war d'mengen Zuel vun chromosomal Evenementer pro entholl 6.5 (Rei 0-18) vun deenen 3,4 (Rei 0-14) Dumouriez a 3,1 (Rei 0-7) Verloschter, an am pyloric drüs adenomas der mëttlerer Zuele goufen respektiv 5.4 (Rei 0-9), 2,4 (Rei 0-7) an 3.0 (Rei 0-7). an der intestinal-Typ gastric adenomas VerfÜgung, observéiert d'Équipe gemeinsam geometrescher Figur sech Iwwerhand iwwert chromosomes 8, 9q an 11q, a Verloschter op chromosomes 5Q, 6, 10 an 13. Am véier adenomas (36.4%), kréien vun chromosome 11q23.3 war mat enger gemeinsamer Regioun vun iwwerlageren vun 2,6 Mb observéiert. Ufank vun chromosome 9q war zu véier adenomas (36.4%) mat engem 12,6 Mo gemeinsam Regioun vun iwwerlageren läit op chromosome 9q33.1-q34.13 observéiert. Ufank vun chromosome 8 war an dräi adenomas (31%) observéiert, zwee vun deenen zougedréckt gewannen vum ganze chromosome adenomas 8, an der drëtter adenoma zougedréckt engem Gewënn vun chromosome 8p-q22.3 mat engem zousätzleche 28,7 Mo gewannen op chromosome 8q24.11 -qter. Ausserdeem goufen Iwwerhand observéiert op chromosomes 1, 3, 6p, 7, 11p, 12P, 13q, 16, 17, 19, 20 a 22q. Nee amplifications waren an der intestinal-Typ adenomas. VerfÜgung geläscht op chromosome 13 goufen observéiert zu siwen intestinal-Typ adenomas (64%) gesinn. Vun dësen, awer fënnef engem 11,9 Mo Läsche vun chromosome 13q21.2-21.33 mat engem zousätzleche 7,7 Mb Läschen op chromosome 13q31.1-31.3. Déi aner zwee adenomas zougedréckt engem 16,6 Mo Läsche vun 13q14.3-31. A Auteuren op chromosome 6 war zu sechs adenomas (55%), mat enger phpNuke Regioun vun 68,9 Mo läit op 6cen-q22.1 observéiert. A Läsche vun chromosome 5Q war zu véier adenomas (36%) mat engem gemeinsamen Regioun vun iwwerlageren läit op chromosome 5q22.1-q23.2 observéiert. Ausserdeem, war en Extrait vum ganze chromosome 10 zu véier adenomas observéiert (36%). Aner observéiert Verloschter am intestinal-Typ adenomas waren wellkomm op chromosomes 8q, 9p, 10, 12q, 20q a 21. Eng Iwwersiicht vun all DNA Kopie Zuel geometrescher Figur vun der intestinal-Typ adenomas ass an Table 1.Table 1 Iwwersiicht vun der DNA dës Kopie Rei Ännerungen an 11 intestinal-Typ adenomas VerfÜgung
zu Chromosomal geometrescher Figur
zu Reliefsailen clones
zu zu klinescher ID
Iwwerhand
Verloschter
Segment Gréisst (Mo) VerfÜgung VerfÜgung Start
End
1 VerfÜgung 1p-p36.11 VerfÜgung 26,68 VerfÜgung RP11-465B22 VerfÜgung RP1-159A19 VerfÜgung 5q13.2-Q23 .2 VerfÜgung 55,26 VerfÜgung RP11-115I6 VerfÜgung CTB-1054G2 VerfÜgung 6p21.33-p21.1 VerfÜgung 13,78 VerfÜgung RP11-346K8 VerfÜgung RP11-227E22 VerfÜgung 6p21.1 -q16.1 VerfÜgung 52,05 VerfÜgung RP11-89I17 VerfÜgung RP3-393D12 VerfÜgung 9q33.1-34.2 VerfÜgung 17.32 VerfÜgung RP11-27I1 VerfÜgung RP11-417A4 VerfÜgung 11q23.3 VerfÜgung 4,80 VerfÜgung RP11-4N9 VerfÜgung RP11-730K11 VerfÜgung 13q21.1-q31.3 VerfÜgung 39,63 VerfÜgung RP11-200F15 VerfÜgung RP11-62D23 VerfÜgung 2 VerfÜgung 1p -1p33 VerfÜgung 46,90 VerfÜgung RP11-465B22 VerfÜgung RP11-330M19 VerfÜgung 6p21.33-p21.1 VerfÜgung 14,12 VerfÜgung RP11-346K8 VerfÜgung RP11-121G20 VerfÜgung 6p21.1 -q16.2 VerfÜgung 54,91 VerfÜgung RP11-554O14 VerfÜgung RP11-79G15 VerfÜgung 8p-q22.3 VerfÜgung 105,67 VerfÜgung GS1-77L23 VerfÜgung RP11-200A13 VerfÜgung 8q24.11 -qter VerfÜgung 28,65 VerfÜgung RP11-278L8 VerfÜgung RP5-1056B24 VerfÜgung 9q33.1-q34.2 VerfÜgung 13,63 VerfÜgung RP11-85O21 VerfÜgung RP11-417A4 VerfÜgung 11p11.2 -q13.5 VerfÜgung 31,69 VerfÜgung RP11-58K22 VerfÜgung RP11-30J7 VerfÜgung 11q23.3 VerfÜgung 2,62 VerfÜgung RP11-4N9 VerfÜgung RP11-62A14 VerfÜgung 12q13.11-Q14 .1 VerfÜgung 10,57 VerfÜgung RP11-493L12 VerfÜgung RP11-571M6 VerfÜgung 13q21.1-q21.33 VerfÜgung 18,24 VerfÜgung RP11-200F15 VerfÜgung RP11-335N6 VerfÜgung 13q31.1 -q31.3 VerfÜgung 12,49 VerfÜgung RP11-533P8 VerfÜgung RP11-62D23 VerfÜgung 16p13.3-Q21 VerfÜgung 57,26 VerfÜgung RP11-243K18 VerfÜgung RP11-405F3 VerfÜgung 16q21-Q22 .1 VerfÜgung 5,97 VerfÜgung RP11-105C20 VerfÜgung RP11-298C15 VerfÜgung 16q22.1-q24.3 VerfÜgung 22,46 VerfÜgung RP11-63M22 VerfÜgung CTC-240G10 VerfÜgung 17
81,24 VerfÜgung GS1-68F18 VerfÜgung RP11-567O16 VerfÜgung 19 VerfÜgung 61,01 VerfÜgung CTB-1031C16 VerfÜgung GS1-1129C9 VerfÜgung 20q11.21-q11.23 VerfÜgung 5,09
RP3-324O17 VerfÜgung RP5-977B1 VerfÜgung 20q13.12-qter VerfÜgung 19,60 VerfÜgung RP1-138B7 VerfÜgung CTB81F12 VerfÜgung 3 VerfÜgung - VerfÜgung - VerfÜgung 4
6p21.1 VerfÜgung 3,32 VerfÜgung RP11-79J5 VerfÜgung RP11-121G20 VerfÜgung 6p12.3-q22.1 VerfÜgung 76,38 VerfÜgung RP11-79G12 VerfÜgung RP11-59D10 VerfÜgung 7 VerfÜgung 156,89 VerfÜgung RP11-510K8 VerfÜgung CTB-3K23 VerfÜgung 8q22.3-q23.3 VerfÜgung 9,69 VerfÜgung RP11-142M8 VerfÜgung RP11-261F23 VerfÜgung 9q33.1-Q34 .13 VerfÜgung 12,58 VerfÜgung RP11-55P21 VerfÜgung RP11-83N9 VerfÜgung 11q23.3 VerfÜgung 3,04 VerfÜgung RP11-4N9 VerfÜgung RP11-8K10 VerfÜgung 13q21.2-q21.33 VerfÜgung 17.05 VerfÜgung RP11-240M20 VerfÜgung RP11-77P3 VerfÜgung 13q31.1-q31.3 VerfÜgung 11,68 VerfÜgung RP11-400M8 VerfÜgung RP11-100A3 VerfÜgung 16q23.2-Q24 .3 VerfÜgung 8,92 VerfÜgung RP11-303E16 VerfÜgung RP4-597G12 VerfÜgung 20p-Q13.2 VerfÜgung 53,40 VerfÜgung CTB-106I1 VerfÜgung RP5-1162C3 VerfÜgung 20q13.31-qter VerfÜgung 8,06 VerfÜgung RP5-1167H4 VerfÜgung CTB-81F12 VerfÜgung 22q VerfÜgung 33,72 VerfÜgung XX-P8708 VerfÜgung CTB-99K24 VerfÜgung 5 zur VerfÜgung 12q24.31-qter
11,75 VerfÜgung RP11-322N7 VerfÜgung RP11-1K22 VerfÜgung 6 VerfÜgung 3 VerfÜgung 193,37 VerfÜgung RP11-299N3 VerfÜgung RP11-279P10 VerfÜgung 6cen-q24.1 VerfÜgung 88,49 VerfÜgung RP11-91E17 VerfÜgung RP11-86O4 VerfÜgung 7 VerfÜgung 156,09 VerfÜgung RP11-510K8 VerfÜgung RP11-518I12 VerfÜgung 8 VerfÜgung 144,26 VerfÜgung RP11-91J19 VerfÜgung RP5 -1118A7 VerfÜgung 13q21.1-q21.33 VerfÜgung 11,86 VerfÜgung RP11-640E11 VerfÜgung RP11-452P23 VerfÜgung 13q31.1-q31.3 VerfÜgung 9,62 VerfÜgung RP11-400M8
RP11-306O1 VerfÜgung 20q13.2-q13.31 VerfÜgung 1.41 VerfÜgung RP11-212M6 VerfÜgung RP4-586J11 VerfÜgung 7 VerfÜgung 5q21.1-qter VerfÜgung 80,52 VerfÜgung CTC -1564E20 VerfÜgung RP11-281O15 VerfÜgung 10 VerfÜgung 132,19 VerfÜgung RP11-29A19 VerfÜgung RP11-45A17 VerfÜgung 13q21.33-31.1 VerfÜgung 8,76m VerfÜgung RP11-209P2 VerfÜgung RP11 -470M1 VerfÜgung 8 VerfÜgung 5q22.1-q23.2 VerfÜgung 13,28 VerfÜgung RP11-276O18 VerfÜgung RP11-14L4 VerfÜgung 6p12.3-q22.1 VerfÜgung 74,37 VerfÜgung RP11 -89l17 VerfÜgung RP11-149M1 VerfÜgung 9p21.1-pter VerfÜgung 31,18 VerfÜgung RP11-147I11 VerfÜgung RP11-12K1 VerfÜgung 10 VerfÜgung 133,18 VerfÜgung RP11-10D13 VerfÜgung RP11 -45A17 VerfÜgung 13q14.3-q31.3 VerfÜgung 39,71 VerfÜgung RP11-211J11 VerfÜgung RP11-306O1 VerfÜgung 17 VerfÜgung 77,65 VerfÜgung GS1-68F18 VerfÜgung RP11-398J5
19 VerfÜgung 63,31 VerfÜgung CTC-546C11 VerfÜgung CTD-3138B18 20 VerfÜgung 60,87
RP4-686C3 VerfÜgung RP4-591C20 VerfÜgung 22q VerfÜgung 31,25 VerfÜgung XX -bac32 VerfÜgung cta-722E9 VerfÜgung 9 VerfÜgung 5q14.3-q23.2 VerfÜgung 33,06 VerfÜgung RP11-302L17 VerfÜgung RP11-14L4 VerfÜgung 6p22.2-q22.3
8,44 VerfÜgung RP11-91n3 VerfÜgung RP11-88h24 VerfÜgung 6p12.1-q24.1 VerfÜgung 88,89 VerfÜgung RP11-7h16 VerfÜgung RP11-368P1 VerfÜgung 8 VerfÜgung 145,95
GS1-77L23 VerfÜgung CTC-489D14 VerfÜgung 9q33.1-qter VerfÜgung 13,60 VerfÜgung RP11-91G7 VerfÜgung GS1-135I17 VerfÜgung 10 VerfÜgung 133,18 VerfÜgung RP11-10D13
RP11-45A17 VerfÜgung 11q23.3 VerfÜgung 3,16 VerfÜgung RP11-4N9 VerfÜgung RP11-215D10 VerfÜgung 13q14.3-qter VerfÜgung 58,59 VerfÜgung RP11-240M20 VerfÜgung RP11-480K16 VerfÜgung 20q13.2-q13.31 VerfÜgung 1,96 VerfÜgung RP11-55E1 VerfÜgung RP5-832E24 VerfÜgung 21cen-q21.3 VerfÜgung 17,39 VerfÜgung RP11-193B6 VerfÜgung RP11-41N19 VerfÜgung 10 VerfÜgung 8q22.3-q23.3 VerfÜgung 12,93 VerfÜgung RP11-142M8 VerfÜgung RP11-143P23 VerfÜgung 10 VerfÜgung 134,52 VerfÜgung RP11-10D13 VerfÜgung RP11-122K13 VerfÜgung 13q21.1-q21.33 VerfÜgung 18,03 VerfÜgung RP11-322F18 VerfÜgung RP11-335N6 VerfÜgung 13q31.1-q31.3 VerfÜgung 8,99 VerfÜgung RP11-533P8 VerfÜgung RP11 -505P2 VerfÜgung 11 VerfÜgung - VerfÜgung - VerfÜgung Déi heefegsten aberration zu pyloric drüs adenomas observéiert goufen Iwwerhand iwwert chromosome 20 a Verloschter op chromosomes 5Q a 6. Iwwerhand iwwert chromosome 20 waren zu véier adenomas gesinn (40 %). Dräi adenomas zougedréckt engem 9,8 Mb Gewënn vun chromosome 20q13.12-q13.33, an gewannen vun ganzt chromosome 20 war am anere adenoma observéiert. Ausserdeem goufen Iwwerhand gesinn op chromosomes 1, 3q, 5Q, 7, 9q, 11q, 12q, 13q, 15q, 17 an 22q. One pyloric drüs adenoma zougedréckt amplifications, läit op 12q13.2-q21.1 an 20q13.3-q13.33. VerfÜgung Five pyloric drüs adenomas (50%) zougedréckt Verloscht vun chromosome 5Q, zwee vun deenen eng ganz chromosome verluer haten Aarm, während zwou adenomas zougedréckt engem 22,4 Mo Läsche vun 5q11.2-Q13.3 an ee adenoma engem 40,3 Mo Läsche vun 5q21.1-q31.2. Verloscht vun chromosome 6 war zu véier pyloric drüs adenomas (40%) observéiert, dräi vun deenen eng komplett Verloscht vun 6q zougedréckt a eent adenoma zougedréckt engem 51,2 Mo Läsche vun 6p21.1-q16.3. Aner chromosomal Verloschter waren op chromosomes observéiert 1p, 2q, 4, 9p, 10, 12q 13q, 14q, 16, 18q, 20q, an 21. Eng Iwwersiicht vun DNA Kopie Zuel geometrescher Figur vun der pyloric drüs adenomas zu Table 2.Table dës ass 2 Iwwersiicht vun der DNA Kopie Rei Ännerungen an 10 pyloric drüs adenomas VerfÜgung zu Chromosomal geometrescher Figur
zu Reliefsailen clones
zu zu klinescher ID
Iwwerhand
Verloschter
Segment Gréisst (Mo )
End Start
12 VerfÜgung 1q21.3-q23.3 VerfÜgung 9,95 VerfÜgung RP11-98D18 VerfÜgung RP11-5K23 VerfÜgung
1q42.13-Q43 VerfÜgung 14,07 VerfÜgung RP11-375H24 VerfÜgung RP11-80B9 VerfÜgung 3q VerfÜgung 111,59 VerfÜgung RP11-312H1 VerfÜgung RP11-23M2 VerfÜgung 5q35.1-Q35 .3 VerfÜgung 9,11 VerfÜgung RP11-20O22 VerfÜgung RP11-451H23 VerfÜgung 6q VerfÜgung 115,76 VerfÜgung RP11-524H19 VerfÜgung RP5-1086L22 VerfÜgung 7 VerfÜgung 156,09 VerfÜgung RP11 -510K8 VerfÜgung RP11-518I12 VerfÜgung 17 VerfÜgung 77,48 VerfÜgung RP11-4F24 VerfÜgung RP11-313F15 VerfÜgung 20 VerfÜgung 63,47 VerfÜgung CTB-106I1 VerfÜgung CTB-81F12
zu - VerfÜgung - VerfÜgung 14 VerfÜgung 4 VerfÜgung 191,13 VerfÜgung CTC-963K6 VerfÜgung RP11-45F23 VerfÜgung 5Q VerfÜgung 128,59 VerfÜgung CTD-2276O24
RP11-281O15 VerfÜgung 14q VerfÜgung 83,81 VerfÜgung RP11-98N22 VerfÜgung RP11-73M18 VerfÜgung 16 VerfÜgung 89,71 VerfÜgung RP11-344L6 VerfÜgung RP4-597G12 VerfÜgung 20q13.2 -q13.33 VerfÜgung 10,84 VerfÜgung RP4-724E16 VerfÜgung CTB-81F12 VerfÜgung 15 VerfÜgung 9q33.2-q34.3 VerfÜgung 16,81 VerfÜgung RP11-57K1 VerfÜgung RP11-83N9 VerfÜgung 11q23.2-q24.3 VerfÜgung 16,04 VerfÜgung RP11-635F12 VerfÜgung RP11-567M21 VerfÜgung 12q14.3-Q15 VerfÜgung 2,58 VerfÜgung RP11-30I11 VerfÜgung RP11-444B24 VerfÜgung 20q13.31-q13.33 VerfÜgung 6,86 VerfÜgung RP5-1153D9 VerfÜgung RP5-963E22 VerfÜgung 22q VerfÜgung 32,53 VerfÜgung XX-p8708 VerfÜgung cta-722E9 VerfÜgung 16 VerfÜgung 9q33.3-qter VerfÜgung 13,57 VerfÜgung RP11-85C21 VerfÜgung GS1-135I17 VerfÜgung 10p12.1-qter VerfÜgung 110,28 VerfÜgung RP11-379L21 VerfÜgung RP11-45A17
11q23.1-q24.3 VerfÜgung 17,72 VerfÜgung RP11-107P10 VerfÜgung RP11-567M21 VerfÜgung 13q31.1-q32.1 VerfÜgung 10,84 VerfÜgung RP11-661D17 VerfÜgung RP11-40H10 VerfÜgung 20q13.2-q13.31 VerfÜgung 1,96 VerfÜgung RP11-55E1 VerfÜgung RP4-586J11 VerfÜgung 17 VerfÜgung 1p34.3-pter VerfÜgung 35,59 VerfÜgung RP1-37J18
RP11-204L3 VerfÜgung 1p33-qter VerfÜgung 203,62 VerfÜgung RP4-739H11 VerfÜgung RP11-551G24 VerfÜgung 2q31.1-qter VerfÜgung 66,00 VerfÜgung RP11-205B19 VerfÜgung RP11-556H17 VerfÜgung 5q21.1-q31.2 VerfÜgung 40,27 VerfÜgung CTD-2068C11 VerfÜgung RP11-515C16 VerfÜgung 5q31.3-qter VerfÜgung 39,06 VerfÜgung CTD-2323H12 VerfÜgung RP11-451H23 VerfÜgung 6q VerfÜgung 113,61 VerfÜgung RP11-89D6 VerfÜgung CTB-57H24 VerfÜgung 10 VerfÜgung 134,52 VerfÜgung RP11-10D13 VerfÜgung RP11-122K13 VerfÜgung 13q31.1-qter
36,14 VerfÜgung RP11-388E20 VerfÜgung RP11-245B11 VerfÜgung 20q13.2-qter VerfÜgung 11,24 VerfÜgung RP11-15M15 VerfÜgung RP5-1022E24 VerfÜgung 18 VerfÜgung 5q11.2-Q21 .2 VerfÜgung 51,24 VerfÜgung CTC-1329H14 VerfÜgung RP1-66P19 VerfÜgung 6p12.1-q16.3 VerfÜgung 51,24 VerfÜgung RP11-7H16 VerfÜgung RP11-438N24 VerfÜgung 9pter-Q13 VerfÜgung 66,82 VerfÜgung GS1-41L13 VerfÜgung RP11-265B8 VerfÜgung 10 VerfÜgung 133,04 VerfÜgung RP11-10D13 VerfÜgung RP11-45A17 VerfÜgung 13q21.1-q21.33 VerfÜgung 18,39 VerfÜgung RP11-240M20 VerfÜgung RP11-335N6 VerfÜgung 13q31.1-q31.3 VerfÜgung 12.45 VerfÜgung RP11-551D9 VerfÜgung RP11-100A3 VerfÜgung 21cen-q21.3 VerfÜgung 17,39 VerfÜgung RP11-193B6 VerfÜgung RP11-41N19 VerfÜgung 19 VerfÜgung 1p32.3-p21.1 VerfÜgung 50,40 VerfÜgung RP11-117D22 VerfÜgung RP5-1108M17 VerfÜgung 5q11.2-Q13 .3 VerfÜgung 24,64 VerfÜgung RP4-592P18 VerfÜgung CTD-2200O3 VerfÜgung 13q12.11-q14.3 VerfÜgung 31,58 VerfÜgung RP11-187L3 VerfÜgung RP11-327P2 VerfÜgung 15q12-Q26 .3 VerfÜgung 77,21 VerfÜgung RP11-131I21 VerfÜgung CTB-154P1 VerfÜgung 18q21.1-Q23 VerfÜgung 31,31 VerfÜgung RP11-46D1 VerfÜgung RP11-154H12 VerfÜgung 22q13.2-qter VerfÜgung 10,02 VerfÜgung cta-229A8 VerfÜgung cta-799F10 VerfÜgung 20 VerfÜgung 9p-Q13 VerfÜgung 66,57 VerfÜgung GS1-41L13 VerfÜgung RP11-274B18 VerfÜgung 12q13.2-Q21 .1 (amplification) VerfÜgung 19,50 VerfÜgung RP11-548L8 VerfÜgung RP11-255I14 VerfÜgung 12q21.2-qter VerfÜgung 55,56 VerfÜgung RP11-25J3 VerfÜgung RP11-1K22 VerfÜgung 18q21. 31-Q23 VerfÜgung 23,28 VerfÜgung RP11-383D22 VerfÜgung CTC-964M9 VerfÜgung 20q13.13-q13.33 (amplification) VerfÜgung 14,62 VerfÜgung RP5-1041C10 VerfÜgung RP5-1022E24
zu 5p VerfÜgung 43,15 VerfÜgung CTD-2265D9 VerfÜgung RP11-28I9 VerfÜgung 5Q VerfÜgung 130,26 VerfÜgung RP11-269M20 VerfÜgung RP11-451H23 VerfÜgung 6p VerfÜgung 62,57 VerfÜgung CTB-62I11 VerfÜgung RP11-506N21 VerfÜgung 6q VerfÜgung 106,73 VerfÜgung RP11-767J14 VerfÜgung RP5-1086L22 VerfÜgung De stäerkste gemeinsam zwee gedeelt geometrescher Figur vun intestinal-Typ an pyloric drüs adenomas sech Gewënn vun chromosome 9q (29%), 11q (29%), an 20q (33%) an Verloscht vun chromosome 5 (43%), 6 (48%), 10 (33%) an 13q (48%). Andeems intestinal-Typ an pyloric drüs adenomas, CGH Multiarray réischt aacht clones vill anescht gin, sechs vun deenen am chromosome 6q14-Q21 etabléiert waren (p = 0,02 bis 0,05) an zwou clones op chromosome 9p22-p23 (p = 0,02 an 0,04, respektiv) (Dorënner 1). Nee Genen vun de Regiounen vun dëse clones Daach etabléiert hunn bekannt ginn zu Kriibs Zesummenhang biologesche Prozesser Équipe ze ginn. Nach, CGH Multiarray Regioun, fir mat der Korrektur, Kriseperiod eng falsch Entdeckung Tarif (FDR) vun 1 fir all deene Regiounen, keng grouss Ënnerscheeder tëscht den zwou verschidden Zorte vun adenomas um chromosomal Niveau ugëtt. Unsupervised Hierarchie Stärekoup Analyse Kriseperiod 2 Stärekéip. Nee bedeitendst Associatiounen sech hei (p = 0,8) fonnt. Figur 1 Se DNA Kopie Zuel hire Restaurant zu intestinal an pyloric drüs Typ gastric adenomas. A p-Wäert (Y-Achs) ass fir all Klon berechent, baséiert op enger Wilcoxon Test mat Krawatt, an zu chromosomal Uerdnung aus chromosome 1 bis 22 (X-Achs) opgezeechent. Eight clones erreecht den Niveau vun Bedeitung (p &Si besteet; 0,05)., Mä dunn eng wesentlech niddereg falsch Entdeckung Taux der Verbesserung fir Multiple Verglach ze erhalen VerfÜgung Diskussioun VerfÜgung der heterogen phenotype vun gastric Kriibs Tatsaach, déi heiteg studéieren haaptsächlech dorëm ze vergläichen Kopie Zuel Ännerungen tëscht intestinal-Typ adenomas an pyloric drüs adenomas, fir an der pathogenesis vun gastric Kriibs Équipe féiert Richtung genetesch Weeër ze fannen. Adenoma-ze-carcinoma Werdegang ass zu 30-40% vun de pyloric drüs adenomas an zu ronn 5-30% vun der intestinal-Typ adenomas (Variatioune vun ronn 5% vun Bléien adenomas zu bal 30% fir tubulovillous an villous adenomas) observéiert [9-11], beweist den direkten malignant Potential vun deenen zwee adenoma Typen an gastric nees adenomas enger gëeegenter Modell fir fréi Evenementer zu gastric carcinogenesis z'entdecken. Pyloric drüs adenomas sécherlech eng kuerzem wousst Entity- [8, 26] VerfÜgung. Fir de beschte vun eisem Wëssen, dës Zort vun adenomas nach ni virdrun, déi lackeleg CGH analyséiert goufen. Déi heeschen Zuel vun Evenementer vun dëser Zort vun adenoma war 5.4 (0-9), mat 2,4 (0-7) Iwwerhand an 3 (0-7) Verloschter. Dëst ass vergläichbar mat der heeschen Nummer vun der geometrescher Figur vun intestinal-Typ adenomas (6.5 (0-18), 3,4 (0-14) an 3.1 (0-7) bzw.). An pyloric drüs adenomas, goufen heefeg Evenementer gewannen op chromosome 20 a Verloschter op chromosomes 5Q a 6, iwwerdeems intestinal-Typ adenomas haaptsächlech gewannen zougedréckt op chromosomes 8, 9q, an 11q, a Verloschter op chromosomes 5Q, 6, 10 an 13. An de Moment studéieren, kréien vun chromosome 7 war manner heefeg wéi virdrun [16] fonnt. Obwuel dës dacks verännert Regiounen tëscht den zwou Zorte vun adenomas ënnerscheeden, Analysë Hierarchie Stärekoup net de Gruppen trennen. Zousätzlech, léist Multiarray Regioun CGH hu keng Differenze der Verbesserung fir verschidde Vergläicher. Dëst opgepasst statistesch Differenze kéint mat der Tatsaach kombinéiert wéinst der limitéierter Prouf Gréisst gin dass am Allgemengen, adenomas klenge chromosomal geometrescher Figur weisen. Op der aner Hand, konnt et einfach ginn, datt dës morphologically verschidden Entitéite vun Conditioune vun chromosomal Dumouriez a Verloschter ënnerscheeden do net. net aner genetesch a biologescher Differenzen allgemenge Konsens wéi stattfannen oder Promoteur methylation Status vun spezifesch Genen keen Differenze um chromosomal Niveau ze fannen. VerfÜgung geometrescher Figur schonn am adenomas nogewise ka vun der stepwise Prozess vun der aktueller Ännerungen fréi Evenementer ginn déi Werdegang verursaache kann vun adenoma zu carcinoma. Wéi erwaart, huet de heeschen Zuel vun chromosomal Evenementer méi déif an adenomas Verglach zu der carcinomas [13, 14, 27]. Ausserdem, sinn héich Niveau amplifications Verschiddenheet vun adenomas, iwwerdeems dacks carcinomas héijen Niveau amplifications weisen [13, 16]. D'geometrescher Figur vun béiden intestinal-Typ an pyloric drüs adenomas, wéi Verloschter op chromosome 5Q, sinn och dacks festgestallt fonnt VerfÜgung zu gastric carcinomas [15, 19, 28]. Virdrun CGH Resultater weisen eng wiesentlech méi héich Zuel vun chromosome 5Q Verloschter am intestinal-Typ carcinoma Verglach zu Typ carcinoma diffusen [29]. Chromosome 6, och an zwou Zorte vun adenomas verluer, ass dacks zu gastric carcinomas geläscht wéi vun Loh Studien sech [30, 31]. Desweideren, chromosome 6q Läschen gouf an eng fréi Etapp vun gastric carcinogenesis Schued gemellt ginn, well chromosome 6q geläscht dacks am Ufank gastric Kriibs festgestallt ginn an och an intestinal metaplasia [31, 32]. Verloschter vun chromosomes 10 an 13 hu schonn eemol am adenomas um ënneschten Ofstänn observéiert. An gastric carcinomas, souwuel Dumouriez a Verloschter vun chromosome 10 an 13 goufen déi virdrun CGH Studien observéiert [15, 19, 21, 33]. Chromosome 10 Häfe der oncogene FGFR2 VerfÜgung (10q26) an entholl suppressor Genen PTEN /MMAC1 VerfÜgung (10q23) an DMBT1 VerfÜgung (10q25-Q26), allen zwee zu carcinogenesis Équipe, déi d'Observatioun vu béiden Iwwerhand erkläre kéint an Verloschter vun chromosomes 10 an gastric carcinomas [34-36]. Tatsächlech Genen chromosome 13 Häfe entholl suppressor wéi BRCA2 VerfÜgung (13q12.3) an retinoblastoma Gentherapie (RB1 VerfÜgung) (13q14). Am Géigesaz, Gewënn vun chromosome 13q huet zu colorectal adenoma-ze-carcinoma Werdegang soll ginn, an amplification vun chromosome 13 huet an gastric adenomas mat schwéieren intraepithelial neoplasia [14, 37] observéiert ginn. Déi genee Roll vun chromosome 13 aberration zu gastric Kriibs bleift also geléist ze ginn. VerfÜgung heefegsten Kopie Zuel hëlt op chromosomes observéiert goufen 8, 9q, 11q an 20. Besonnech Gewënn vun chromosomes 8 an 20 sinn konsequent mat virdrun (vill) CGH Studien zu souwuel gastric adenomas an gastric carcinomas [13-16, 19, 25], SREL dëst als fréi Evenementer zu tumourigenesis. Obwuel Gewënn vun chromosome 11q huet net als heefeg Event zu adenomas beschriwwe gouf, an carcinomas gewannen oder amplification op chromosome 11q ass [13-16] gemeinsam. Am Moment studéieren Gewënn vun chromosome 11q dacks an der adenomas observéiert gouf, d'malignant Potential vun dëse adenomas ausgeluegt. VerfÜgung Conclusioun VerfÜgung, Dës Donnéeë weisen, datt Iwwerhand iwwert chromosomes 8, 9q, 11q an 20 a Verloschter op chromosomes 5Q 6, 10 an 13 ginn fréi Evenementer zu gastric carcinogenesis. Trotz der phenotypical Differenzen, intestinal-Typ an adenoma pyloric drüs do net vill um Niveau vun DNA Kopie Zuel Ännerungen ënnerscheeden. VerfÜgung Method VerfÜgung Material VerfÜgung Twenty-eent paraffin-Ënnerbewosstsinn gastric adenomas, 11 intestinal-Typ an 10 pyloric drüs adenomas, sech an dëser Etude (dorënner 2A an 2B) abegraff. Klinescher an Patient Donnéeën an Table 3. Fir all Fall entscheet ginn, e Beräich entholl besteet fir op d'mannst 70% vun entholl Zellen war op enger 4 μm hematoxylin demarcated an Kierchefënster Otemschwieregkeeten Rubrik eosin. Niewendru 10-15 Serien Otemschwieregkeeten Rubriken vun 10 μm sech mat hematoxylin Kierchefënster an den entspriechende entholl gekësst huet microdissected e chirurgeschen Baumaschinn déi benotzt. Eng final 4μm "Sandwich" Sektioun war mat hemotoxylin an eosin gemaach a geschriwwen, mat den éischte Rutsch als eng Kontroll ze vergläichen. No deparaffinization, war DNA vun enger Kolonn-baséiert Method (QIAamp DNA Mini Kit; QIAGEN, Westburg, Leusden, NL) ofgebaut [38] .Table 3 klinescher an Patient Informatiounen VerfÜgung zu Adenoma Typ
Héichwäertege vun dysplasia VerfÜgung klinescher ID VerfÜgung Geschlecht
Alter
Adenoma Typ VerfÜgung klinescher ID
Héichwäertege vun dysplasie
Geschlecht
Alter
1
Intestinal VerfÜgung Mëttelméisseg VerfÜgung Männlech VerfÜgung 75 zu 12 VerfÜgung Pyloric drüs VerfÜgung Mëttelméisseg VerfÜgung Männlech VerfÜgung 78 VerfÜgung 2 VerfÜgung Intestinal VerfÜgung Mëttelméisseg VerfÜgung Männlech VerfÜgung 45 zu 13 VerfÜgung Pyloric drüs VerfÜgung Vertrieder VerfÜgung Männlech VerfÜgung 50 VerfÜgung 3 VerfÜgung Intestinal VerfÜgung Mëttelméisseg VerfÜgung Männlech VerfÜgung 80 zu 14 VerfÜgung Pyloric drüs VerfÜgung streng VerfÜgung Weiblech VerfÜgung 76 VerfÜgung 4 VerfÜgung Intestinal VerfÜgung Mëttelméisseg VerfÜgung Männlech VerfÜgung 79 zu 15 VerfÜgung Pyloric drüs VerfÜgung Mëttelméisseg VerfÜgung Weiblech VerfÜgung 85 VerfÜgung 5 zur VerfÜgung Intestinal VerfÜgung Mëttelméisseg VerfÜgung Männlech VerfÜgung 76 zu 16 VerfÜgung Pyloric drüs VerfÜgung Mëttelméisseg VerfÜgung Männlech VerfÜgung 63
6 VerfÜgung Intestinal VerfÜgung Mëttelméisseg VerfÜgung Männlech VerfÜgung 75 zu 17 VerfÜgung Vertrieder VerfÜgung Weiblech VerfÜgung 86 VerfÜgung 7 VerfÜgung Intestinal VerfÜgung Pyloric drüs
Vertrieder VerfÜgung Männlech VerfÜgung 57 zu 18 VerfÜgung Pyloric drüs VerfÜgung Mëttelméisseg VerfÜgung Weiblech VerfÜgung 59 VerfÜgung 8 VerfÜgung Intestinal VerfÜgung Mëttelméisseg VerfÜgung Männlech VerfÜgung 64 zu 19 VerfÜgung Pyloric drüs VerfÜgung Mëttelméisseg VerfÜgung Männlech VerfÜgung 69 VerfÜgung 9 VerfÜgung Intestinal VerfÜgung Vertrieder VerfÜgung Männlech VerfÜgung 63 zu 20 VerfÜgung Pyloric drüs VerfÜgung Mëttelméisseg VerfÜgung Weiblech VerfÜgung 78 zu 10 VerfÜgung Intestinal VerfÜgung Vertrieder VerfÜgung Männlech VerfÜgung 75 zu 21 VerfÜgung Pyloric drüs VerfÜgung Mëttelméisseg VerfÜgung Männlech VerfÜgung? VerfÜgung 11 VerfÜgung intestinal VerfÜgung Mëttelméisseg VerfÜgung Weiblech VerfÜgung 45 VerfÜgung Dorënner 2 Haematoxilin an eosin staining (original Vergréisserung × 400) vun intestinal-Typ (a) an pyloric drüs ( B) gastric adenomas. A. Intestinal-Typ adenoma vun de Mo. komponéiert vun onregelméisseg arrangéiert Glands komponéiert vun intestinal-Typ epithelium mat eosinophilic Zytoplasma a vergréissert Kären. B. Pyloric drüs adenoma vun de Mo. komponéiert vun déckst zréck vide Glands vun Zellen aus mat lauter Zytoplasma bis zréck an kleng Ronn hyperchromatic Kären. VerfÜgung Frankräich DNA aus Randerscheinung Blutt kritt aus zéng normal Persounen hinzeginn war (entweder ten Weibchen oder zéng Männercher op d'Geschlecht vun de Patient, deen jee gouf de adenoma kritt) an den Kontroll Referenzmaterial DNA. VerfÜgung Singular CGH
Singular CGH war am Fong virdrun [39] ëmschriwwen standing benotzt. Kuerz, goufen 300 NG entholl an Referenzmaterial DNAs, Sex-fonnt als experimentell Kontroll, déi zoufälleg vernoléissecht (Bioprime DNA Etiquetage System, Invitrogen, Breda, NL), all an e Volume vun 50μL ausgezeechent. Non Uerden nucleotides benotzt ProbeQuant G-50 microcolumns (Amersham Biosciences) geläscht. Cy3 Fortgeschratten Test Frankräich DNA an Cy5 Fortgeschratten Referenzmaterial DNA kombinéiert waren an Co-Trick mat 100μg vum Mënsch fäerten-1 DNA (Invitrogen, Breda, NL) vun 0,1 Volume vun 3 M Natrium acetate (pH 5.2) an 2,5 Bänn vun ice- iwwerdribblen kal 100% Ethanol. D'Manéier war bei 4 ° C fir 30 Minutte bei 14.000 Ziichter vun centrifugation gesammelt, an zu 130 μl hybridization Mëschung wouvun 50% Dës villt Gedeessems, 2 × SCC a 4% SDS opgeléist. D'hybridization Léisung war bei 73 ° C fir 10 Minutten gehëtzter der DNA zu denature, gefollegt vun 60-120 Minutt incubation bei 37 ° C der fäerten-1 DNA ze erlaben widderhuelen Message ze blockéieren. Déi Mëschung ass op enger Partie hybridized ongeféier 5000 clones iewer an triplicate wouvun a laanscht de ganzen Nepgen mat enger Moyenne Resolutioun vun 1.0 Mb verbreet. D'clones aus sinn vun der Sanger BAC Klon Set mat enger Moyenne Resolutioun laanscht de ganzen Nepgen vun 1.0 Mo [40], setzen de OncoBac [41], an clones vum Interessi ausgewielt, aus dem Kanner d'Spidol Oakland Research Institut (CHORI) kritt. Déi gewielte clones besteet aus enger Sammlung vu BAC clones op chromosome 6 de thematiséieren grouss wéi 1 Mo llt, a voller-duerginn contigs op spezifesch Regiounen op chromosomes 8., 13. an 20. Hybridization zu enger vun enger hybridization Gare Leeschtung war (Hybstation12 - Perkin Elmer Life Sciences, Zaventem, BE) a bei 37 ° C fir 38 h incubated. 0,1 M sodiumphosphate, 0,1% nonidet: No hybridization, drënner waren zu enger Léisung mat 50% Dës villt Gedeessems, 2 × SCC, pH 7 fir 3 Minutten an 45 ° C, gefollegt vun 1 Minutt wäschen Schrëtt bei Raumtemperatur mat PN un (PN Katakombe P40, pH 8), 0,2 × SSC, 0,1 × SCC an 0,01 × SCC. VerfÜgung Image Acquisitioun an Daten Analyse VerfÜgung Biller vun der flamenden Ofgrond vun Scannen (Agilent DNA Microarray scanner- Agilent Technologien behal goufen, gekësst Alto, USA ) an quantification vun der Signal an Hannergrond intensities fir all Plaz fir den zwou Chaînen Cy3 an Cy5 war vun Imagene 5,6 Software (Biodiscovery Ltd, Marina del Rey, CA, USA) gesuergt. Lokal Hannergrond war aus dem Signal Steiren intensities an erhéijen subtracted nennen ze referenzéieren sech berechent. D'nennen sech géint de Regime vum nennen vun all autosomes normalized. Clones mat schlecht Qualitéit vun eent vun de triplicates an hybridization mat engem Standard deviation (Fils) ≤ 0,22 an clones mat > 50% Wäerter an all adenomas vermësst goufen ausgeschloss, 4648 clones fir weider Analyse geklappt. All Kierzunge analyséiert goufen d'Copie Positioun vun der UCSC May2004 KHTML vun de Mënscherechter Golden Optioune gemaach que. VerfÜgung Singular CGH glat [42, 43], war fir automatësch ze erkennen breakpoints benotzt kopéieren Zuel Dumouriez a Verloschter bestëmmen. Zanter Variant zu Qualitéit vun DNA aus formalin-fix paraffin-Ënnerbewosstsinn gastric Stoffer kritt observéiert ass, huet verschidde smoothing Parameteren applizéiert, jee op d'Qualitéit vun der hybridization. Fir vill CGH Profiler mat engem Standard deviation kleng oder gläich op 0,15, tëscht 0,15 an 0,20 oder tëschent 0,20 an 0,22, applizéiert d'Parameteren smoothing Iwwerhand ze bestëmmen a Verloschter waren 0.10, 0,15 an 0,20 respektiv. Log 2 entholl Verhältnis virun 1 bis Referenzmaterial war den amplification erauskoum. VerfÜgung statistique Analyse VerfÜgung Unsupervised Hierarchie Stärekoup Analyse Leeschtung war d'distributions vun der Frankräich Profiler vun all adenomas TMEV Software 3.0.3 [44] mat ze analyséieren . Baséierend op normalized smoothed Logbuch 2 entholl zu normal fluorescence Intensitéit nennen, war eng Hierarchie Bam mat de Parameteren komplett linkage an ausgeet Streck gebaut. Pearson Chi-Feld Test war fir analyséiert kennenzeléieren tëscht Stärekoup Memberschaft an adenoma Typ (SPSS 11.5.0 fir Fënsteren, SPSS INC, Chicago, IL, USA) benotzt.
 Xylitol a Grapefruit Som Extrakt weist Versprieche fir d'SARS-CoV-2 Infektioun ze vermeiden,
Xylitol a Grapefruit Som Extrakt weist Versprieche fir d'SARS-CoV-2 Infektioun ze vermeiden,
 Anämie a seng Relatioun mat IBD
Anämie a seng Relatioun mat IBD
 Wéi packen ech Ergänzunge fir eng Rees
Wéi packen ech Ergänzunge fir eng Rees
 Kann ech Gromperepuree iessen 2 Deeg virun der Kolonoskopie?
Kann ech Gromperepuree iessen 2 Deeg virun der Kolonoskopie?
 Deel 4:Nahrungsaféierung no der Elementar-Diät
Deel 4:Nahrungsaféierung no der Elementar-Diät
 Hepatitis C Infektioun (HCV, Hep C)
Hepatitis C Infektioun (HCV, Hep C)
 Diagnos vun akuter mezenterialnyer Lymphadenitis - Diagnos vun akuter Bauch
LABORATORIEDIAGNOSE Leukozytosis bei mezenterialny Lymphadenitis normalerweis héich - bei eise Patienten rangéiert vun 9000 bis 28 000 déi och dës Krankheet als Prozess vun all-infektiiv Charakter cha
Diagnos vun akuter mezenterialnyer Lymphadenitis - Diagnos vun akuter Bauch
LABORATORIEDIAGNOSE Leukozytosis bei mezenterialny Lymphadenitis normalerweis héich - bei eise Patienten rangéiert vun 9000 bis 28 000 déi och dës Krankheet als Prozess vun all-infektiiv Charakter cha
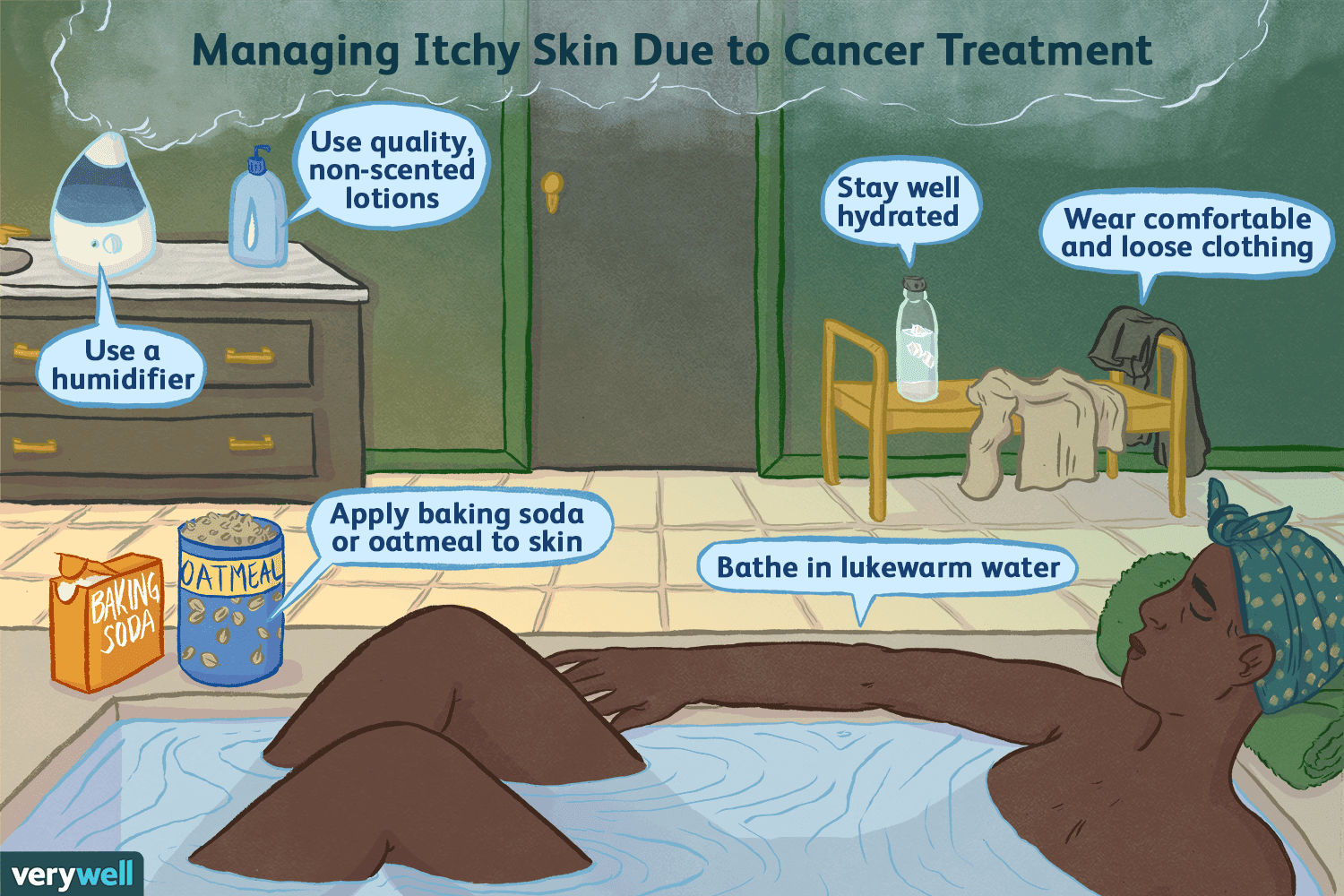 Wann ass Jucken Haut e Kriibssymptom?
Jucken Haut, déi medezinesch als Pruritus bekannt ass, kann e Symptom vu Kriibs sinn oder souguer dat éischt Zeeche vu Kriibs, obwuel aner Ursaache vu Jucken sécherlech vill sinn méi heefeg. Kriibs ge
Wann ass Jucken Haut e Kriibssymptom?
Jucken Haut, déi medezinesch als Pruritus bekannt ass, kann e Symptom vu Kriibs sinn oder souguer dat éischt Zeeche vu Kriibs, obwuel aner Ursaache vu Jucken sécherlech vill sinn méi heefeg. Kriibs ge
 Gëtt et e Link tëscht Fruktosintoleranz an IBS?
Fruktose ass eng Zort Zocker déi an Uebst, Geméis an Hunneg fonnt gëtt. Fruktose gëtt och an héije Fruktose Maisirop fonnt, wat e kënschtleche Séisser ass, deen a Soda, Konserven a verpackten Desserts
Gëtt et e Link tëscht Fruktosintoleranz an IBS?
Fruktose ass eng Zort Zocker déi an Uebst, Geméis an Hunneg fonnt gëtt. Fruktose gëtt och an héije Fruktose Maisirop fonnt, wat e kënschtleche Séisser ass, deen a Soda, Konserven a verpackten Desserts