De ontwikkeling van bepaalde soorten kanker - maagkanker en baarmoederhalskanker, bijvoorbeeld - gevestigde microbiële banden hebben. Onze technologieën voor het bestuderen van het menselijk microbioom zijn de afgelopen twee decennia snel vooruitgegaan en blijven zich dagelijks ontwikkelen. Als resultaat, we hebben nu veel nieuwe technieken om gegevens te genereren over de samenstelling en activiteiten van het menselijk microbioom, en veel kankeronderzoekers werken eraan om deze informatie te gebruiken om nieuwe microbiële verbanden te begrijpen.
Echter, omdat de analytische methoden zo nieuw zijn, de software die nodig is om deze data om te zetten in nieuwe kennis ontbreekt op dit moment. Met deze financiering we zullen die softwareleemte opvullen met de ontwikkeling van nieuwe open source software om het menselijk microbioom te relateren aan kanker. We verwachten dat dit ons uiteindelijk in staat zal stellen de ontwikkeling van kanker beter te begrijpen, om kanker eerder op te sporen en de behandeling en het herstel van kanker te verbeteren."
Greg Caporaso, Regisseur, Centrum voor Toegepaste Microbioomwetenschap
Het door NCI gefinancierde project stelt Caporaso en zijn team in staat om QIIME 2 te verbeteren, het bioinformatica-softwareplatform dat ze eind 2016 voor het eerst uitbrachten, om de toegang tot bio-informaticamethoden en -gegevens van het kankermicrobioom te verbeteren. Caporaso's team bij NAU bestaat uit afgestudeerde en niet-gegradueerde studenten en fulltime software-ingenieurs.
Matthew Dillon en Evan Bolyen, twee onderzoekssoftware-ingenieurs in het laboratorium van Caporaso en co-eerste auteurs van het QIIME 2-document, zal centraal worden betrokken bij alle ontwerp- en ontwikkelingsaspecten van dit project. Het team is van plan om QIIME 2 te ontwikkelen tot een microbioom multi-omics bio-informaticaplatform, ondersteunende analyse en integratie van genomische, metagenomisch, metabolomics en andere "omics"-gegevens, gedreven door de behoeften van de kankeronderzoeksgemeenschap.
"Veel belangrijke kankermicrobioomprojecten hebben vooruitgang geboekt door verschillende gegevenstypen te integreren, toch blijven er aanzienlijke technische hindernissen bestaan om microbioom multi-omics bio-informatica toegankelijk te maken voor alle onderzoekers wiens projecten baat zouden hebben bij deze methoden, ' zei Caporaso.
Met een achtergrond in software engineering, Caporaso's vorige project, QIIME 1, is 12 jaar geleden gestart in samenwerking met zijn postdoctoraal adviseur Rob Knight, nu de directeur van het Center for Microbiome Innovation aan de Universiteit van Californië, San Diego (UCSD). QIIME 1 is ontworpen om hun eigen studies naar microbiomen - zoals die in mensen of in de bodem worden gevonden - te vergemakkelijken, maar ook om deze methoden toegankelijk te maken voor alle microbioomonderzoekers.
Caporaso trad in 2011 toe tot de faculteit NAU waar hij zijn werk aan QIIME 1 voortzette. Door zijn werk met het Partnership for Native American Cancer Prevention, en vervolgens tijdens een sabbatical bij het NCI, Caporaso realiseerde zich het potentiële belang van het menselijke microbioom voor kanker. Zijn primaire artikelen over QIIME 1 en 2 zijn nu bijna 25 geciteerd, 000 keer in de primaire onderzoeksliteratuur, waardoor hij een van de meest geciteerde onderzoekers van de NAU is, volgens Google Scholar, en Caporaso merkt op dat bijna 20 procent van die citaten afkomstig zijn uit studies over kanker. Dit bracht hem ertoe zijn inspanningen te richten op een betere ondersteuning van de kankeronderzoeksgemeenschap met QIIME, en uiteindelijk tot deze vijfjarige onderscheiding van het NCI.
"Dit is opwindend voor kankeronderzoekers omdat het een nieuw type onderzoek in microbioomonderzoek mogelijk zal maken, " zei hij. "QIIME is typisch gebruikt om een taxonomisch begrip van het microbioom te genereren - welke microben in deze omgeving aanwezig zijn, en hoe gemeenschappen van microbiomen zich tot elkaar verhouden op basis van hun taxonomische samenstelling. Nieuwe technologieën beginnen te worden toegepast om ons te helpen rekening te houden met andere factoren, zoals met welke biologische activiteiten de microben bezig zijn, en de stofwisselingsproducten van die activiteiten. Door deze gegevens te integreren, samen met gegevens over de gastheer, zoals hun genoom, zal ons zeker leiden naar nieuwe mechanistische inzichten over de rol van het microbioom bij kanker."
QIIME 2 ondersteunt de analyse van nieuwe soorten gegevens, zoals metagenomics en metabolomics, om vragen over de activiteit van microben te beantwoorden. Dit omvat informatie over de functionele genen die worden gecodeerd in microbiële genomen en de metabolieten die aanwezig zijn in de omgeving - kleine moleculen zoals cafeïne of ethanol en producten die door microben worden geproduceerd - en hoe deze de gastheer kunnen beïnvloeden.
"Met microbioomprofilering, we krijgen een idee van de biologie; met metabolietprofilering, we krijgen een beeld van de chemie. Dat helpt ons de grotere, meer holistische kijk op wat er gaande is in deze oneindig complexe omgeving van het darmmicrobioom waar je biljoenen cellen hebt die met elkaar en hun omgeving in wisselwerking staan, alle creëren en consumeren van metabolieten, die hun gedrag en het gedrag van onze cellen beïnvloeden. We zullen niet alleen kunnen weten wie er is in termen van micro-organismen, maar wat ze doen, waar ze wonen en hoe ze met elkaar omgaan."
Net als bij QIIME 1, QIIME 2 is een open-source softwareplatform, gratis en voor iedereen beschikbaar. QIIME 2 is ontworpen om geautomatiseerde methoden voor tracking en rapportage uit te breiden om de reproduceerbaarheid van onderzoek te verbeteren, en met deze financiering zal het team nieuwe tools creëren om te helpen bij het archiveren van gegevens op de lange termijn. Updates voor QIIME 2 worden driemaandelijks uitgebracht door het team van Caporaso, en ze zijn al begonnen te werken aan enkele van de doelstellingen van deze subsidie.
"Dit is een ongelooflijk opwindend project voor de onderzoeksgemeenschap van kankermicrobioom, " zei Melissa Herbst-Kralovetz, universitair hoofddocent aan het University of Arizona Cancer Center en directeur van het Women's Health Research Program aan het UA College of Medicine-Phoenix. "Mijn lab onderzoekt de rol van microbiota bij gynaecologische kanker, seksueel overdraagbare aandoeningen en de gezondheid van vrouwen. Momenteel, we maken gebruik van 3D in vitro menselijke modellen om de rol van microbiota bij de ontwikkeling en progressie van kanker beter te begrijpen, die afhankelijk is van de integratie van verschillende gegevenstypen uit klinische monsters en onze laboratoriumgebaseerde 3D-modellen. De nieuwe functionaliteit die voor QIIME 2 wordt ontwikkeld, zal ons helpen de nauwkeurigheid van deze modellen te beoordelen, en uiteindelijk de informatie die we uit deze 3D-modellen halen, terug te vertalen naar de kliniek om kanker te bestrijden."
"Als we in staat zijn om de gastheerbiologie met elkaar te verbinden, de microbiologie en de chemie, dat is wanneer we echt in staat zullen zijn om enkele ontbrekende schakels te achterhalen tussen het microbioom en de ontwikkeling van kanker of de behandeling van kanker, ' zei Caporaso.
 Hoe fecale impactie kan worden behandeld
Hoe fecale impactie kan worden behandeld
 Bewustwording van primaire biliaire cholangitis in september
Bewustwording van primaire biliaire cholangitis in september
 Hoe lang leeft u met NAFLD? Overlevingspercentages
Hoe lang leeft u met NAFLD? Overlevingspercentages
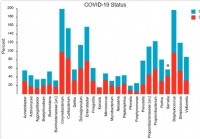 Samenstelling en structuur van het nasofaryngeale microbioom houden verband met de ernst van de ziekte van COVID-19
Samenstelling en structuur van het nasofaryngeale microbioom houden verband met de ernst van de ziekte van COVID-19
 PENTAX Medical haalt $ 125 op,
PENTAX Medical haalt $ 125 op,
 Kun je leven met stadium I-nierziekte?
Kun je leven met stadium I-nierziekte?
 Zika kan via tranen worden doorgegeven, Sweat:Report
Laatste nieuws over infectieziekten In de oudheid hadden zelfs de rijken parasieten CDC waarschuwt voor toename van hondsdolheid in verband met vleermuizen E. Coli-uitbraak in 6 staten door verpakte
Zika kan via tranen worden doorgegeven, Sweat:Report
Laatste nieuws over infectieziekten In de oudheid hadden zelfs de rijken parasieten CDC waarschuwt voor toename van hondsdolheid in verband met vleermuizen E. Coli-uitbraak in 6 staten door verpakte
 Wat kun je het beste eten als je een maagzweer hebt?
Wat is een maagzweer? Maagzweren zijn open zweren die op het maagslijmvlies ontstaan wanneer zuren uit verteerd voedsel de maagwand beschadigen. Voedingsmiddelen, fruit en groenten die rijk zijn
Wat kun je het beste eten als je een maagzweer hebt?
Wat is een maagzweer? Maagzweren zijn open zweren die op het maagslijmvlies ontstaan wanneer zuren uit verteerd voedsel de maagwand beschadigen. Voedingsmiddelen, fruit en groenten die rijk zijn
 Hoe kan ik mijn nieren thuis controleren?
Albumin-thuistestkits en voor smartphones geschikte apparaten voor urineanalyse voor thuis zijn beschikbaar om uw nierfunctie thuis te controleren. Als een persoon lijdt aan chronische nierziekte o
Hoe kan ik mijn nieren thuis controleren?
Albumin-thuistestkits en voor smartphones geschikte apparaten voor urineanalyse voor thuis zijn beschikbaar om uw nierfunctie thuis te controleren. Als een persoon lijdt aan chronische nierziekte o