Gli studi hanno dimostrato che un modo per promuovere la crescita di tali microrganismi benefici e modulare la loro composizione per un sano equilibrio consiste nell'aggiungere determinate forme di fibra, come l'inulina, alla nostra dieta. Però, tra tutte le decine di trilioni di microrganismi nel microbiota intestinale, è stato difficile determinare quali e come i microrganismi rispondono alla fibra alimentare. Questo perché le tecniche attuali si basano sulla disponibilità di genomi di riferimento nei database di sequenze di DNA per una classificazione tassonomica precisa e assegnazioni funzionali accurate di organismi specifici, ma in realtà, si stima che la metà delle specie intestinali umane non abbia un genoma di riferimento. Inoltre, le tecniche esistenti richiedono ore o addirittura giorni per completare l'attività.
Per affrontare questo problema, Gli scienziati della Waseda University hanno ideato una nuova tecnica chiamata genomi amplificati a cellula singola nella piattaforma di sequenziamento delle perline di gel (SAG-gel), che può fornire più bozze di genomi del microbiota intestinale contemporaneamente e identificare i batteri che rispondono alla fibra alimentare a livello di specie senza la necessità di genomi di riferimento esistenti. Cosa c'è di più, il vantaggio di questa tecnica è che ci vogliono solo 10 minuti per ottenere bozze di genomi dai dati grezzi del sequenziamento dell'intero genoma poiché ogni dato è puramente derivato da singoli microbi. Questo accelera notevolmente il tempo necessario per il processo.
Il nostro nuovo, la tecnica di sequenziamento del genoma unicellulare può ottenere ciascun genoma batterico separatamente e caratterizzare batteri non coltivati con funzioni specifiche nel microbiota, e questo può aiutarci a stimare i lignaggi metabolici coinvolti nella fermentazione batterica delle fibre e gli esiti metabolici nell'intestino in base alle fibre ingerite. Introduce un'analisi funzionale avanzata ed efficiente dei batteri non coltivati nell'intestino".
Masahito Hosokawa, assistente professore presso la Facoltà di Scienze e Ingegneria della Waseda University e corrispondente autore di questo studio
Quello che gli scienziati hanno fatto è stato nutrire i topi con una dieta a base di inulina per due settimane e hanno usato la tecnica per catturare casualmente le singole cellule batteriche trovate nei campioni fecali dei topi in minuscole perle di gel. Le cellule batteriche sono state quindi processate individualmente nelle sfere di gel che galleggiano in una provetta, e più di 300 genomi amplificati a cellula singola (SAG), o genomi da un organismo unicellulare come batteri, sono stati ottenuti mediante sequenziamento massicciamente parallelo. Poiché ogni SAG è composto in media da decine di migliaia di letture, consente un sequenziamento dell'intero genoma estremamente conveniente delle cellule bersaglio. Dopo il controllo di qualità e la classificazione dei SAG, gli scienziati hanno determinato quali batteri erano responsabili della decomposizione dell'inulina e dell'estrazione di energia da essa.
"Secondo i nostri risultati, la dieta ricca di inulina ha aumentato le attività del batterioidi specie all'interno dell'intestino del topo, "Hosokawa spiega. "Inoltre, dalla bozza di genomi di nuova scoperta batterioidi specie, abbiamo scoperto lo specifico cluster di geni per la scomposizione dell'inulina e la specifica via metabolica per la produzione di specifici acidi grassi a catena corta, un metabolita prodotto dal microbiota intestinale. Risultati come questi aiuteranno gli scienziati in futuro a prevedere la fermentazione metabolica delle fibre alimentari in base alla presenza e all'abilità dei rispondenti specifici".
Questa tecnica potrebbe essere applicata ai batteri che vivono ovunque, se è all'interno dell'intestino umano, nell'oceano, o anche nel terreno. Sebbene sia necessario migliorarne l'accuratezza poiché la lettura della sequenza del DNA per alcune regioni geniche è ritenuta difficile, Hosokawa spera che questa tecnica venga applicata in medicina e nell'industria e venga sfruttata per migliorare la salute umana e animale.
I loro risultati sono stati pubblicati in microbioma il 23 gennaio 2020.
 I pazienti in immunoterapia dovrebbero consumare più fibre,
I pazienti in immunoterapia dovrebbero consumare più fibre,
 PENTAX Medical raccoglie 125 dollari,
PENTAX Medical raccoglie 125 dollari,
 Una variante genetica comune spiega perché l'immunoterapia spesso fallisce nella malattia di Crohn
Una variante genetica comune spiega perché l'immunoterapia spesso fallisce nella malattia di Crohn
 I ricercatori manipolano le specie batteriche nell'intestino usando la dieta
I ricercatori manipolano le specie batteriche nell'intestino usando la dieta
 Quello che mangi può cambiare il modo in cui gli antibiotici influenzano il tuo intestino
Quello che mangi può cambiare il modo in cui gli antibiotici influenzano il tuo intestino
 Microbiota spermatico rivelato con il sequenziamento dell'RNA
Microbiota spermatico rivelato con il sequenziamento dell'RNA
 Il rotavirus gioca un ruolo nello sviluppo del diabete di tipo 1
Un nuovo articolo pubblicato sulla rivista PLOS Pathogens il 10 ottobre, 2019, sostiene che il comune rotavirus potrebbe essere responsabile di alcuni casi di diabete di tipo 1, una forma di diabete c
Il rotavirus gioca un ruolo nello sviluppo del diabete di tipo 1
Un nuovo articolo pubblicato sulla rivista PLOS Pathogens il 10 ottobre, 2019, sostiene che il comune rotavirus potrebbe essere responsabile di alcuni casi di diabete di tipo 1, una forma di diabete c
 L'analisi del sangue per il DNA microbico potrebbe mettere in guardia dal cancro
Un nuovo studio pubblicato sulla rivista Natura l11 marzo 2020, potrebbe trasformare le opinioni attuali sulla causa e sulla diagnosi del cancro. I ricercatori hanno escogitato una nuova tecnica per
L'analisi del sangue per il DNA microbico potrebbe mettere in guardia dal cancro
Un nuovo studio pubblicato sulla rivista Natura l11 marzo 2020, potrebbe trasformare le opinioni attuali sulla causa e sulla diagnosi del cancro. I ricercatori hanno escogitato una nuova tecnica per
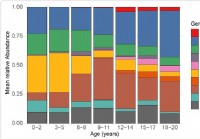 Cambiamento del microbioma respiratorio superiore nei bambini correlato alla suscettibilità alla SARS-CoV-2
I ricercatori hanno testato il microbioma nasofaringeo dei bambini fino a 21 anni e hanno scoperto che il microbioma cambia con letà. batteri specifici, la cui abbondanza cambia anche con letà, sono a
Cambiamento del microbioma respiratorio superiore nei bambini correlato alla suscettibilità alla SARS-CoV-2
I ricercatori hanno testato il microbioma nasofaringeo dei bambini fino a 21 anni e hanno scoperto che il microbioma cambia con letà. batteri specifici, la cui abbondanza cambia anche con letà, sono a