Rivista in cui è stato pubblicato lo studio: Scoperta del cancro , una rivista dell'Associazione americana per la ricerca sul cancro
Autori:co-autori senior Andrea Sottoriva, dottorato di ricerca, Master, Chris Rokos Fellow in Evolution and Cancer e team leader presso l'Institute of Cancer Research, Londra e Nicola Valeri, dottore, dottorato di ricerca, leader del team di biologia e genomica del cancro gastrointestinale presso l'Istituto di ricerca sul cancro, Londra, e consulente medico oncologo presso The Royal Marsden NHS Foundation Trust
Contesto:"Combinando frequenti campionamenti longitudinali di DNA privo di cellule con modelli matematici dell'evoluzione del tumore, siamo stati in grado di fare previsioni statistiche sui pazienti a rischio di progressione, " ha detto Sottoriva. "Potremmo anche determinare quando un cancro stava per tornare, paziente per paziente. Questa è la prima volta che una previsione quantitativa di questo tipo è stata utilizzata con successo nel cancro".
Sebbene i medici utilizzino spesso le biopsie tumorali per la genotipizzazione del cancro, molti tumori hanno eterogeneità intratumorale che può determinare resistenza al trattamento; perciò, sono necessarie più biopsie nel tempo e nello spazio per capire meglio come i tumori si evolvono per resistere alla terapia, ha spiegato Valerio.
Le biopsie liquide non sono invasive, consentendo la raccolta del DNA tumorale circolante in molti momenti senza rischi aggiuntivi per il paziente. Per di più, l'analisi del DNA tumorale circolante può catturare l'eterogeneità intratumorale meglio di un piccolo pezzo di tumore, disse Valerio.
Sebbene molte ricerche si siano concentrate sull'utilità clinica del DNA privo di cellule (cfDNA) per il monitoraggio delle malattie, l'uso di biopsie liquide come strumento predittivo nella stima del tempo alla progressione della malattia non è stato studiato a fondo, Sottoriva ha notato.
Come è stato condotto lo studio e risultati:i ricercatori hanno analizzato i risultati dello studio PROSPECT-C che ha valutato i biomarcatori sia della risposta che della resistenza alle terapie anti-EGFR in pazienti con carcinoma colorettale metastatico RAS wild-type. Le biopsie tumorali sono state prelevate da pazienti in momenti predefiniti di pre-trattamento (baseline) e post-trattamento (progressione della malattia), e a risposta parziale in alcuni. Inoltre, i pazienti hanno fornito campioni di plasma ogni quattro settimane fino alla progressione della malattia.
Anche se la genotipizzazione standard del tumore ha classificato i pazienti come affetti da cancro del colon-retto metastatico con RAS wild-type, l'analisi del cfDNA al basale ha rivelato che molti di questi tumori avevano aberrazioni nelle proteine RAS, il che potrebbe spiegare perché erano resistenti al cetuximab, un inibitore dell'EGFR, ha notato Valeri. Per di più, il sequenziamento ultra-profondo dei nuclei della biopsia tumorale al basale ha rivelato mutazioni di RAS, evidenziando ulteriormente i limiti dei metodi standard per la genotipizzazione del tumore, Ha aggiunto.
Valeri e colleghi hanno generato modelli matematici che hanno utilizzato cfDNA e livelli di antigene carcinoembrionario (CEA) dal plasma dei singoli pazienti per prevedere il tempo alla progressione. I risultati sono stati convalidati utilizzando misurazioni RECIST da dati di imaging radiologico.
Il modello matematico che utilizza misurazioni CEA è stato applicato a sei pazienti per prevedere il tempo alla progressione clinica. Di queste previsioni, tre erano entro il 10% del tempo di progressione misurato da RECIST.
In particolare, previsioni generate con profili cfDNA ad alta sensibilità hanno permesso di prevedere il tempo di progressione con diverse settimane di anticipo, rispetto ai modelli che utilizzano misurazioni CEA.
Con le informazioni raccolte dal cfDNA, i ricercatori potrebbero generare più modelli basati sulla crescita prevista di singoli subcloni guidati da diverse mutazioni. L'accuratezza dei modelli che utilizzano cfDNA si basa sull'identificazione dei subcloni dominanti in pazienti con meccanismi di resistenza policlonale, ha detto Valerio.
Commenti dell'autore:"Integrazione di nuove tecnologie di monitoraggio come cfDNA, in combinazione con modelli matematici di previsione del tumore, può offrire l'opportunità di agire in anticipo, interrompere la terapia, o cambiare trattamento per stare un passo avanti rispetto alla malattia, " ha detto Valeri. "Il nostro metodo consente una previsione più accurata e un migliore monitoraggio della risposta alla terapia".
Limiti dello studio:i limiti dello studio includono una piccola dimensione del campione, oltre a concentrarsi sulle aberrazioni del percorso RAS nei modelli matematici, poiché altri determinanti genetici e non genetici probabilmente causeranno resistenza e progressione della malattia, nota Sottoriva. Questo modello dovrà essere validato prospetticamente in studi futuri, Ha aggiunto.
 La funzione epatica può essere importante nel rischio di malattia di Alzheimer
La funzione epatica può essere importante nel rischio di malattia di Alzheimer
 Gli scienziati sviluppano un approccio per vaccinare contro l'infiammazione intestinale
Gli scienziati sviluppano un approccio per vaccinare contro l'infiammazione intestinale
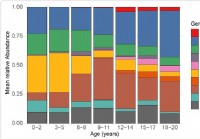 Cambiamento del microbioma respiratorio superiore nei bambini correlato alla suscettibilità alla SARS-CoV-2
Cambiamento del microbioma respiratorio superiore nei bambini correlato alla suscettibilità alla SARS-CoV-2
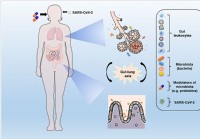 La modulazione del microbiota e il ripristino dell'eubiosi potrebbero aiutare a frenare le complicanze del COVID-19
La modulazione del microbiota e il ripristino dell'eubiosi potrebbero aiutare a frenare le complicanze del COVID-19
 Gravi complicanze del COVID-19 legate alla rottura della barriera intestinale
Gravi complicanze del COVID-19 legate alla rottura della barriera intestinale
 Il microbioma intestinale cambia con la cottura di alimenti vegetali,
Il microbioma intestinale cambia con la cottura di alimenti vegetali,
 Il trapianto di liquido vaginale potrebbe aiutare a curare la vaginosi batterica ricorrente
La vaginosi batterica è un problema comune e fastidioso per molte donne. Ora i ricercatori hanno scoperto che il trapianto di fluidi vaginali di una donna sana in donne con fastidiose vaginosi batteri
Il trapianto di liquido vaginale potrebbe aiutare a curare la vaginosi batterica ricorrente
La vaginosi batterica è un problema comune e fastidioso per molte donne. Ora i ricercatori hanno scoperto che il trapianto di fluidi vaginali di una donna sana in donne con fastidiose vaginosi batteri
 Microbiota intestinale e IBD:la connessione forse nella dieta dice uno studio
Un nuovo studio ha mostrato prove conclusive che la malattia infiammatoria intestinale o IBD è intimamente associata allambiente microbico intestinale che può essere alterato con una dieta prescritta.
Microbiota intestinale e IBD:la connessione forse nella dieta dice uno studio
Un nuovo studio ha mostrato prove conclusive che la malattia infiammatoria intestinale o IBD è intimamente associata allambiente microbico intestinale che può essere alterato con una dieta prescritta.
 Le diete a base vegetale migliorano la salute del cuore attraverso il microbioma intestinale
Un nuovo studio pubblicato nel febbraio 2020 su Giornale dellAmerican College of Cardiology riporta che una ridotta assunzione di alimenti di origine animale e una dieta a base di piante possono com
Le diete a base vegetale migliorano la salute del cuore attraverso il microbioma intestinale
Un nuovo studio pubblicato nel febbraio 2020 su Giornale dellAmerican College of Cardiology riporta che una ridotta assunzione di alimenti di origine animale e una dieta a base di piante possono com