 Alpha Tauri 3D -grafik | Shutterstock
Alpha Tauri 3D -grafik | Shutterstock
Undersøgelsen antydede også, at en lignende tilgang kunne bruges til at bestemme dyre- og svampekomponenter i vores kost.
DNA -stregkodning er en teknik, der bruger korte DNA -sekvenser til hurtigt at identificere en organismes art. Verden over, forskere har samarbejdet for at få alle levende ting DNA stregkodet.
DNA -metabarkodning er en særlig type stregkodning, der kan anvendes på prøver, der indeholder mere end én organisme. Den bruger de samme referencedatabaser som stregkodning gør, men tillader taxa fra blandede prøver at blive identificeret ved hjælp af sekvenseringsmetoder med høj kapacitet. Teknikkens potentiale er ekspansivt, med det, at artssammensætning kan bestemmes i stort set enhver prøve.
Den aktuelle undersøgelse, som for nylig blev offentliggjort i tidsskriftet mSystemer , viste, at planteplante -DNA i humane afføringsprøver kan amplificeres og sekvenseres ved hjælp af teknikker, der almindeligvis anvendes i dyrelivsstudier.
"DNA -sekventering har givet os en stor mængde nye data om ting som mikrobiologi i tarmen og personlig genetik. Denne undersøgelse tyder på, at den samme kraftfulde teknologi også kunne begynde at fortælle os om, hvad vi spiser, hvilket ofte er svært at måle.
Seniorforfatter Lawrence David, fra Duke Center for genomisk og beregningsbiologi
Mange teknikker bruges allerede til at vurdere kostindtag, men de fleste af dem afhænger af folks selvrapportering af, hvad de har spist. Denne dataindsamlingsmetode er genstand for fejl i hukommelsen og bias i rapportering samt afhængig af en persons kognitive evne til at besvare undersøgelser.
Ved hjælp af DNA -metabarkodning, forskere kan forstærke DNA til stede i en afføringsprøve og bruge en referencedatabase til at kortlægge sekvenserne til fødevarer.
"Jeg tænker på DNA -metabarkodning meget gerne en stregkode i et supermarked. Vi kan tænke på en bestemt DNA -sekvens som en unik identifikator for en bestemt fødevareart, " siger medforfatter Brianna Petrone, også fra Duke University.
David og co-first forfatter Aspen Reese fra Harvard University, blev bedt om at starte undersøgelsen, efter at de mødte økologer Rob Pringle fra Princeton University og Tyler Kartzinel fra Brown University. Disse forskere brugte DNA -metabarkodning til at analysere komplekse madbaner blandt planteædere i den afrikanske savanne.
David og Reese spekulerede på, om metoden også kunne bruges til at studere mennesker.
Der er en voksende mængde arbejde på mikrobiomområdet, der indikerer, at specifikke fødevarer sandsynligvis vil ændre eller forme niveauer af specifikke bakterier i tarmen, men vi har ofte ikke ledsagende kostdata til mikrobiomundersøgelserne. "
Lawrence David
Til undersøgelsen, forskerne tog DNA ud af kølerummet, der var blevet ekstraheret fra afføring, der blev brugt i en af deres tidligere undersøgelser.
"Vi lavede tilfældigt en undersøgelse for et par år siden, hvor vi forberedte mad til deltagere i en mikrobiom diætintervention, og vi vidste nøjagtigt, hvad de spiste i en given uge, hvor deres afføring blev indsamlet, " siger David.
De sekventerede en stregkode -region fra chloroplast -DNA i prøver taget fra 11 personer, der havde fulgt både frit udvalgte og kontrollerede kostvaner.
De amplificerede med succes plante -DNA for cirka halvdelen af prøverne, som steg til 70%, når prøver fra mennesker, der indtog en kontrolleret, der blev brugt planterig kost.
Det meste af det DNA, der blev sekventeret, matchede menneskelige fødeplanter, der i vid udstrækning blev konsumeret af mennesker, herunder korn, grøntsager og frugt.
"Samlet set, der var god bred overensstemmelse mellem de fødevarer, der var anført i de dagbøger, der blev opbevaret af undersøgelsesdeltagerne, og dem, som vi sekventerede fra afføring, " siger David. "Hvis der blev skrevet en mad i kostrekorden, omkring 80% af tiden, vi fandt det også ved denne metabarkodende tilgang. "
Den relativt høje PCR -svigt og manglende evne til at skelne visse planter på sekvensniveau giver plads til forbedringer i fremtiden.
For eksempel, kål, broccoli, kålrabi og rosenkål er alle plantesorter inden for samme art, og forskerne kunne ikke skelne mellem dem ved hjælp af kloroplast -DNA -sekvenserne.
Den eneste mad, der blev registreret i kosten, der ikke var påviselig, var kaffe, muligvis på grund af dets DNA, der er blevet forringet eller fortyndet ved ristning og brygning.
David forventer, at DNA -metabarkodning vil blive brugt i fremtidige undersøgelser, samt til kostanalyse i tidligere undersøgelser.
Ligner denne undersøgelse, Jeg kunne forestille mig, at dette blev brugt på arkiveret DNA for at se, om der er underliggende diætforskelle, der kan forklare nogle af de mikrobiom -mønstre, der kan være blevet observeret i en undersøgelse. Fremadrettet, vi kan også forestille os, at dette bruges i nye mikrobiomundersøgelser til at identificere forholdet mellem bestemte fødevarer og tarmbakterier, såvel som i bredere undersøgelser af ernæring som et supplement til traditionelle kostvurderingsteknikker. "
Lawrence David
 5 grunde til IKKE at skære ned på kulhydrater for tarmens sundhed
I denne uge (27. november) på #AskADietitian taler jeg om mine top 5 grunde til HVORFOR det måske ikke er i din bedste interesse at hoppe på denne ketogene, low carb dille, og hvad vi skal overveje om
5 grunde til IKKE at skære ned på kulhydrater for tarmens sundhed
I denne uge (27. november) på #AskADietitian taler jeg om mine top 5 grunde til HVORFOR det måske ikke er i din bedste interesse at hoppe på denne ketogene, low carb dille, og hvad vi skal overveje om
 Sød og krydret SCD Pumpkin Chili
Hvis du ikke har skåret alle dine græskar ud i jack-o-lanterner, er det tid til at lave mad med dem. Og hvad er bedre at gøre end at lave en stor gryde lækkert, som du kan spise i dagevis (eller nemt
Sød og krydret SCD Pumpkin Chili
Hvis du ikke har skåret alle dine græskar ud i jack-o-lanterner, er det tid til at lave mad med dem. Og hvad er bedre at gøre end at lave en stor gryde lækkert, som du kan spise i dagevis (eller nemt
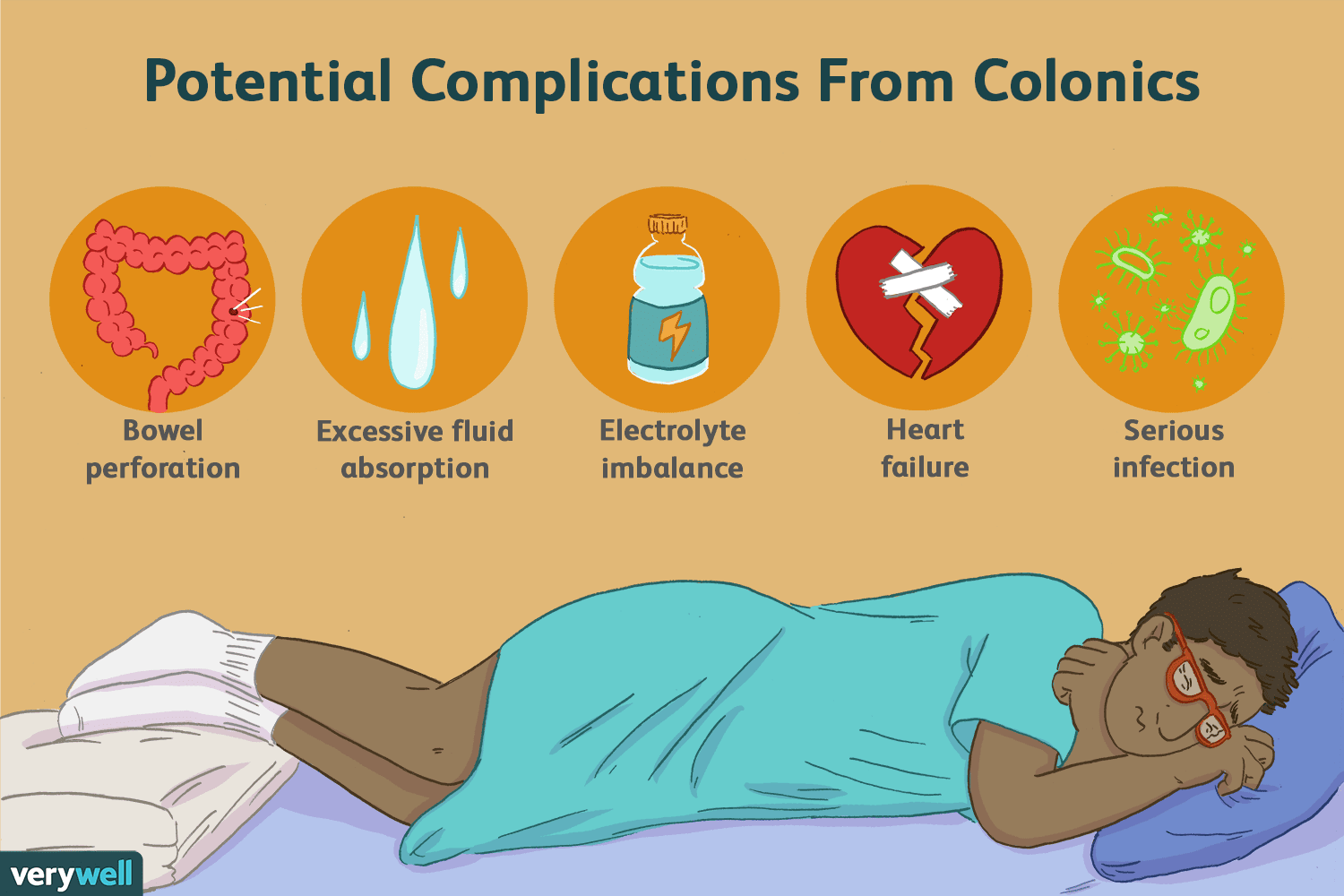 Kolonanvendelser, historie og komplikationer
En tyktarm er infusion af vand eller andre væsker i endetarmen af en tyktarmsterapeut for at rense og skylle tyktarmen ud. Det kaldes også colon hydroterapi eller colon irrigation. Colonics og lavem
Kolonanvendelser, historie og komplikationer
En tyktarm er infusion af vand eller andre væsker i endetarmen af en tyktarmsterapeut for at rense og skylle tyktarmen ud. Det kaldes også colon hydroterapi eller colon irrigation. Colonics og lavem