Abstrakt
chitinaser hydrolyserer de β-1-4 glycosidbindinger af chitin, en vigtig strukturel komponent af svampe, krebsdyr og insekter. Selvom pattedyr ikke producerer kitin eller dets syntase, de udtrykker to aktive kitinaser, chitotriosidase (Chit1) og sure pattedyr chitinase (AMCase). Disse mammale chitinaser har tiltrukket sig betydelig opmærksomhed på grund af deres øgede ekspression i individer med en række patologiske tilstande, herunder Gauchers sygdom, Alzheimers sygdom og astma. bidrag af disse enzymer til patofysiologien af disse sygdomme er dog stadig, der skal fastlægges. Den kvantificering af de Chit1 og AMCase mRNA niveauer og sammenligningen af disse niveauer med niveauerne af kendte referencepunkter gener kan generere nyttige og biomedicinsk relevante oplysninger. I begyndelsen, vi etableret en kvantitativ realtids-PCR system, der bruger standard DNA fremstillet ved ligering af cDNA-fragmenter af målgenerne. Dette system gjorde det muligt at kvantificere og sammenligne ekspressionsniveauerne af de chitinaser og reference- generne i samme målestok. Vi fandt, at AMCase mRNA syntetiseres på ekstraordinært høje niveauer i musen maven. Niveauet for denne mRNA i musen maven var 7- til 10 gange højere end niveauerne af housekeeping-gener og var sammenlignelig med niveauet af mRNA for pepsinogen C (progastricsin), en vigtig komponent i maveslimhinden. , AMCase mRNA er således en stor udskrift i mus maven, hvilket tyder på, at AMCase fungerer som et fordøjelsesfremmende enzym, der nedbryder polymer kitin og som del af værtens forsvar mod chitin-holdige patogener i maveindhold. Vores metode er anvendelig til kvantificering af mRNA for flere gener på tværs af flere prøver vha den samme skala
Henvisning:. Ohno M, Tsuda K, Sakaguchi M, Sugahara Y, Oyama F (2012) chitinase- mRNA-niveauer ved kvantitative PCR Brug af Single Standard DNA: Sur Mammalian chitinase- er en stor udskrift i Mouse mave. PLoS One 7 (11): e50381. doi: 10,1371 /journal.pone.0050381
Redaktør: Dominik Hartl, University of Tübingen, Tyskland
Modtaget: August 6, 2012; Accepteret: 19 oktober 2012; Udgivet: November 21, 2012 |
Copyright: © 2012 Ohno et al. Dette er en åben adgang artiklen distribueres under betingelserne i Creative Commons Attribution License, som tillader ubegrænset brug, distribution og reproduktion i ethvert medie, forudsat den oprindelige forfatter og kilde krediteres
Finansiering:. Dette arbejde blev støttet af Project Research Grant fra Research Institut for Videnskab og Teknologi, Kogakuin Universitet. De finansieringskilder havde ingen rolle i studie design, indsamling og analyse af data, beslutning om at offentliggøre, eller forberedelse af manuskriptet
Konkurrerende interesser:.. Forfatterne har erklæret, at der ikke findes konkurrerende interesser
Introduktion
chitin, en lineær polymer af β-1-4-koblet N Chit1 niveau markant forhøjede i plasma af patienter med Gauchers sygdom, en autosomal recessiv lysosomal storage disorder [4]. Chit1 var den første mammale chitinase, der skal oprenses og klonet [5], [6]. Et recessivt nedarvet mangel på Chit1 aktivitet er almindeligt observeret i kaukasiere [7]. AMCase blev opdaget på grund af dets kompenserende rolle og er opkaldt efter sin sure pH-optimum [8]. Disse mammale chitinaser betragtes som en del af værtens forsvar mod chitin-indeholdende patogener og parasitter [3], [9]. Både Chit1 og AMCase er udskilte proteiner med molekylvægte på ca. 50 kDa. Begge proteiner indeholder en N-terminal katalytisk domæne, en hængselregion og et C-terminalt chitin-bindende domæne [6], [8]. Mus AMCase viser sekvenshomologi til Chit1, med en identitet på 52% og en lighed på 60% [10]. På trods af disse strukturelle ligheder, disse enzymer adskiller sig væsentligt med hensyn til deres enzymatiske adfærd ved sur pH. AMCase viser en markant pH-optimum ved pH 2 og en mindre tydelig optimum ved pH 4-7 [8], mens Chit1 viser kun et bredt pH-optimum på omkring pH 5 [5], [11]. Pattedyr chitinaser har tiltrukket sig betydelig opmærksomhed på grund af deres øgede ekspression i individer med forskellige patologiske tilstande. Chit1 øges hos personer med Gauchers sygdom [4], kronisk obstruktiv lungesygdom (KOL) [12], og Alzheimers sygdom [13] og hos rygere [14]. AMCase ekspression og aktivitet er også opreguleret under allergiske luftvejsresponser i musemodeller for astma [15]. Desuden polymere chitin inducerer AMCase udtryk og rekruttering af immunceller forbundet med allergi og astma [16]. Disse resultater antyder kraftigt, at kitinolytisk enzymer spiller en vigtig rolle i mange patofysiologiske tilstande. bidrag af disse enzymer til patofysiologien af disse sygdomme er dog stadig, der skal fastlægges. kvantificering af de Chit1 og AMCase mRNA niveauer og sammenligningen af disse niveauer med niveauerne af kendte referencepunkter gener er vigtige skridt i at få indsigt i in vivo I denne undersøgelse har vi udviklet en kvantitativ real-time RT-PCR system, der bruger standard DNA produceret ved ligering af cDNA-fragmenter af målgenerne. Dette system gjorde det muligt at kvantificere og sammenligne ekspressionsniveauerne af de chitinaser og reference- generne i samme målestok. Vores resultater viser, at AMCase er et stort transkript i muse maven, hvilket antyder, at det tilsvarende protein fungerer som en fordøjelsessystemet enzym, der nedbryder chitin-holdige fødevarer og som en del af værtens forsvar mod chitin-indeholdende patogener i maveindholdet. Materialer og metoder RNA, RNA Isolation og cDNA Forberedelse mus Total RNA Master Panel (Clontech Laboratories) blev anvendt til at undersøge vævsfordelingen af udskrifter. Vi analyserede fire forskellige embryonale stadier og otte voksne væv. Derudover blev RNA isoleret fra lungerne og maver 3-måneder gamle hanmus. C57BL /6J mus (CLEAR Japan) blev avlet på Riken Brain Science Institute Animal Facility. Alle eksperimenter dyr blev udført i overensstemmelse med de institutionelle retningslinier. Protokollen blev godkendt af Udvalget for den etiske dyreforsøg af Riken Brain Science Institute (Godkendelse nr H19-2B013). Alle operation blev udført ved hjælp af diethylether som et bedøvelsesmiddel, og blev gjort alle bestræbelser på at minimere lidelse. Disse væv til mRNA forberedelse blev leveret af Drs. Miyazaki og Nukina på Riken Brain Science Institute. Totalt RNA blev fremstillet ud fra lunger og maver ved hjælp TRIzol Reagent (Invitrogen) ifølge producentens instruktioner. At fjerne spor af kontaminerende genomisk DNA blev de samlede RNA-prøver behandlet med RQ1 RNase-fri DNase (Promega) ifølge producentens anbefalede protokol. Koncentrationerne af nukleinsyrerne blev bestemt ved at måle absorbansen ved 260 nm ved anvendelse af en BioPhotometer Plus (Eppendorf). Hver af de samlede RNA-prøver (3 ug) blev underkastet revers transkription med vilkårlige hexamerer som primere. Reaktionsblandingen (15 pi) indeholdt enzymet puffer [50 mM Tris-HCI (pH 8,3), 75 mM KCI, og 3 mM MgCl 2], 100 ng af tilfældige hexamerer, 10 mM dithiothreitol og 0,5 mM deoxynukleotid triphosphater (dNTP'er). Efter opvarmning af opløsningen til 60 ° C i 5 minutter og inkubering af blandingen ved 37 ° C i 5 minutter, 200 U af rekombinant murin leukæmivirus revers transkriptase (Invitrogen) blev tilsat, og blandingen blev inkuberet ved 37 ° C i 45 minutter . Den revers transkription blev afsluttet ved opvarmning til 95 ° C i 5 min. Primere for real-time RT-PCR blev udformet på grundlag af Primer Express Software (Applied Biosystems) og blev syntetiseret kommercielt (Sigma-Genosys, Sigma-Aldrich). PCR reaktioner blev udført i et slutvolumen på 13 pi indeholdende 2 × SYBR Green Master Mix (Brilliant II SYBR Green QPCR Master Mix, Agilent), 2,7 ng af muse cDNA eller passende fortyndinger af de eksterne standarder (se nedenfor), og den passende koncentrationer af primerne til den chitinaser, pepsinogen C, glyceraldehyd-3-phosphat-dehydrogenase (GAPDH) eller β-actin. Standard real-time PCR betingelser for systemet (Mx3005P, Agilent) blev anvendt: en initial denaturering og polymerase aktiveringstrin i 10 minutter ved 95 ° C, efterfulgt af 40 cykler af denaturering ved 95 ° C i 1 min, 55 ° C i 30 sek og 72 ° C i 1 min. Smeltekurver blev genereret efter amplificering. PCR-produkterne blev elektroforesebehandlet på en 10% polyacrylamidgel og analyseret ved anvendelse af Luminescent Image Analyzer (ImageQuant LAS 4000, GE Healthcare). PCR opløsning blev behandlet med ExoSAP-IT (USB-produkter) i overensstemmelse med producentens instruktioner for at fjerne ikke-inkorporerede primere og dNTP'er, og produkterne blev sekventeret under anvendelse af ABI PRISM Big-Dye Terminator v3.1 Cycle Sequencing Kit og et 3130 Genetic Analyzer ( Applied Biosystems). Nukleotidsekvenserne af de udvalgte for real-time PCR-primere er vist i tabel S1. standard template cDNA (913 baser) til kvantificering af transkriptniveauer af real-time PCR blev konstrueret som følger. CDNA-fragmenterne, der dækker det PCR-target region plus 9-120 baser af de flankerende regioner i AMCase, Chit1, pepsinogen C, GAPDH, og β-actin blev amplificeret fra muse maven cDNA ved PCR under anvendelse KOD Plus DNA-polymerase (Toyobo) og oligonukleotider primer indeholder restriktionsstederne af Bgl For at validere vores absolutte real-time PCR-metode, vi forberedt fuld kodende cDNA'er ved PCR under anvendelse af primersæt der er anført i tabel S3. Fem cDNA'er dækker hele den kodende regioner af to chitinaser (Chit1 og AMCase), og henvisningen gener (GAPDH, p-actin og pepsinogen C) blev amplificeret ved PCR fra muse mave-cDNA under anvendelse af KOD Plus DNA-polymerase og blev subklonet ind i pGEM-T let vektor gennem TA kloning, som beskrevet ovenfor. Sekvenserne af cDNA'erne blev verificeret ved sekventering, som beskrevet i figur S1. De subklonede fragmenter blev reamplificeret fra plasmid-DNA'erne med de samme primere og blev derefter anvendt som hele den kodende region cDNA'er. Den molære koncentration af multigenfamilien-holdigt DNA standard blev beregnet baseret på koncentrationen og molekylvægten. Serielle fortyndinger blev fremstillet startende med den standardskabelon koncentration, hvilket gav en Ct på ca. 13 (Ct = fraktioneret tærskelværdi cyklus værdi). Standarden DNA blev underkastet 10 gange serielle fortyndinger, der spænder fra 10 0 til 10 7 molekyler og de portioner blev holdt nedfrosset ved -20 ° C indtil anvendelse. hver prøve blev forstærket i tre eksemplarer, og hvert forsøg blev gentaget mindst to gange. Under anvendelse af standardkurven, blev antallet af Chit1, AMCase, GAPDH, β-actin, og pepsinogen C mRNA-molekyler ekstrapoleret automatisk ved hjælp af MxPro QPCR Software version 4.10 (Agilent). Alle værdier blev udtrykt som molekyler pr 10 ng totalt RNA. I nogle tilfælde blev de værdier normaliseret mod niveauerne af GAPDH eller β-actin mRNA. Til evaluering vores nuværende metoder vi også ansat den ΔΔ Ct-metoden [21], som kræver måling af tærskelcyklus (Ct) af målgener. Ct-værdier blev beregnet af MxPro QPCR Software hjælp GAPDH eller β-actin som normalizer. Etablering af Real-time PCR-analyse Etableringen af et pålidelig metode til måling af ekspressionen af chitinase udskrifter er et vigtigt skridt i at undersøge betydningen af mus chitinaser. Real-time RT-PCR er i øjeblikket den mest følsomme kvantitative metode til påvisning af både med lav hyppighed mRNA'er og rigelige transkripter. Vi sammenlignede første gen-ekspressionsniveauer af Chit1 og AMCase gener (se figur 1). For at evaluere de chitinase niveauer, vi brugte to husholdning gener, GAPDH og β-actin, som referencepunkter gener, fordi de konstitutivt udtrykkes på højt niveau i de fleste væv og celler [22]. Desuden valgte vi pepsinogen C (også kendt som progastricsin) som reference gen i maven. Pepsinogen C er en asparaginprotease, der fungerer som et fordøjelsesenzym og produceres i maven. Dette enzym udgør en hovedbestanddel af maveslimhinden [23]. Ved hjælp af disse tre referencepunkter gener, afprøvede vi genekspressionen af Chit1 og AMCase i musevæv (figur 1). Vi først designet flere sæt af primere for kvantitativ PCR og evalueret deres egnethed baseret på, om de gav enkeltprodukter , hvilket fremgår af en enkelt smeltetemperatur (Tm) og et enkelt bånd på en 10% polyacrylamidgel. Nukleotidsekvenserne af produkterne blev også kontrolleret. For at undersøge specificiteten af primerne, blev hver af PCR-produkterne amplificeret under realtids-PCR betingelser ved anvendelse af en muse væv cDNA blanding bestående af væv fra fire embryonale stadier og otte voksne væv og blev analyseret i kombination med forskellige metoder. Som vist i figur 2A-E, kun én top dukkede op i dissociation kurverne for Chit1 (Tm = 79,7 ° C), AMCase (Tm = 79,1 ° C), GAPDH (Tm = 81,4 ° C), β-actin (Tm = 82,0 ° C) og pepsinogen C (Tm = 81,4 ° C). Ved afslutningen af PCR, analyserede vi produkterne på en 10% polyacrylamidgel. Gelelektroforese viste klare enkeltbånd på de forventede størrelser af Chit1 (69 bp), AMCase (81 bp), GAPDH (77 bp), β-actin (71 bp) og pepsinogen C (82 bp) (figur 2F). Nukleotidsekvenserne af PCR-produkterne blev bestemt direkte som beskrevet i afsnittet Materialer og fremgangsmåder. Vi bekræftede, at PCR-produkterne blev amplificeret fra mål-cDNA'er (se figur 3B). Disse resultater viser, at PCR-produkterne er særlige amplikoner fra målet cDNA'erne og at mispriming er ubetydelig under vores eksperimentelle betingelser. Vi søgte at sammenligne ekspressionsniveauerne af to chitinaser og tre referencepunkter gener på samme skala (figur 1). Til dette formål har vi oprettet en kvantitativ real-time PCR-system, for hvilken en standard template var nødvendig for nøjagtig kvantificering (figur 3A). Vi konstruerede en standard template DNA til realtids-PCR ved at ligere de fem target-fragmenter i en en-til-en-forhold, og derefter dette ligerede DNA fragment blev klonet ind i plasmidvektoren som beskrevet i materialer og fremgangsmåder (figur 3A) . Den 913-nukleotid-lang template DNA indeholdt fem cDNA-fragmenter dækker PCR-target region plus 9-120 baser af de flankerende regioner og indeholdt restriktionsstederne af Bgl Validering af standardkurven og kvantitativ real-time PCR System Den kvantificering af både chitinaser og reference- mRNA afhængig standardkurver. Seriefortyndinger af standard template-DNA blev anvendt til at konstruere en standardkurve for at sammenligne og evaluere real-time RT-PCR kvantificering strategier, der blev anvendt til at analysere de fem mRNA'er. Hver standardkurve blev genereret ved anvendelse af 10-gange serielle fortyndinger af standard DNA og de fem forskellige primerpar (figur 4A-E, venstre). Eksponentiel amplifikation blev opretholdt over en lang række cyklusser, hvilket gav et dynamisk område på syv størrelsesordener (figur 4A-E, venstre). Ved at bruge standard template DNA, der indeholder de fem cDNA fragmenter, kan lige mængder tildeles alle fem gener i hver fortynding brug for at konstruere standard kurver (Figur 4A-E, til højre). For at teste absolut lighed af kurverne, blev en kendt koncentration af den fulde kodende cDNA amplificeret og efterfølgende analyseret som en ukendt prøve. Dette assay blev udført for at verificere, at hver testet fortynding resulterede i den forventede mængde. Som vist i figur 4A-E, højre blev lige mængder observeret for hver testet fortynding anvendes til at konstruere standardkurven (se figur S2). Kvantificeringen af med lav hyppighed transkripter og rigelige transkripter tillader os at validere følsomhed og pålidelighed realtids-PCR, hvilket indikerer, at vores real-time PCR-metoden tilbyder et stort dynamisk område på kvantificering, høj nøjagtighed og høj følsomhed (figur 4A- E, højre). Således er vores real-time PCR-metode giver pålidelige værdier for to chitinase gener og som reference gener på samme skala. For at studere in vivo Vi derefter sammenlignet forholdene mellem AMCase til Chit1. Kopiantallet af hvert mRNA blev bestemt under anvendelse af samme fortyndinger. Vi fandt, at maven og mandibulære kirtel overvejende udtrykt AMCase mRNA (figur 5C, øvre panel). De andre undersøgte væv i denne undersøgelse produceret mere AMCase end Chit1, og Chit1 var fremherskende kun i øjnene (figur 5C, lavere panel). Vi næste undersøgte den relative kvantificering i de oprindelige data præsenteret i figur 5, der blev udtrykt som molekyler pr 10 ng af total RNA, ved anvendelse af housekeeping-gener som beskrevet i Materialer og metoder. Resultaterne blev vist som underbyggende oplysninger i figur S3 (data blev normaliseret ved GAPDH) og figur S4 (normaliseret ved β-actin). Derefter analyserede vi data vist i figur 5 ved ΔΔ Ct-metoden [21], og der er vist som supplerende oplysninger i figur S5 (normaliseret ved GAPDH) og figur S6 (normaliseret ved β-actin). Når man sammenligner den relative ekspression detekteret ved de foreliggende absolutte og relative kvantificering, var der ingen signifikant forskel blandt de data, undtagen for Chit1 mRNA-ekspression, som var mest rigeligt udtrykt i øjnene, når normaliseret ved β-actin (se figur 5, figur S3 og Figur S4). Lignende resultater blev også opnået ved relativ kvantificering under anvendelse af ΔΔ Ct-metoden [21] (se figur 5, figur S5 og S6 figur). Men vi kunne ikke umiddelbart vurdere gensidige ekspressionsniveauer af Chit1 og AMCase når analyseret af ΔΔ Ct-metoden. Mange undersøgelser har været udført på patofysiologien af mammale chitinaser i lungevæv. I denne undersøgelse viste vi, at AMCase mRNA overvejende udtrykkes i muse mave væv (figur 5). Vi sammenlignede også ekspressionsniveauerne af de chitinaser og referencematerialer gener ved anvendelse cDNA'erne fremstillet ud fra de lunge- og mave væv af 3-måneder gamle mus (n = 5). De kvantitative data er vist i figur 6. Når Chit1 niveauer blev sat til 1,0, de relative ekspressionsniveauer af cDNA'er var AMCase, 14; GAPDH, 196; og β-actin, 681 i muse lungevæv (figur 6A). Dette resultat indikerer, lungevæv udtrykker mere AMCase end Chit1, selvom AMCase ekspressionsniveauet var lavere end den af de to housekeeping-gener. I mave væv, når det Chit1 blev sat til 1,0, de relative ekspressionsniveauer var AMCase, 721; pepsinogen C, 2261; GAPDH, 61; og β-actin, 127 (figur 6B). Både GAPDH og p-actin gener er velkendte husholdningsgener og er konstitutivt udtrykt i høje niveauer i de fleste væv og celler. Pepsinogen C (progastricsin) er en asparaginprotease, der fungerer som et fordøjelsesenzym og produceres i maven. Dette enzym er en vigtig del af maveslimhinden [23]. Ekspressionsniveauet af AMCase var meget højere end for GAPDH og β-actin og var sammenlignelig med niveauet af pepsinogen C. Disse resultater indikerer, at maven AMCase er et stort transkript i maveslimhinden og foreslå, at dette enzym kan forventes at spille vigtige fysiologiske roller i maven. Vi har også fornyet overvejelse relative udtryk for Chit1, AMCase og pepsinogen C vist i figur 6 ved hjælp af rengøring gener ved nuværende fremgangsmåde som beskrevet ovenfor. Resultaterne blev vist som underbyggende oplysninger i figur S7 (normaliseret ved GAPDH) og figur S8 (normaliseret ved β-actin). Der var ingen signifikant forskel mellem figur 6, figur S7 og Figur S8. Både Chit1 og AMCase menes at støtte i værtens forsvar mod chitin-holdige patogener [3], [ ,,,0],9]. Desuden AMCase er et effektormolekyle i allergisk inflammation. Disse kitinolytiske enzymer spiller en vigtig rolle i patofysiologien af allergiske luftvejsresponser i musemodeller for astma. Relativt lidt er kendt, men om in vivo Kvantitativ real-time RT-PCR repræsenterer et vigtigt fremskridt i kvantificere mRNA og muliggør påvisning af ekspressionen af et særligt gen af interesse på det molekylære niveau . Der er mange rapporter om påvisningen af ekstremt lave niveauer af mRNA anvender denne teknologi. Imidlertid real-time RT-PCR er ikke en meget anvendt, fordi den stadig har begrænsninger med at kvantificere multiple mRNA'er, der anvender samme skala. Vi konstruerede en standard template DNA omfattende fem cDNA-fragmenter (hver -200 baser lange). Disse fragmenter inkluderet fragmenter til to chitinaser, én markør og to husholdningsgener og blev kombineret i en-til-en-forhold (figur 3A). GAPDH og β-actin, de mest almindeligt anvendte husholdning gener, blev inkluderet som interne kontroller og referencer til PCR-amplifikation. Derudover brugte vi pepsinogen C som markør-gen, fordi det er produceret på et højt niveau i maven [23]. Denne fremgangsmåde tillod den absolutte kvantificering af antallet af Chit1 og AMCase mRNA-molekyler, såvel som antallet af henvisningen mRNA-molekyler pr 10 ng af total RNA (mol /10 ng) (figur 5 og figur 6). Desuden kunne vi undersøge relative kvantificering af Chit1 og AMCase mRNA niveauer ved hjælp af to husholdning gener, GAPDH og β-actin (Støtte Information Figur S3 og figur S4). Relativ kvantificering er lettere at udføre end absolut kvantificering, fordi en kalibreringskurven er ikke nødvendig. I relativ kvantificering, er mRNA-niveauer af genet af interesse i forhold fra beskrivelserne husholdningsgener [21]. Denne kvantificering metode imidlertid ikke at sammenligne niveauet for de forskellige gen udskrifter i samme målestok. Selvom vores metode kræver flere trin i forbindelse med opførelsen af den standard DNA og med valideringsprocesser, kan denne metode give genekspression data, der er direkte sammenlignelige mellem gener (figur 5 og figur 6). Vores realtids-PCR gav et stort dynamikområde for kvantificering og høj følsomhed, og kvantificering af med lav hyppighed transkripter tilladt os at validere følsomhed og pålidelighed vores metode (figur 4). Denne teknik er meget velegnet til kvantificering og sammenligning af mRNA niveauer på tværs af flere gener, der bruger den samme skala. Vores nuværende metode kan anvendes på biomedicinsk teknik samt kliniske og praktiske anvendelser. I denne undersøgelse fandt vi, at AMCase mRNA syntetiseres på ekstraordinært høje niveauer i musen maven i forhold til niveauerne af husholdning gener (figur 5 og figur 6). Niveauet af AMCase mRNA er sammenlignelig med den for pepsinogen C (progastricsin) mRNA, en vigtig komponent i maveslimhinden i maven (figur 6B). Ud over maven, blev også fundet de mandibulære kirtler til at udtrykke store mængder af AMCase (figur 5). Som til syntese mammale chitinase mRNA'er, er både maven og mandibulære kirtler betragtes som bemærkelsesværdige væv til fremstilling af enorme AMCase i mus. Saltsyre udskilles i maven, hvilket skaber sure betingelser for fordøjelsen af proteiner ved pepsin ved ca. pH 2 [23], [24]. Mus AMCase viser dyb syre stabilitet og er mest aktive ved pH 2,0, som adskiller den fra andre mus enzymer [8]. Den usædvanlige pH-stabilitet og afhængighed af muse AMCase på sure betingelser tillader effektiv fordøjelse af chitinous materialer under sure betingelser. Den iagttagelse, at AMCase overvejende udtrykkes i maven peger på dens mulige funktion i forarbejdning af fødevarer. Vilde mus spiser chitin-holdige fødevarer såsom insekter, hvorimod mus holdes i laboratoriet spiser tørfoder indeholdende tørrede ølgær, som også indeholder chitin i cellevæggen. Således kan AMCase fungere som et fordøjelsesfremmende enzym, der nedbryder polymere chitin i maveindhold. Det højeste niveau af Chit1 blev også udtrykt i musen maven. Imidlertid Chit1 ekspressionsniveau i maven var 1/721 det færdige AMCase ekspressionsniveauet og var lavere end ekspressionsniveauerne af to husholdningsgener, GAPDH og β-actin (figur 6A). Hertil kommer, at Chit1 ikke besidder nogen kitinolytiske aktivitet ved pH for mavesafter, som er omkring pH 2 [5], [11]. Det er blevet vist, at Chit1 produceres på steder med nær-neutral pH, såsom den ikke-glandulære del af maven og tyndtarmen [25]. Således ikke Chit1 ikke bidrager til chitinase aktivitet i muse maven. Selvom AMCase udtrykkes overvejende i maven og mandibulære kirtler, alle andre undersøgte væv i denne undersøgelse udtrykte lave, men detekterbare, niveauer af Chit1 og AMCase . Ekspressionsniveauerne af Chit1 og AMCase var lavere end husholdning gener i disse væv. Chit1 viser et bredt pH-optimum på omkring pH 5 [5], hvorimod den primære optimale pH AMCase er pH 2 og den sekundære pH-optimum er ved pH 4-7, med AMCase bibeholder mindre end 30% af sin aktivitet ved pH 2 i dette pH-område [8]. Både Chit1 og AMCase fordøje naturlig kitin og chitosan via endo-kitinase aktivitet og producere chitobiose [8], [26]. Selvom AMCase er dominerende over Chit1 i mange mus væv, kan der ikke være nogen signifikante forskelle i deres kitinolytisk aktivitet i muse væv. Flere AMCase mRNA end Chit1 mRNA blev observeret i de fleste mus væv, bortset fra øjnene. Vores resultater antyder også, at genekspression af Chit1 kan udviklingsmæssigt reguleret, hvilket antyder, at det spiller en rolle i ontogenese. Studere reguleringen af ekspressionen af de pattedyr chitinaser kunne give indsigt i de fysiologiske roller af disse enzymer. En detaljeret karakterisering af promotor-regioner Chit1 og AMCase gener samt identifikation af cis Selvom pattedyr ikke frembringer chitin eller chitin synthase, de er konstant udsat for denne polymer ved udsættelse for chitin-holdige parasitter og patogener. Substratet for mammale chitinaser er formentlig miljømæssig chitin, som den, der findes i svampe eller parasitiske nematoder. I lungerne, kan begge enzymer fungere som en del af værtens forsvar mod chitin-indeholdende patogener, såsom miljømæssig skimmel og mider, som inducerer luftvejs allergi.
acetyl-D-glucosamin, er den anden mest forekommende polysaccharid findes i naturen. Den fungerer som en større strukturel bestanddel af svampe, krebsdyr og insekter, men er ikke fundet i pattedyr [1]. Chitinaser hydrolyserer de β-1-4 glycosidbindinger af kitin polymer. Selvom pattedyr ikke producerer kitin eller dets syntase, de udtrykker to aktive kitinaser, chitotriosidase (Chit1) og sure pattedyr chitinase (AMCase) [2], [3].
regulering af pattedyr chitinaser. For nylig er real-time RT-PCR blevet anvendt til at kvantificere mRNA-niveauer i mange genekspressionsstudier [17] - [20], fordi denne metode er tilstrækkelig følsom til at detektere mRNA fra selv en enkelt celle. Real-time PCR almindeligvis involverer normalisering af ekspressionsniveauerne af genet af interesse med specifikationerne for de housekeeping-gener, der menes at være konsekvent udtrykkes i alle prøverne. Denne kvantificering metode imidlertid ikke at sammenligne niveauet for de forskellige gen udskrifter i samme målestok.
Real-time PCR
Konstruktion af standard DNA
II, Xho
jeg, Pst
jeg, eller Ikke
i (ved 5'- og 3 ' - ends) ifølge fabrikantens protokol. De forreste og reverse primere er anført i tabel S2. PCR-produkterne blev oprenset ved anvendelse af Wizard SV Gel og PCR Clean-Up System (Promega) og derefter spaltet med de tilsvarende restriktionsenzymer. DNA-fragmenterne blev oprenset under anvendelse af agarosegelelektroforese og Clean-Up System og derefter ligeret sammen med T4 DNA-ligase (Toyobo). De ligerede fragmenter blev amplificeret under anvendelse af forward primeren 5'-GTGGATTCTGTGCCGACAAAGCAGATGGCC-3 'og den reverse primer 5'-TGGGTACATGGTGGTACCACCAGACAGCAC-3' med KOD Plus DNA-polymerase. 3'-dA blev tilsat til den amplificerede DNA under anvendelse Takara Taq HS (Takara Bio), og produktet blev oprenset ved gelelektroforese som beskrevet ovenfor. Det resulterende DNA blev klonet i pGEM-T Easy-vektoren (Promega) gennem TA kloning, ifølge producentens instruktioner. Nukleotidsekvensen af det resulterende plasmid blev bekræftet ved sekventering. Det lineariserede multigenfamilie-holdigt DNA-fragment blev fremstillet ved reamplifikation fra plasmid-DNA med de samme primere ved PCR under anvendelse KOD Plus DNA-polymerase. Fragmenterne blev renset og kvantificeret som beskrevet ovenfor og anvendt som standard DNA.
Udarbejdelse af Fem cDNA dækker hele Coding Region
standardkurver
Kvantificering af mRNA af Real-time PCR ved hjælp Standard Kurver eller ΔΔ Ct Method
Resultater
Konstruktion af Standard Template DNA til kvantificering og sammenligning af Gene Expression Blandt Fem Gener
II, Xho
I , Pst
i og Ikke
i (se detaljer i figur 3B).
Angivelse af Chit1 og AMCase i Mouse væv
regulering af Chit1 og AMCase genekspression, total RNA-prøver ekstraheret fra fire embryonale stadier, og fra forskellige væv fra voksne, blev analyseret med en kvantitativ real-time PCR-analyse under anvendelse af enkelt standard DNA (figur 3). Resultaterne blev udtrykt som molekyler pr 10 ng af total RNA (figur 5 og figur 6). Både Chit1 og AMCase mRNA'er blev bredt udtrykt i musevæv (figur 5A og 5B). Klare væv særlige blev observeret i udtrykket hos såvel chitinase mRNA. blev påvist høje niveauer af Chit1 mRNA i muse maven (figur 5A, øvre panel), efterfulgt af øjne og lungen. Chit1 mRNA blev påvist i lave, men let påviselige, niveauer i andre væv (figur 5A, lavere panel). AMCase mRNA blev overvejende detekteret i maven, efterfulgt af mandibulære kirtel (figur 5B, øvre panel), men var også til stede i andre væv (figur 5B, nedre panel).
Analyse af Chit1, AMCase, Pepsinogen C, GAPDH og β-actin i Lung og Mave Væv
Diskussion
gensidig ekspressionsniveauer af Chit1 og AMCase. Kvantificeringen af mRNA'er kan generere nyttig og biomedicinsk relevant information. I denne undersøgelse har vi etableret en kvantitativ real-time PCR-system stand til at bestemme mRNA-niveauerne af to mammale chitinaser og sammenligne disse niveauer med de reference- gener efter samme tabel. Vores resultater viser, at AMCase overvejende udtrykkes i musen maven.
- og trans
virkende faktorer vil være forpligtet til at forstå den selektive udtryk for disse enzymer <. br>
 Sådan får du perfekte afføringer (i radioen)
Sådan får du perfekte afføringer (i radioen)
 Hvornår skal jeg se lægen for blod i afføringen?
Hvornår skal jeg se lægen for blod i afføringen?
 Fedttabsquizzen:6 grunde til, at du ikke taber dig
Fedttabsquizzen:6 grunde til, at du ikke taber dig
 Steve's SCD Healing Journal:Uge 26 – Coconut Water is Mother Nature's Gatorade!
Steve's SCD Healing Journal:Uge 26 – Coconut Water is Mother Nature's Gatorade!
 Håndtering af fordøjelsesproblemer efter en hysterektomi
Håndtering af fordøjelsesproblemer efter en hysterektomi
 SANDHEDEN om glutenfri mad — hjælper det eller skader tarmen?
SANDHEDEN om glutenfri mad — hjælper det eller skader tarmen?
 Opbevaring af dit SIBO butiksskab – del 1
At opbevare dit SIBO butiksskab med de rigtige ingredienser kan gøre hele forskellen, når du behandler din SIBO. Årstiderne skifter. Uanset om du er på den nordlige halvkugle og varmer op, eller den s
Opbevaring af dit SIBO butiksskab – del 1
At opbevare dit SIBO butiksskab med de rigtige ingredienser kan gøre hele forskellen, når du behandler din SIBO. Årstiderne skifter. Uanset om du er på den nordlige halvkugle og varmer op, eller den s
 Drug Stelara kan lindre Crohns sygdom
Seneste fordøjelsesnyt Når nyretransplantation mislykkes, er det bedst at prøve igen Lad ikke halsbrand ødelægge din feriefest Efterspørgslen efter levertransplantation stiger kraftigt Lavdosis CT-sc
Drug Stelara kan lindre Crohns sygdom
Seneste fordøjelsesnyt Når nyretransplantation mislykkes, er det bedst at prøve igen Lad ikke halsbrand ødelægge din feriefest Efterspørgslen efter levertransplantation stiger kraftigt Lavdosis CT-sc
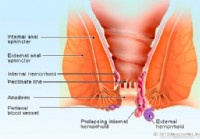 Indre og ydre hæmorider
Hvad skal du vide om hæmorider? Billede af indre og ydre hæmorider Hvad er hæmorider? Hæmorider (bunker) er blodkar placeret i de glatte muskler i væggene i endetarmen og anus. De er en normal del a
Indre og ydre hæmorider
Hvad skal du vide om hæmorider? Billede af indre og ydre hæmorider Hvad er hæmorider? Hæmorider (bunker) er blodkar placeret i de glatte muskler i væggene i endetarmen og anus. De er en normal del a