Die markante Magenflüssigkeit Proteom bei Magenkrebs zeigt ein Multi-Biomarkers Diagnoseprofil für Zusammenfassung
Hintergrund
Gesamt-Überleben Magenkrebs schlecht bleibt vor allem, weil es keine zuverlässigen Methoden für sehr heilbar frühen Stadium der Erkrankung zu identifizieren. Multi-Protein-Profilierung von Magensaft, von der anatomischen Stelle der Pathologie erhalten, könnte offenbaren diagnostische Proteom Fingerabdrücke.
Methoden
Proteinprofile von Magensaft Proben von 19 Magenkrebs erzeugt wurden und 36 gutartige gastritides Patienten nach elektiven unterzogen, klinisch -indicated Gastroskopie unter Verwendung von oberflächenverstärkten Laser-Desorptions /Ionisations-Time-of-flight-Massenspektrometrie auf mehreren Proteinchip-Arrays. Proteom-Merkmale wurden von Bedeutung Analyse von Microarray-Algorithmus und Zweiweg-clustering hierarchischen verglichen. analysyis von Microarray, eine zweite verblendet Probensatz (24 Magenkarzinome und 29 klinisch gutartig gastritides) für die Validierung verwendet wurde.
Ergebnisse von Bedeutung
60 Proteom-Züge waren hochreguliert und 46 wurden nach unten reguliert bei Magenkrebs Proben (p
< 0,01). Multimarker-Clustering zeigte zwei unterschiedliche Proteom-Profile unabhängig von Alter und ethnischer Zugehörigkeit. Achtzehn von 19 Krebsproben zusammen gruppiert (Sensitivität 95%), während 27/36 von Nicht-Krebs-Proben in einer zweiten Gruppe gebündelt. Neun Nicht-Krebs-Proben, die mit Krebsproben gruppierten inklusive 5 prämalignen Läsionen (1 adenomatöser Polyp und 4 intestinale Metaplasie). Validierung eines zweiten Probensatz mit zeigte die Sensitivität und Spezifität 88% und 93% betragen, beziehungsweise. Positive prädiktive Wert der kombinierten Daten betrug 0,80. Ausgewählte Peptidsequenzieren Pepsinogen C identifiziert und Pepsin A Aktivierungspeptid als signifikant herunterreguliert und alpha-Defensin als signifikant hochreguliert.
Fazit
Dieser einfache und reproduzierbare Multimarker Proteom-Test konnte die klinische gastroskopischen Bewertung der symptomatischen Patienten ergänzen zu verbessern diagnostische Genauigkeit für Magenkrebs und prämalignen Läsionen.
Hintergrund
Im Gegensatz zu anderen häufigen Krebsarten, ist die Prognose für die meisten Patienten mit Magenkrebs arm und hat wenig in den letzten Jahrzehnten verbessert. Fünf-Jahres-Überlebensrate für Magenkrebs sind deutlich niedriger als alle wichtigen Krebsarten außer Krebserkrankungen der Leber, der Bauchspeicheldrüse und Speiseröhre [1]. Da Magenkrebs frühen Stadium hat eine viel bessere Prognose (5-Jahres-Überlebens ca. 90%) als fortgeschrittenem Magenkrebs (5-Jahres-Überlebens 3-10%) [2, 3], die globale Sterblichkeit von Magenkrebs sollte wesentlich zu verringern, indem Maßnahmen, die in Downstaging von Tumoren zum Zeitpunkt der Erstdiagnose zur Folge haben.
Obwohl Gastroskopie der Goldstandard für Magenkrebs-Diagnose ist, ist die Genauigkeit nicht so hoch wie bei gutartigen Magenerkrankungen wie Magengeschwüren, insbesondere in geografischen Regionen ist von niedrigen bis mittleren Magenkrebs-Prävalenz. Der Prozentsatz von Anrufen in Abwesenheit der Krebsdiagnose, angegeben als 4,6%, 14% und sogar 33% [4-6], nicht unerheblich ist. Auch in Japan wurde die falsch-negative Rate berichtet 19% sein [7]. Diese Daten sind konsistent mit dem positiven prädiktiven Wert von nur 0,4 bis 0,7 für die endoskopische Diagnose von Magenkrebs in verschiedenen Zentren [8-10]. Obwohl der Anteil der verpassten Diagnosen klein erscheint, ist die absolute Zahl der Patienten, verweigert der Nutzen der Diagnose in einem heilbaren Stadium nicht zu vernachlässigen ist. Selbst bei einer bescheiden niedrige False-Positive-Diagnoserate von 5%, mehr als 47.000 Magen-Krebs in einer geringen Prävalenz Land allein (USA) in einem einzigen Jahr, 2000 [11] verpasst hätte. Endoskopische Beurteilung umfasst häufig Schleimhautbiopsien, aber es gibt keine klinischen Standards entweder für die optimale Anzahl von Biopsien oder die anatomischen Regionen, die abgetastet werden soll. Eine häufig zitierte Empfehlung ist mindestens sieben Biopsien zu nehmen richtig Magenkrebs [12] zu diagnostizieren. In dieser Studie jedoch voll 17% aller Läsionen anschließend maligne erwiesen wurden bei der Endoskopie als gutartig. So leidet die endoskopische Schleimhaut Untersuchung von Interobserver Variation, suboptimalen Korrelation mit der Histopathologie, Schwierigkeiten bei der Submukosa-Krebs und ungehinderte Visualisierung aller anatomischen Subregionen Erfassung z.B. . Nach vorherigem Magen-Operation [13, 14]
Magenflüssigkeit aus einer Mischung von sezernierten löslichen und exfolierten zellulären Proteine aus der gesamten Magenschleimhaut besteht - einschließlich der Regionen, die nicht ausreichend durch fiberoptische Gastroskopie beurteilt werden kann. Wir folgerten daher, dass das Proteom-Profil von Magenflüssigkeit, in der Regel als Abfall angesehen Nebenprodukt bei gastroskopischen Prüfung, nutzbringend herkömmliche klinische Bewertung ergänzen könnte eine "molekulare Biopsie", die effektiv Proben die gesamte Magenschleimhaut, vor allem als Proteinnachweisverfahren durch die Bereitstellung solcher wie Massenspektrometrie können sehr empfindlich sein. Wenn im Verlauf klinisch indiziert Gastroskopie durchgeführt wird, hat Magenflüssigkeit erhalten nicht die Invasivität des Verfahrens erhöhen. Im Gegensatz zum Plasma-Proteom ist die Magenflüssigkeit Proteom wahrscheinlich weniger komplex zu sein, aber in krankheitsspezifischen Biomarkern angereichert, wobei direkt an der erkrankten Stelle erzeugt. Die gleichen Biomarker, auch dann vor, wenn im Plasma kann über die Grenzen der Erkennung und vermischt mit anderen häufiger systemische Proteine verdünnt werden, die gleichzeitig pathophysiologischen Bedingungen (zB komorbiden Krankheiten) reflektieren, anstatt anatomischen ortsspezifische Erkrankung.
Wir haben durch Profilierungs lösliche sezernierte Peptide, die in endoskopisch aspiriert Magenflüssigkeit und Proteinen extrahiert Produkten aus expandiertem Epithelzellen einen neuartigen Ansatz zur Entwicklung von Biomarkern für Magenkrebs untersucht, auch während der Endoskopie durch oberflächenverstärkte Laser-Desorptions-Ionisations-Flug (SELDI erholt TOF) Massenspektrometrie. Unsere Ergebnisse legen nahe, dass mehrere Protein-Biomarkern aus einem Organ-spezifische Quelle Magenflüssigkeit, dh zur Verbesserung der diagnostischen Genauigkeit von Gastroskopie eine ausgeprägte Magenkrebs Signatur, die Verdienste weitere Entwicklung als Instrument erzeugen und hat das Potenzial für frühen Stadium Magenkrebs und prämalignen Erkennung Läsionen (intestinale Metaplasie und Dysplasie).
Methoden
klinischen Proben
Magenflüssigkeiten während Gastroskopie im Singapore General Hospital gesehen von über Nacht gefastet Patienten erhalten wurden. Das Studienprotokoll wurde von der Ethikkommission des Singapore General Hospital genehmigt. und entsprachen den Bestimmungen der Deklaration von Helsinki 1995 Indikationen für die Gastroskopie ausschließlich klinische und waren unabhängig von der Studie. Eine erste Analyse wurde auf einem Trainingssatz von 19 Proben aus histologisch nachgewiesenen Magen-Adenokarzinome durchgeführt (13 Darm-Typ, 4 diffuse Art, 1 Mischtyp, 1 unbestimmt) [15] und 36 Proben von Patienten mit klinisch gutartigen Magenbedingungen. Das Durchschnittsalter von 19 Magenkrebs-Patienten (13 männlich, 6 weiblich; 17 Chinesen, zwei Inder) war 68 Jahre. Verteilung von American Joint Committee on Cancer (AJCC) klinische Inszenierung war der Stufe 0 (1 Patient), Stufe I (4 Patienten), Stufe II (2 Patienten), im Stadium III (2 Patienten) und Stufe IV (10 Patienten). Das mittlere Alter der 36 Patienten mit gutartigen Magenbedingungen (19 männlich, 17 weiblich; 33 Chinesisch, 2 Malay, 1 Inder) war 57 Jahre. Klinische Diagnose nach der Endoskopie nicht-Krebs-Patienten normal waren (9), Antrum-Gastritis (9), Gastritis (6), Geschwüre (4), Hiatushernie (3), hyperplastische Polypen (2), Barrett-Ösophagus (1), Fundus Narbe (1) und adenomatöser Polyp (1). die Klassifikationsalgorithmus aus dem Trainingssatz entwickelt wurde durch verblendet Analyse eines Validierungssatzes getestet
histologisch bestätigtem Magenadenokarzinomen eines anderen 24 aus (10 intestinalen Typ, 7 diffuse Art, 1 gemischt Typ, 5 unbestimmt, 1 neuroendokrinen) und 29 klinisch gutartigen Magenproben. Das mittlere Alter dieser 24 Magenkrebs-Patienten (18 Männer, 6 Frauen, 21 Chinesen, 3 Malay) war 70 Jahren. Verteilung von AJCC klinischen Inszenierung war im Stadium I (5 Patienten), Stufe II (4 Patienten), im Stadium III (2 Patienten) und Stufe IV (12 Patienten). Ein Patient in der Validierungssatz sank weitere Untersuchungen und nicht inszeniert werden konnte. Das Durchschnittsalter von 29 Nicht-Krebs-Patienten (11 männlich, 18 weiblich; 26 Chinesisch, 2 Indian, 1 Malay) war 47 Jahre. Klinische Diagnose nach Gastroskopie von Nicht-Krebs-Patienten waren Gastritis (14), Fundusdrüsen Polypen (2), akutes Magengeschwür (2), Duodenitis (2), Hiatushernie (1) und normal (8).
Keines die Magenkrebs-Patienten hatten zum Zeitpunkt der Gastroskopie jede Form der Krebsbehandlung erhalten.
Ausbildung und Validierung Fälle zusammengenommen, 19% (8/43) und 29% (19/65) der Patienten mit Magenkrebs und gutartigen Magen Bedingungen waren jeweils positiv für H. pylori
, eine Differenz, die von Fisher-Test (2-seitig p
Wert = 0,4508).
Probensammlung und Verarbeitung
Magensaft nicht signifikant war, aspiriert in der Endoskopie einen sterilen Behälter zu Beginn, eine anonymisierte Code und sofort auf Eis gestellt zugeordnet. Blood- oder Galle befleckten Proben wurden abgelehnt. Nur klinisch verdächtige Schleimhautläsionen wurden im Ermessen des endoscopist biopsiert. Magenflüssigkeiten wurden bei 4 ° C bei 180 g für 6 Minuten zentrifugiert, von dem der Überstand erneut bei 16 100 g für 30 Minuten bei 4 ° C zentrifugiert. Pellets aus beiden Zentrifugationen wurden vereinigt. Die Hochgeschwindigkeitsüberstände wurden getrennt von den Pellets bei -80 ° C.
Protein Profilierungs
Nach dem Auftauen gelagert wurden 10 ul jeder Probe Magenflüssigkeit zu verschiedenen chemischen Oberflächen von Proteinarrays angewendet wurde (Ciphergen Biosystems Inc, California USA,): (a) Kupfer (II) immobilisierte Metall Affinitätseinfangen (IMAC3) in Gegenwart von 100 &mgr; l 1 mol /l Harnstoff, 1 g /L 3 - [(3-Cholamidopropyl) dimethylammonio] -1-propansulfonat ( CHAPS), 0,3 mol /L KCl, Protease-Inhibitor-Cocktail (Roche Diagnostics, Mannheim, Deutschland), 50 mol /L Tris-HCl, pH 7,5; (B) Schwach Cation Exchange (WCX2 und CM10) in Gegenwart von 100 &mgr; l 50 mmol /l Natriumacetat, 1 g /L Octylglucopyranosid, Proteaseinhibitorcocktail, pH 5; (C) starke Anionenaustausch (SAX2) in Gegenwart von 100 &mgr; l 50 mmol /l TrisHCl, 1 g /L CHAPS, Protease-Inhibitor-Cocktail, pH 8; und (d) Hydrophobe Interaktionschromatographie (H50) in Gegenwart von 100 &mgr; l von 5 ml /l Trifluoressigsäure. Nach dem Waschen mit 100 ul der gleichen entsprechenden Puffer wurde Sinapinsäure hinzugefügt Desorption und Ionisation zu erleichtern. Die Chips wurden durch SELDI-TOF-MS (pBSII, Ciphergen Biosystems Inc.) analysiert. Cancers und Kontrollen wurden verwirbelt und gleichzeitig auf dem gleichen Chip und auf mehreren Chips laufen Chip-zu-Chip-Variation zu minimieren.
Die Magenflüssigkeit Pellets in 25 ul 6 mol /L Guanidinthiocyanat suspendiert wurden, 5 g /l Octyl glucopyranosid, 0,1 mol /L Hepes pH 7 und 100-200 &mgr; l von 9 mol /l Harnstoff, 2 g /L CHAPS, 50 mmol /L Tris-HCl, pH 7,5 bei 4 ° C für 45 Minuten verwirbelt. Nach Zentrifugation bei 20 000 g für 5 Minuten, 10 &mgr; l des Extrakts wurde wie oben beschrieben auf Proteinarrays angewendet. Ein Retentat Karte drucken, bei dem generiert wurde einzelne Proteine als getrennte Peaks auf der Basis ihrer Masse angezeigt wurden Ladungs-Verhältnis . Die Daten der Proteom-Spektren wurden von Ciphergen Express Data Manager Software mit Pattern-Spur und Zwei-Wege-hierarchischen Clustering-Algorithmus analysiert. Ausgerichtete Peaks mit Signal-Rausch-Verhältnis oberhalb von 3 wurden durch Gesamtionenstrom normiert. mit der Bedeutung Analyse von Microarrays (SAM) Software von der Stanford University Proteomic Funktionen wurden weiter analysiert. Das Paket wurde entwickelt, um Probleme spezifisch für Microarray-Datenanalyse (Signal-Rauschverhältnis die Varianz verschieden von Gen zu Gen, eine große Anzahl von Datenpunkten aus einer kleinen Anzahl von Proben) zu adressieren, aber wir fanden es auch anwendbar auf proteomic Datenanalyse zu sein. Der Algorithmus der Software wurde von Tusher et al
beschrieben. [16]. Kurz gesagt definiert es eine Metrik die relative Differenz zum Messen der Differenz zwischen zwei oder mehr Gruppen von Daten anstelle des p
Wert bezeichnet. Es verwendet eine Variation des Verfahrens Bootstrapping und unterteilt wiederholt einen gegebenen Datensatz zufällig in zwei Gruppen (Spektren, die Proteom-Funktionen in dieser Studie enthält) die relative Differenz für jede der Permutationen zu berechnen. Die Anzahl der Permutationen wurde auf 1000 in dieser Studie zu sein, und die Software berechnet 1000 relativen Differenzwerte für jede proteomic Funktion. Die relative Differenz der jeweiligen Gruppierung von Interesse (relative Unterschied beobachtet) wurde zu der durchschnittlichen relativen Differenz aus allen Permutationen verglichen (erwartete relative Differenz) jedes Merkmals und die Funktion wurde oben oder nach unten reguliert werden beurteilt, je nachdem, ob ihre beobachteten relativen Differenz größer oder kleiner als die erwartete relative Differenz von einigen Schwellenwert. Die Software geschätzt, um eine falsche Entdeckung Rate (auch in Bezug definiert [16]) für jeden Schwellenwert, der eine indirekte Mittel vorgesehen, um den Cutoff einzustellen. Die Marker durch dieses Verfahren identifiziert wurden, waren statistisch signifikant. Die falsche Entdeckung Rate eingestellt wurde auf weniger als 0,05 in dieser Studie.
Die Marker von SAM, eine zweite Charge von 53 Proben wurden verblindet für hierarchische Clustering zu dem Datensatz hinzugefügt identifiziert Zur Validierung mit der Ciphergen Express Data Manager-Software. Während die bekannten Proben von SAM verwendet, um die Markierungen zu wählen wurden in der Clustering gut erwartet auszuführen, wurden die Proben blind gemacht, wie gut zu testen, enthalten die Markierungen auf unbekannte Proben verallgemeinern. Die Ergebnisse des Clustering wurden einfach im Vergleich gegen die wahre Identität der Proben und keine erweiterte Klassifizierungsverfahren oder jede andere Software für die Validierung verwendet wurde.
Biomarkeridentifizierung
Magensaft-Proteine wurden fraktioniert durch Anionenaustauschchromatographie (Q HyperD , Ciphergen Biosystems Inc.), schrittweise Änderungen des pH-Wertes für die Elution verwendet. Proteine in der 50 mmol /l TrisHCl, 1 g /L Octylglucopyranosid, pH 8 Eluate wurden zur weiteren Reinigung auf eine Kationenaustauschmatrix (LWCX30) unter Verwendung von 50 mmol /l Natriumacetat, 1 g /L Octylglucopyranosid, pH 5 als Binde- und Waschpuffer. Nach Zugabe von alpha-Cyano-4-Hydroxyzimtsäure energieabsorbierende Moleküle (Ciphergen Biosystems Inc.), die zurückgehaltenen Proteine durch pBSII und Q-TOF (Waters /Micromass), ausgestattet mit einem Proteinchip Interface (PCI 1000, Ciphergen Biosystems Inc.) analysiert wurden, . Die Proteine wurden durch MS /MS-Fragmentierung und zur Identifizierung gekennzeichnet durch Datenbanksuche durchgeführt wurde mit Mascot (Matrix Science Ltd., London, UK).
Biomarker- Validierung
Dies wurde in einem dritten Satz von Magensaft Proben von gutartigen entnommen worden waren Magen-und Magenkrebs-Patienten. Jeweils frisch gesammelte Probe wurde verarbeitet feste Rückstände zu entfernen und um den Proteingehalt zu konzentrieren, wie folgt. Phenylmethansulfonylfluorid bis zu einer Endkonzentration von 0,2 mM wurde die Probe bei 500 g für 15 Minuten zentrifugiert und 4 ° C nach der Zugabe. Protease-Inhibitoren (Complete Mini ™, Roche Applied Science, Indianapolis, IN, USA) wurden zu der durch zentrifugale Membranfiltration bei 2 900 g und 15 ° C (Amicon Ultra-4 Zentrifugalfilter Vorrichtung 5 000 nominalen Molekulargewichtsgrenze gefolgt Überstand gegeben; - 20% seines ursprünglichen Volumens Millipore, Billerica, MA, USA) bis die Probe auf 10 reduziert wurde. Gesamtproteinkonzentration wurde durch die 2-D Quant Kit (Amersham Biosciences, Pisctaway, NJ, USA) bestimmt. Pepsinogen C und alpha-Defensin 1-3-Konzentrationen wurden durch einen Enzym-linked Immunoassay bestimmt (ELISA) unter Verwendung von Kits von Alpco Diagnostics (Salem, NH, USA) und Hycult Biotechnologie B.V. (Uden, Niederlande), respectively. Jede verarbeitete Probe wurde in zweifacher Ausfertigung für Pepsinogen C und Defensin Ebenen unter Verwendung der Lieferanten Protokolle getestet. Die Proben für die Pepsinogen C-Test wurden 120-fach vorverdünnt. Die Konzentrationen von Pepsinogen C und alpha-Defensin 1-3 wurden unter Bezugnahme auf ihre jeweiligen Standardkurven und als ng (Pepsinogen C) oder pg (Defensin) pro Mikrogramm Gesamtmagenflüssigkeit Protein abgeleitet. Helicobacter pylori
Die Anwesenheit von H. pylori
in Magengewebe wurde durch Visualisierung von Mikroorganismen in spiral Histologieschnitten und /oder durch Immunhistochemie identifiziert. Vier micron Gewebeschnitte wurden entwachst in Xylol und abnehmenden Qualitäten von Ethanol. Antigen Retrieval wurde durch Erhitzen in Citratpuffer, pH 6,0. Der primäre Antikörper gegen H. pylori
(Verdünnung 1:50; DAKO A /S, Glostrup, Dänemark) wurde mit dem sekundären Antikörper Polymerverbindung (Envision Chem Mate, DAKO), gefolgt und visualisiert Diaminobenzidin als Chromogen verwendet
. Ergebnis einschränken Mehrere oben oder nach unten reguliert Protein-Biomarkern bei Magenkrebs wurden in der Magenflüssigkeit entdeckt. Eine repräsentative proteomic Karte von Magenflüssigkeit ist in Abbildung 1. Es ist ein Gel Ansicht eines Massenspektrums Magenflüssigkeit Proteine selektiv gebunden an immobilisierte Kupfer (II) Metallion in dem Molekulargewichtsbereich von 1500 Da bis 6000 Da darstellt. Signifikante Proteinmarker herunterreguliert Magenflüssigkeit in Krebs festgestellt (p
< 0,01) sind durch Pfeile angezeigt. Abbildung 1 Expression Differenzkarte von Magensaft auf Kupfer (II) immobilisiert Metall Affinitätsabfang-Proteinchip-Array (IMAC3). Die Pfeile zeigen die Protein-Biomarkern signifikant unterschiedlich in Expressionsniveau zwischen den beiden Gruppen von Proben.
Ein Vertreter Proteom Karte von Magenflüssigkeit Pellet-Extrakt ist in Abbildung 2 Proteine wurden selektiv gebunden an ein Kationenaustausch Array-Oberfläche gezeigt. Signifikante Proteinmarker gefunden bei Magenkrebs Flüssigkeit Pellet (p
< 0,01) nach oben oder nach unten reguliert werden durch Pfeile angezeigt. Abbildung 2 Expression Differenzkarte von Magenflüssigkeit Pellet-Extrakt auf Kationenaustauschproteinchip-Array (WCX2). Pfeile zeigen Proteinbiomarker signifikant unterschiedlich in Expressionsniveau zwischen den beiden Gruppen von Proben
Average CV. (Variationskoeffizient; kumulativer 10-15 großen Magenflüssigkeit Peaks pro Spektrum, n = 8) für immobilisierte Kupfer (II) Proteinchip-Array (IMAC3) betrug 12,8%, für die Kationenaustauschmatrix (WCX2) betrug 15%, für die Anionenaustausch-array (SAX2) betrug 17,3% und für die hydrophobe Interaktionschromatographie-Chip (H50) betrug 13,6%. Diese CV-Werte stehen im Einklang mit der Reproduzierbarkeit Beurteilung in der SELDI Literatur [17, 18]. Mit dem SAM Analyse aller Proteom-Funktionen (Gesamtzahl der Funktionen 41 800, die durchschnittliche Anzahl pro Retentat Karte 314) in der Magenflüssigkeit und Pellet-Extrakt
, 46 Proteom Merkmale wurden deutlich nach unten reguliert werden bei Magenkrebs und 60 Proteom-Features waren signifikant hochreguliert bei Magenkrebs gefunden. (Daten aus unterschiedlichen Bedingungen, beispielsweise Flüssigkeit und Pellet sowie verschiedenen Oberflächen, wurden einfach aggregiert zusammen als besondere Merkmale für SAM. Marker von SAM in beiden Pellet und Überstand-Fraktionen gemeldet wurden manuell identifiziert und repräsentiert nur einmal in der Liste, nachdem sie waren biologisch signifikant) angesehen. Deutlich nach unten reguliert Marker enthalten 1884, 2428, 2594, 2840, 4050, 11720, 13700 Da; signifikant hochreguliert Marker enthalten 1761, 1831, 3372, 3443, 3605, 5160, 6780 Da. (Die meisten der bedeutenden Marker wurden auf WCX2 entdeckt und IMAC-Kupfer (II), durch SAX2 gefolgt). Basierend auf den 106 signifikant unterschiedliche Proteom-Features (Weitere Datei 1), Zwei-Wege-hierarchischen Clustering-Analyse (zweidimensionale vollständige Bindung) durchgeführt wurde. Die meisten der Magenkrebsfälle wurden zusammen gruppiert, um einen unverwechselbaren Gruppe zu bilden (Abbildung 3 und zusätzliche Datei 2). Hauptkomponentenanalyse der gleichen Daten zeigten auch, dass Krebs und gutartigen Proben konnten auch in zwei Gruppen unterteilt werden, mit 2 falsch negative Ergebnisse (die doppelte Analyse derselben Fall) und 9 Fehlalarme, bzw. (Abbildung 4). Eine Magenkrebs Flüssigkeitsprobe (von einem Fall der Stufe I schlecht differenzierten Adenokarzinom des Magens) gruppierten unter Nicht-Krebsproben; alle anderen 4 frühen Stadium (Stadium 0 und I) Patienten korrekt mit Proben von 14 Patienten mit Stadium II gruppierten - IV Magenkrebs, eine Gesamt diagnostische Sensitivität von 95% (18/19 Patienten mit Magenkrebs) auf dem Trainingssatz gibt. Abbildung 3: Expression Differenzkarte von Magenflüssigkeit und Pellet-Extrakt Proteine Trainingssatz Proben auf vier Proteinchip Arrays angezeigt in Zwei-Wege-hierarchischen Clustering. Signifikante Proteom Merkmale sind vertikal angezeigt. Die Intensität der Graustufen gibt den Grad der relativen Proteinebene, höher oder niedriger ist als der Medianwert. Patientenfälle sind horizontal dargestellt; die meisten Magenkrebs-Patienten sind eng miteinander gebündelt. Diese Abbildung zeigt den oberen Quartil des Vollbildes (bitte Weitere Datei 2 für das Vollbild zu sehen). 4 Hauptkomponentenanalyse Grundstück von Proteom-Funktionen von Trainingssatz Proben
Abbildung. Eine einzelne Ebene (durch die schwarze Linie bezeichnet) teilt die Proben in zwei Gruppen mit 1 falsch negativ (in zweifacher Ausfertigung Flecken gezeigt) und 9 Fehlalarme.
Neun von 36 Nicht-Krebs-Proben im Trainingssatz mit den Krebsproben gruppierten (Spezifität 75%). Von diesen hatten 1 eine dysplastische adenomatöse Polyp - eine Präkanzerose [19]. Unter den anderen 8 Patienten hatten 6 klinisch Biopsien gerichtet, die intestinale Metaplasie bei 4 Patienten ergab (67%). Acht Nicht-Krebs-Patienten, deren Magenflüssigkeit Proteinprofile in der normalen Gruppe geclustert waren auch klinisch Schleimhautbiopsien gerichtet, die nur 2 Patienten in intestinalen Metaplasie zeigten (25%). Eine Überprüfung von 1000 aufeinander folgenden Magen für alle Indikationen durchgeführt Biopsien zeigten eine allgemeine Prävalenz der intestinalen Metaplasie im Singapore General Hospital während der Studiendauer von 30%. Dies steht im Gegensatz mit der Prävalenz von mindestens 67% der intestinale Metaplasie unter klinisch gutartigen Fällen deren Proteom Profile enger mit Magenkrebs Fällen gruppierten als bei anderen Normalen, im Einklang mit intestinaler Metaplasie ein Zwischenzustand im Übergang von normalen Magen-Epithel zu einem Adenokarzinom des Magens zu sein . Die genaue Identifizierung der intestinalen Metaplasie durch Endoskopie ist bekannt, ungenau zu sein [20]. So kann ein Magenkrebs-Typ Proteom-Fingerabdruck ist möglicherweise ein empfindlicher Indikator für das Vorhandensein dieser prämalignen Läsion bei Patienten klinisch als mit gutartigen Magenerkrankungen diagnostiziert. Magenkrebspatienten in der Ausbildung
eingestellt waren signifikant älter (Durchschnittsalter 67,7 Jahre) als Patienten mit gutartigen Magenbedingungen (Alter 56,6) (p = 0,0062
) bedeuten. Um die Möglichkeit zu begegnen, dass die Proteinprofile wurden nach Alter oder ethnischer Zugehörigkeit im Zusammenhang, wir Daten von der Untergruppe der chinesischen Patienten über 55 Jahren erneut analysiert. Dies führte zu 1/17 Krebs fehlklassifiziert (das gleiche Tumor, der jedoch nicht korrekt angezeigt wurde, wenn alle 19 Krebsarten untersucht wurden; Sensitivität 94%) und 17.4 Kontrollen fehlklassifiziert (die gleichen 4 steuert, die unter den 9 fehlklassifiziert gutartigen Fällen waren; Spezifität 76,5%) .
getestet Wir als nächstes die tatsächliche Leistung der Proteom-Profile bei der Unterscheidung zwischen Krebs und gutartigen Proben in einer zweiten Serie von 53 verblendet Magenflüssigkeit und Pellet-Extraktproben (24 Magenkarzinome und 29 gutartige Magenerkrankungen) (Weitere Datei 3). Einundzwanzig von 24 Magenkarzinome korrekt identifiziert wurden (Sensitivität 88%) und 2 von 29 gutartigen Proben wurden falsch klassifiziert (Spezifität 93%) (5). Abbildung 5 Expression Differenzkarte von Magenflüssigkeit und Pellet-Extrakt Proteine Validierungsset Proben auf vier Proteinchip Arrays angezeigt in Zwei-Wege-hierarchischen Clustering. Signifikante Proteom Merkmale sind horizontal angezeigt. Die Intensität der roten oder grünen Farben gibt den Grad der relativen Proteinebene, höher oder niedriger als der Medianwert. Patientenfälle sind vertikal präsentiert; die meisten Patienten mit Magenkrebs fest zusammen gruppiert sind.
Proteom-Marker Ausgewählte (basierend auf Bedeutung von SAM bestimmt Score) waren halb gereinigter auf Proteinchip Arrays und direkt auf Flecken durch stoßinduzierte Dissoziation Sequenzierung (Abbildung 6) identifiziert. Mehrere der deutlich herunterreguliert Markern bei Krebspatienten in den 1 und 2, 1881,9 Da, 2041,0 Da, 2188,1 Da und 2387,3 Da, wurden identifiziert Pepsinogen C zu sein und eine Aktivierungspeptidfragmente (Tabelle 1) Pepsin. Die hochreguliert Triplett Marker bei Krebspatienten in den 2, 7 und Zusatzdatei 4 wurden identifiziert alpha-Defensin-1,2,3 sein. Intensity Streudiagramme zeigen hoch signifikante Unterschiede in den mittleren Intensitäten von Defensin und Pepsin-Fragment zwischen gutartigen Kontrolle und Magenkrebs Flüssigkeitsproben (p = 0,003
und 0,00002, beziehungsweise) (Abbildung 8). Mit ELISA spezifisch für Pepsinogen C bestätigten wir deutlich niedrigere Konzentrationen bei Magenkrebs Flüssigkeiten (11,9 ± 0,1 ng /ug Gesamtprotein; Mittelwert ± SEM n = 6) im Vergleich zu gutartigen Proben (21,5 ± 1,4 ng /ug Gesamtprotein n =. 23) in einer dritten Probe Satz (p = 0,0126
; Student ungepaarten two-tailed t
Test). ELISA auf demselben Probensatz für Defensin Ebenen zeigten höhere Konzentrationen bei Magenkrebs Proben (63,4 ± 9,2 pg /ug Gesamtprotein; Mittelwert ± SEM n = 6) als bei gutartigen Proben (46,2 pg /ug Gesamtprotein; Mittelwert ± SEM n = 23) ((p = 0,0654
; Student t
Test) .Tabelle 1 Peptidsequenzen von MS //MS identifiziert
Peptid m /z
Sequence
Protein Spiel
Mowse † Score
Mowse mit signifikanter Homologie Score
2.386,29
FLKKHNLNPARKYFPQWKA
Pepsin A 35
Aktivierungspeptid
> 28
2.187,12
FLKKHNLNPARKYFPQW
Pepsin A 18
Aktivierungspeptid
> 26
2.040,03
LKKHNLNPARKYFPQW
Pepsin A 28
Aktivierungspeptid
> 26
1.775,95
FLKKHNLNPARKYF
Pepsin A 47
Aktivierungspeptid
> 26
1.628,84
LKKHNLNPARKYF
Pepsin A 40
Aktivierungspeptid
> 28
1.880,92
LRTHKYDPAWKYRF
Pepsinogen C Aktivierungspeptid
31
> 22
† Mowse score = -10Log ( P), wobei P = Wahrscheinlichkeit, dass das Spiel ein zufälliges Ereignis ist (P
< 0,05) m /z
, Masse /
6 Hochauflösendes Massenspektrum von fraktionierte Magenflüssigkeit Proteine auf LWCX30 Proteinchip-Array auf einem mit einer PCI1000 Schnittstelle ausgestattet QTOF erhalten aufzuladen. Boxed Peaks wurden zu einer Fragmentierung Analyse durch stoßinduzierte Dissoziation MS /MS unterzogen.
7 Hochauflösendes Massenspektrum von Magenflüssigkeit Proteine auf H50 Proteinchip-Array auf einem mit einer PCI1000 Schnittstelle ausgestattet QTOF erhalten Abbildung. Diese Abbildung zeigt die hochreguliert Triplett Marker bei Magenkrebs. Bitte beachten Sie Weitere Datei 4 für das Gesamtbild.
Abbildung 8 Streudiagramme von Intensitätswerten von Defensin und Pepsin-Fragment in der Magenflüssigkeit Proben von gutartigen Kontrolle und Magenkrebs-Patienten aus dem Trainingssatz.
Diskussion
Unsere Daten deuten darauf hin, dass die spektrale Profil von unfraktioniertem Magenflüssigkeit eine sinnvolle Ergänzung für die Krebsdiagnose und Erkennung von frühen Stadium der Erkrankung sein könnte, wenn sie mit klinischen Gastroskopie kombiniert. Neue Versuche zur Protein-Biomarkern für Magenkrebs identifiziert haben untersucht, Serum [21-29] und Gewebe [24, 30-37] und zunehmend Massenspektrometrie eingesetzt. Ältere Berichte von serologischen Assays einzelner bekannten Tumormarkern z.B. CEA, CA 19-9, CA 72-4, CA242 und TAG-72, haben im allgemeinen eine geringe Empfindlichkeit (< 50%) [38-41]. Darüber hinaus gibt es erhebliche Quer Positivität dieser Tumormarker in nicht-Magenkarzinome z.B. angehoben CEA und MG7-Ag Ebenen sind bei Kolorektalkarzinomen, cholangiocarcinoma, Pankreaskarzinom häufig, und auch bei gesunden Kontrollen [40, 23]. Nicht überraschend, wie Serumtumormarker haben keine etablierte Rolle bei Magenkrebs Diagnose und Screening, obwohl sie dienen als prognostische Indikatoren und frühe Marker rezidivierender Erkrankung nach Gastrektomie [39, 41, 42].
Wir haben uns für Proteom-Profile zu untersuchen von Magenflüssigkeit für Krankheit Biomarker, weil es schien wahrscheinlich, dass Magen-Protein-Sekretion in malignen und prämalignen Zuständen, verbunden mit der möglichen Anwesenheit von abgeblätterten Krebszellen gestört, unverwechselbare Proteom Profile erzeugen könnte. Wie bei der Suche nach Serum-Biomarkern haben mehrere Gruppen den diagnostischen Nutzen von bekannten Tumormarkern im Magensaft untersucht. Weder CEA noch CA 19-9 Positivität in der Magenflüssigkeit hat diagnostische Genauigkeit nachgewiesen [43-46]. Alpha-1-Antitrypsin im Magensaft wurde vor kurzem als Magenkrebs Biomarker berichtet [47, 48].
Unser Ansatz eine empfindliche Methode für Magenkrebs Diagnose aus früheren Studien auf drei Arten unterschieden sich zu entwickeln. Zunächst haben wir uns für eine biologische Probe, die organspezifische war (das heißt endoskopisch aspiriert Magenflüssigkeit) anstelle einer systemischen (das heißt Serum), logisches Denken, dass die molekularen Merkmale eher krankheitsspezifische wäre. Zweitens ermöglicht Massenspektrometrie uns eine unvoreingenommene Discovery-basierte Ansatz. Drittens unsere Daten generierte Profile mehrerer Proteom Marker, die in zunehmendem Maße gelten als höhere Sensitivität und Spezifität als einzelne Tumormarker [49, 50]. Für Magenkrebs, die Kombination von sogar 2 oder 3 Tumormarker erreicht bessere diagnostische Genauigkeit im Vergleich zu einem einzelnen Marker allein [38, 40].
Protein Fingerabdrücke von Magenflüssigkeit von Magenkrebs-Patienten zeigten insgesamt 106 Proteom Features, die deutlich nach oben waren - oder herunterreguliert (Zusätzliche Dateien 1 und 3). Zwei prominente Marker wurden zur Identifizierung von MS /MS ausgewählt. Pepsin A und Pepsinogen C-Aktivierung Peptide wurden herunterreguliert in Magenflüssigkeiten aus Mägen mit histologisch bestätigten Adenokarzinome entfernt. Eine Untersuchung von Gefrierschnitten von Magenkrebs hat auch berichtet signifikante Herabregulierung von Pepsinogen C, identifiziert durch MS /MS, in Tumorgewebe [51].
 Hängt Ihr tägliches Wohlbefinden von Ihrem Magen ab? Sie sind nicht allein!
Hängt Ihr tägliches Wohlbefinden von Ihrem Magen ab? Sie sind nicht allein!
 Länder mit älterer Bevölkerung haben höhere SARS-CoV-2-Infektionen und -Todesfälle,
Länder mit älterer Bevölkerung haben höhere SARS-CoV-2-Infektionen und -Todesfälle,
 Zika wird möglicherweise unter Tränen und Schweiß weitergegeben:Bericht
Zika wird möglicherweise unter Tränen und Schweiß weitergegeben:Bericht
 Melanosis Coli (Pseudomelanosis Coli)
Melanosis Coli (Pseudomelanosis Coli)
 Die 10 besten Rezepte mit niedrigem FODMAP-Gehalt
Die 10 besten Rezepte mit niedrigem FODMAP-Gehalt
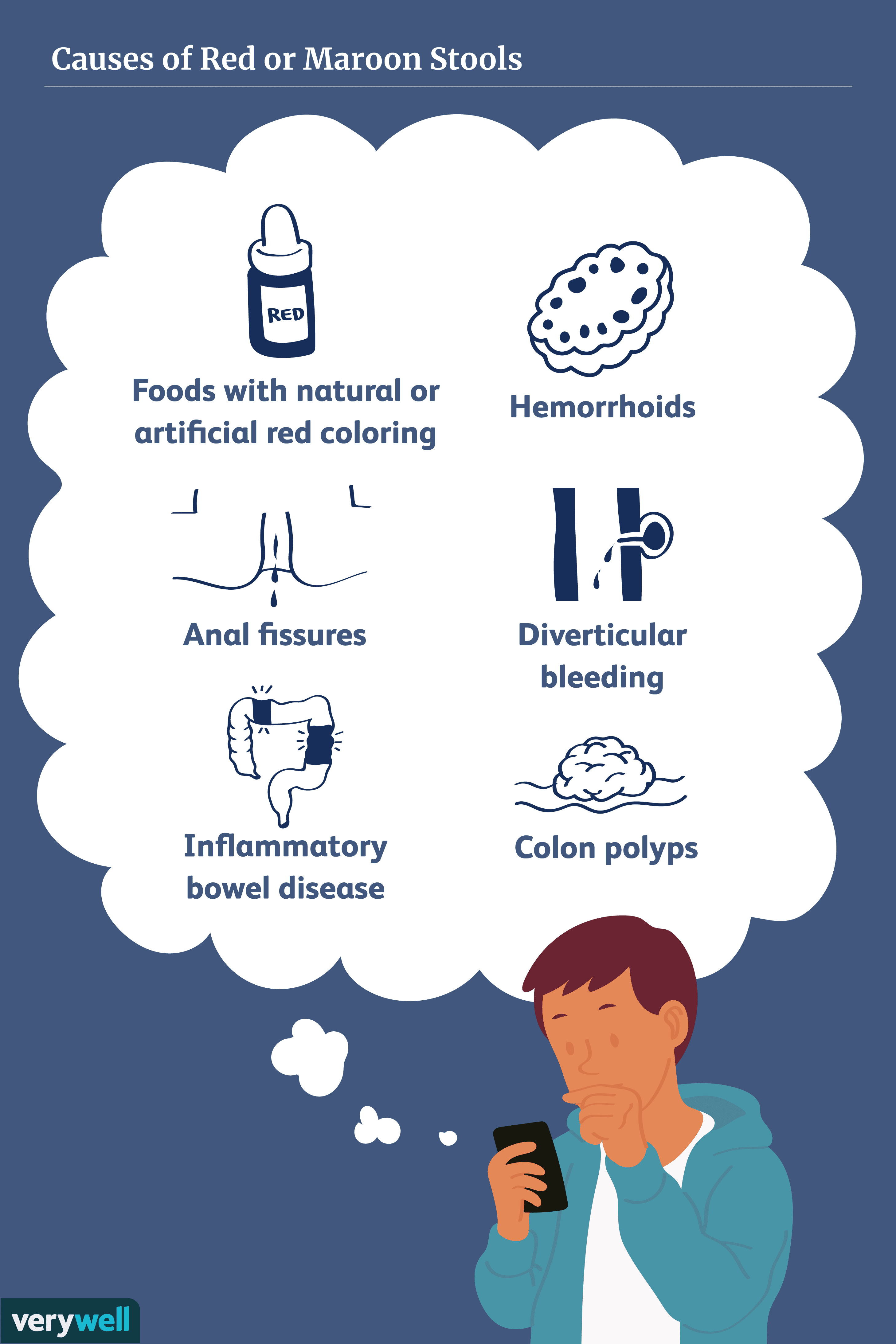 Was verursacht roten oder kastanienbraunen Stuhl?
Was verursacht roten oder kastanienbraunen Stuhl?
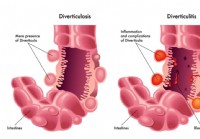 Divertikelkrankheit
Divertikulose ist definiert als Ausstülpung der Schleimhaut und Submukosa durch die Muscularis propria (falsches Divertikel, da nur 2 Schichten betroffen sind). Dies ist eine sehr häufige Erkrankung i
Divertikelkrankheit
Divertikulose ist definiert als Ausstülpung der Schleimhaut und Submukosa durch die Muscularis propria (falsches Divertikel, da nur 2 Schichten betroffen sind). Dies ist eine sehr häufige Erkrankung i
 Gesündeste Darmbakterien bei pflanzlicher oder mediterraner Ernährung
Eine neue Studie zeigt, dass bestimmte Lebensmittel, die entweder durch eine pflanzliche oder eine mediterrane Ernährung zugeführt werden, den Darm vor entzündlichen Erkrankungen schützen können. durc
Gesündeste Darmbakterien bei pflanzlicher oder mediterraner Ernährung
Eine neue Studie zeigt, dass bestimmte Lebensmittel, die entweder durch eine pflanzliche oder eine mediterrane Ernährung zugeführt werden, den Darm vor entzündlichen Erkrankungen schützen können. durc
 Gesundheitstipp:Anzeichen von Gallensteinen
Neueste Verdauungsnachrichten Wenn eine Nierentransplantation fehlschlägt, ist es am besten, es noch einmal zu versuchen Lassen Sie sich Ihr Weihnachtsfest nicht von Sodbrennen ruinieren Nachfrage na
Gesundheitstipp:Anzeichen von Gallensteinen
Neueste Verdauungsnachrichten Wenn eine Nierentransplantation fehlschlägt, ist es am besten, es noch einmal zu versuchen Lassen Sie sich Ihr Weihnachtsfest nicht von Sodbrennen ruinieren Nachfrage na