Para el estudio, publicado el 26 de mayo en Celda , investigadores internacionales recogieron casi 5, 000 muestras durante un período de tres años en 60 ciudades de 32 países y seis continentes. Los investigadores analizaron las muestras utilizando una técnica de secuenciación genómica llamada secuenciación de escopeta para detectar la presencia de varios microbios. incluyendo bacterias, arqueas (organismos unicelulares distintos de las bacterias), y virus que utilizan el ADN como material genético. (Otros tipos de virus que utilizan ARN como material genético, como el SARS-CoV-2, el virus que causa COVID-19, no se habría detectado con los métodos de análisis de ADN utilizados en este estudio prepandémico).
Este campo de investigación tiene importantes implicaciones para detectar brotes de infecciones conocidas y desconocidas y para estudiar la prevalencia de microbios resistentes a los antibióticos en diferentes entornos urbanos.
"Cada vez que te sientas en el metro, es probable que esté viajando con una especie completamente nueva, "dijo el autor principal, el Dr. Christopher Mason, codirector de la Iniciativa WorldQuant para la Predicción Cuantitativa y profesor de fisiología y biofísica en Weill Cornell Medicine. El Dr. Mason también es cofundador y consultor remunerado de Biotia y Onegevity Health, y un orador pagado para WorldQuant LLC.
El estudio actual condujo al descubrimiento de 10, 928 virus y 748 bacterias que no están presentes en ninguna base de datos de referencia.
El Dr. Mason fundó MetaSUB (abreviatura de Metagenomics and Metadesign of Subways and Urban Biomes) en 2015, junto con el Dr. Evan Afshin, quien entonces era estudiante de pregrado en Macaulay Honors College en Queens College y ahora es investigador clínico en fisiología y biofísica en Weill Cornell Medicine y consultor pagado para Onegevity Health.
El estudio recientemente publicado fue dirigido por los Dres. Masón, David Danko, un estudiante de doctorado de Weill Cornell Graduate School en el laboratorio del Dr. Mason durante el estudio, y Daniela Bezdan, quien era investigador asociado en biomedicina computacional en Weill Cornell Medicine en ese momento.
Al recolectar muestras de microbios y analizar sus genes, conocidos colectivamente como microbioma, los investigadores esperan aprender más sobre las bacterias. virus y otros microorganismos que viven entre los humanos. Por ejemplo, la investigación puede ayudar a identificar la aparición de cepas resistentes a los antibióticos.
Predecir la resistencia a los antibióticos solo a partir de secuencias genéticas es un desafío, pero los investigadores pudieron mapear algunos genes que se sabe que están relacionados con la resistencia, cuantificar su abundancia y confirmar la capacidad de los marcadores genéticos para conferir resistencia. Descubrieron que algunas ciudades tenían más genes de resistencia que otras, y que podría haber firmas específicas de la ciudad para algunos de estos genes.
La resistencia a los antimicrobianos sigue siendo un importante desafío para la salud mundial. "Si bien se necesitan más investigaciones, este conjunto de datos demuestra el valor y el potencial del mapeo y monitoreo del microbioma, y la información que puede proporcionar a los médicos, científicos y funcionarios de salud pública, "Dijo el Dr. Afshin.
Es más, conocer las pequeñas moléculas y proteínas producidas por microbios también podría conducir al descubrimiento de nuevos antibióticos, así como de otras moléculas que tienen el potencial de desarrollarse como fármacos. Muchos antibióticos y fármacos que se utilizan actualmente se han derivado de fuentes microbianas.
Los descubrimientos realizados sobre nuevas especies microbianas también podrían conducir a nuevas herramientas y enfoques de laboratorio. como formas novedosas de utilizar la herramienta de edición molecular conocida como CRISPR. En este estudio, los investigadores encontraron 838, 532 matrices CRISPR novedosas (fragmentos de ADN viral que se encuentran dentro de las bacterias) y 4,3 millones de péptidos nuevos (proteínas pequeñas).
Debido a estos esfuerzos de muestreo, El Dr. Mason dijo que puede predecir con un 90 por ciento de precisión dónde vive una persona, simplemente secuenciando el ADN en sus zapatos. Se encontró que muchos factores influyen en el microbioma de una ciudad, incluida la población general y la densidad de población, elevación, proximidad al océano y al clima. Los hallazgos sobre estas firmas distintas podrían permitir futuros estudios forenses.
Un microbioma contiene ecos moleculares del lugar donde fue recolectado. Una muestra costera puede contener microbios amantes de la sal, mientras que una muestra de una ciudad densamente poblada puede mostrar una biodiversidad sorprendente, "Dijo el Dr. Danko.
Dr. David Danko, Estudiante Soctoral, Escuela de Graduados Weill Cornell.
Drs. Mason y Afshin comenzaron a recolectar y analizar muestras microbianas en el sistema de metro de la ciudad de Nueva York en 2013. Después de publicar sus primeros hallazgos, apodado PathoMap, fueron contactados por investigadores de todo el mundo que querían hacer estudios similares para sus propias ciudades.
El interés internacional inspiró al laboratorio del Dr. Mason a crear MetaSUB y reclutó a Daniela Bezdan como directora de investigación. "Necesitábamos protocolos aceptados internacionalmente, convenios de logística y colaboración con científicos, vendedores, oficinas gubernamentales y fundaciones filantrópicas para potencialmente 100 ciudades en 20 países, ", Dijo Bezdan.
Hoy, MetaSUB continúa creciendo y se ha expandido para recolectar muestras de ARN y ADN del aire, agua y alcantarillado, además de superficies duras. Esto ha llevado a una subvención de $ 5 millones para secuenciación de aguas residuales y rastreo viral en tres estados (Florida, Nueva York y Wisconsin), y que es parte del nuevo Sistema Nacional de Vigilancia de Aguas Residuales (NWSS) del Centro para el Control y la Prevención de Enfermedades.
El grupo también supervisa proyectos como Global City Sampling Day (gCSD), celebrado todos los años el 21 de junio, y ha realizado estudios de amplio alcance, incluido un análisis microbiano integral de Río de Janeiro antes, durante y después de los Juegos Olímpicos de Verano de 2016. Muchas de las muestras analizadas en el estudio actual se recolectaron en el Día Global de Muestreo de Ciudades en 2016 y 2017.
El esfuerzo de muestreo de la ciudad de Nueva York se realizó con el apoyo del Centro de Ciencias Clínicas y Traslacionales de Medicina Weill Cornell (CTSC), en colaboración con el gerente senior del programa CTSC, Jeff Zhu. El Dr. Mason y sus colegas se están preparando actualmente para el evento de este año.
"Cuando comenzamos en 2015, el consorcio estaba formado por 16 ciudades; seis años después tenemos más de 100 ciudades. Es genial tener este grupo de curiosos co-investigadores entusiastas y emprendedores, "dijo el Dr. Mason, quien también es profesor de genómica computacional en biomedicina computacional en el Instituto SAR el Príncipe Alwaleed Bin Talal Bin Abdulaziz Al-Saud de Biomedicina Computacional en Weill Cornell Medicine.
"Aunque las muestras se recolectan en todo el mundo, gran parte del análisis se realiza aquí mismo, en la ciudad de Nueva York, en Weill Cornell Medicine, "dijo el Dr. Mason. El análisis y ensamblaje de secuencias también aprovechó Bridges y Bridges-2, Supercomputadoras Extreme Science and Engineering Discovery Environment (XSEDE) en el Pittsburgh Supercomputing Center.
Los investigadores de MetaSUB en Suiza (los doctores Andre Kahles y Gunnar Rätsch) utilizaron estos conjuntos y datos brutos para crear un portal de secuencias de ADN global (MetaGraph) que indexaba todas las secuencias genéticas conocidas (incluidos los datos de MetaSUB). El portal mapea cualquier elemento genético conocido o recién descubierto a su ubicación en la Tierra y puede ayudar en el descubrimiento de nuevas interacciones microbianas y funciones putativas.
El aislamiento de ADN de las muestras se realizó en gran parte con el apoyo de Zymo Research y Promega, y secuenciado en colaboración con el Dr. Shawn Levy en el Instituto HudsonAlpha de Biotecnología, Dr. Klas Udekwu de la Universidad de Estocolmo y el Centro del Genoma de Nueva York. Los estudios futuros y en curso analizarán el ARN y el ADN con lecturas largas y métodos de imágenes espaciales, así como rastrear los metabolitos de los sitios globales, y continuar actualizando el mapa genético a escala planetaria.
 ¿Cuánto tiempo lleva recuperarse de un procedimiento de gastroyeyunostomía?
¿Cuánto tiempo lleva recuperarse de un procedimiento de gastroyeyunostomía?
 Cómo se realiza una biopsia de colon
Cómo se realiza una biopsia de colon
 Una variante genética común explica por qué la inmunoterapia a menudo falla en la enfermedad de Crohn
Una variante genética común explica por qué la inmunoterapia a menudo falla en la enfermedad de Crohn
 Por qué los síntomas del SII pueden empeorar durante su período
Por qué los síntomas del SII pueden empeorar durante su período
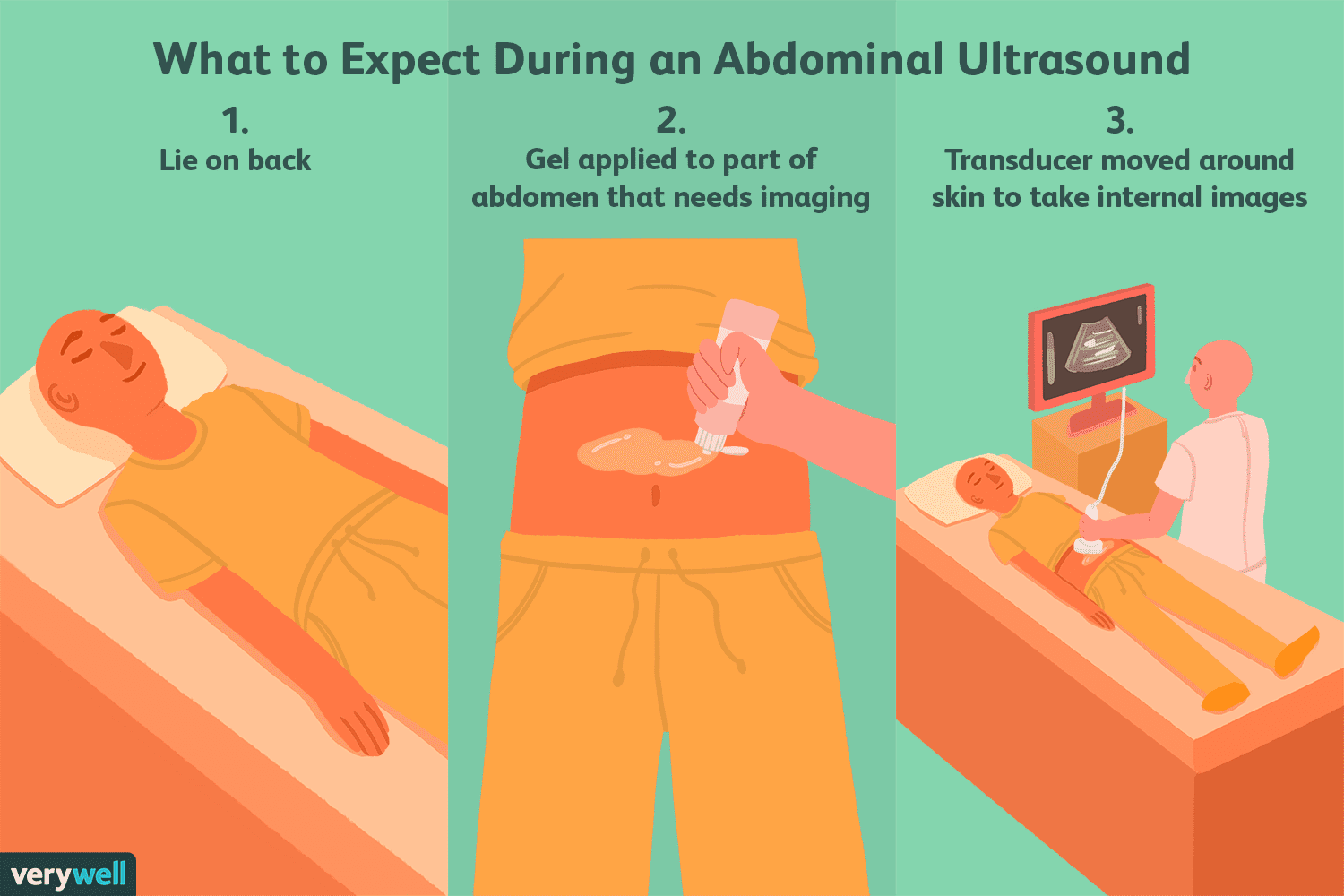 ¿Qué es una ecografía abdominal?
¿Qué es una ecografía abdominal?
 ¿Qué es la sigmoidoscopia flexible?
¿Qué es la sigmoidoscopia flexible?
 Registros de interés del Programa de Coaching SIBO
Inscripciones de interés para el programa de entrenamiento SIBO ahora abiertas Mencioné la semana pasada que tenía algunas noticias emocionantes para compartir con ustedes. Estoy encantado de anuncia
Registros de interés del Programa de Coaching SIBO
Inscripciones de interés para el programa de entrenamiento SIBO ahora abiertas Mencioné la semana pasada que tenía algunas noticias emocionantes para compartir con ustedes. Estoy encantado de anuncia
 Nutrición personalizada
Gut Microbiota for Health Conference Update 2019 - Post Series 4 Estamos en la semana 3 de la actualización de la conferencia Gut Microbiota for Health:verá 5 artículos diferentes sobre la conferenc
Nutrición personalizada
Gut Microbiota for Health Conference Update 2019 - Post Series 4 Estamos en la semana 3 de la actualización de la conferencia Gut Microbiota for Health:verá 5 artículos diferentes sobre la conferenc
 Qué causa la psoriasis (y cómo detenerla naturalmente)
Para aquellos a quienes se les ha diagnosticado una enfermedad autoinmune como la psoriasis, supongo que están contentos de tener finalmente una etiqueta para esas manchas rojas que pican en la piel.
Qué causa la psoriasis (y cómo detenerla naturalmente)
Para aquellos a quienes se les ha diagnosticado una enfermedad autoinmune como la psoriasis, supongo que están contentos de tener finalmente una etiqueta para esas manchas rojas que pican en la piel.