I nostri risultati iniziali hanno superato le nostre più rosee aspettative. Come test abbiamo selezionato una sfida computazionale che abbiamo precedentemente misurato come richiedere circa 900 ore di tempo utilizzando CPU di classe server, o circa 13, 000 ore core della CPU. Abbiamo scoperto che potrebbe essere completato in sole 8 ore su una singola GPU NVIDIA Tesla V100, o circa 30 minuti se si utilizzano 16 GPU, che potrebbe ridurre i tempi di analisi di diversi ordini di grandezza. Una NVIDIA RTX 2080TI di classe workstation lo completerebbe in circa 12 ore."
Igor Sfiligoi, Il principale sviluppatore di software scientifico di SDSC per l'elaborazione ad alto rendimento
"Il nuovo eseguibile sarà anche di enorme valore per il lavoro esplorativo, poiché il set di dati EMP di dimensioni moderate che richiedeva 13 ore su una CPU di classe server ora può essere eseguito in poco più di un'ora su un laptop contenente una GPU mobile NVIDIA GTX 1050, "aggiunse Sfiligoi.
Sfiligoi ha collaborato con Rob Knight, direttore fondatore del Center for Microbiome Innovation, e professore di Pediatria, Bioingegneria e Computer Science &Engineering presso l'università, e Daniele McDonald, direttore scientifico dell'American Gut Project. I microbiomi sono il materiale genetico combinato dei microrganismi in un particolare ambiente, compreso il corpo umano.
"Questo lavoro non è iniziato inizialmente come parte della risposta COVID-19, " disse Sfiligoi. "Abbiamo iniziato la discussione su una simile accelerazione molto prima, ma UniFrac è una parte essenziale della pipeline di ricerca COVID-19."
UniFrac confronta i microbiomi tra loro utilizzando un albero evolutivo che mette in relazione le sequenze di DNA tra loro. "UniFrac ha giocato un ruolo chiave nel Progetto Microbioma Umano, permettendoci di capire come i microbi sono correlati attraverso i nostri corpi, e nel Progetto Microbioma Terra, permettendoci di capire come sono collegati i microbi in tutto il nostro pianeta, " ha detto Knight. "Lo stiamo usando per capire come il microbioma di una persona potrebbe renderla più o meno suscettibile al COVID-19, e quali microbi in ambienti che vanno dalle strutture sanitarie alle acque reflue agli spruzzi oceanici rendono l'ambiente più o meno ospitale per SARS-CoV-2, il coronavirus che causa il COVID-19".
Knight ha notato che Sfiligoi aveva velocizzato l'ultima versione dell'algoritmo, pubblicato meno di due anni fa in Metodi della natura , che di per sé rappresentava già un notevole miglioramento della velocità rispetto alle precedenti implementazioni.
"Man mano che i dati sulla sequenza microbica aumentano in modo esponenziale, da decine di sequenze a miliardi, dobbiamo re-implementare tutti gli algoritmi, " ha affermato. "Questo ultimo passo mostra davvero come l'ottimizzazione dell'infrastruttura di ricerca possa ridurre drasticamente il tempo per il risultato preservando l'accuratezza dei risultati e consentendo di porre domande su scale completamente nuove".
Nello specifico, Sfiligoi ha utilizzato OpenACC, un utente guidato, modello di programmazione parallela basato su direttiva per portare l'implementazione esistente di Striped UniFrac sulle GPU perché ciò consente un'unica base di codice sia per il codice della CPU che per quello della GPU. Un'ulteriore velocità è stata ottenuta sfruttando attentamente la località della cache. È stato inoltre esplorato l'uso della matematica in virgola mobile di precisione inferiore per sfruttare in modo efficace le GPU di livello consumer che si trovano tipicamente nei computer desktop e laptop.
UniFrac è stato originariamente progettato e sempre implementato utilizzando la matematica in virgola mobile di maggiore precisione, spesso chiamato percorso del codice fp64. La matematica in virgola mobile di precisione più elevata è stata utilizzata per massimizzare l'affidabilità dei risultati. Dopo aver implementato la matematica in virgola mobile a precisione inferiore, solitamente chiamato percorso del codice fp32, i ricercatori hanno osservato risultati quasi identici, ma con tempi di calcolo significativamente più brevi.
"Abbiamo riscontrato un'accelerazione di 3 volte nel percorso del codice fp32 per le GPU di gioco come la 2080 Ti e la 1050 mobile, e riteniamo che la precisione dovrebbe essere adeguata per la stragrande maggioranza degli studi, " ha spiegato Sfiligoi.
Inoltre, le modifiche al codice introdotte per accelerare il calcolo della GPU hanno anche accelerato notevolmente l'esecuzione sulle risorse della CPU. La sfida computazionale sopra menzionata può ora essere completata in circa 200 ore sulla stessa CPU di classe server, un'accelerazione 4x, secondo i ricercatori.
"Rendere disponibile il calcolo su dispositivi personali abilitati per GPU, anche laptop, elimina una grande barriera all'interno dell'infrastruttura delle risorse per molti scienziati, " disse Sfiligoi.
 Stile di vita, microbioma e sindrome dell'ovaio policistico
Stile di vita, microbioma e sindrome dell'ovaio policistico
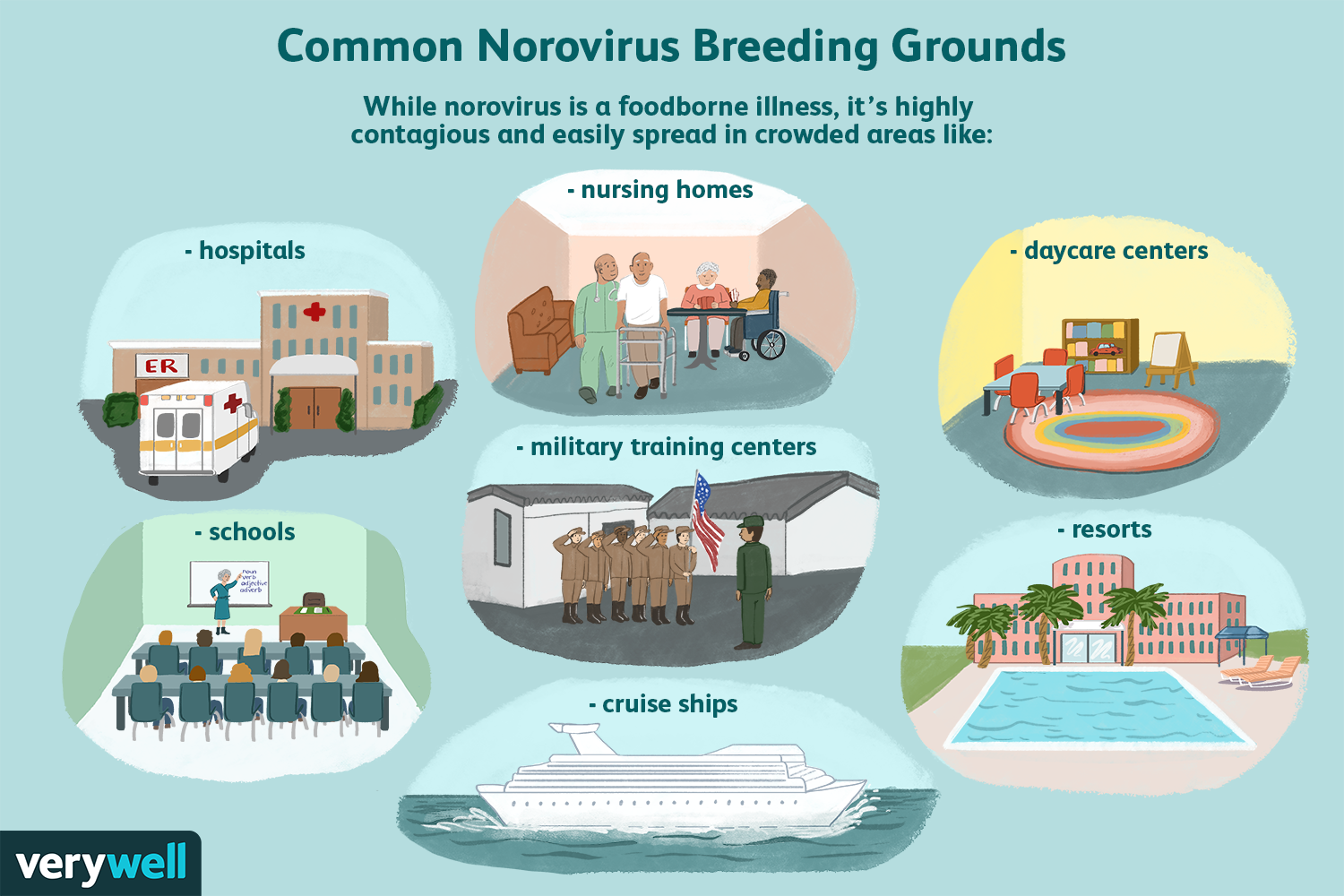 Che cos'è il norovirus?
Che cos'è il norovirus?
 Miglior rilevatore a ultrasuoni di gravidanze ectopiche pericolose, risultati di studio
Miglior rilevatore a ultrasuoni di gravidanze ectopiche pericolose, risultati di studio
 5 cose che ho imparato di nuovo al WAPF 2012
5 cose che ho imparato di nuovo al WAPF 2012
 5 motivi per investire subito nella salute dell'intestino
5 motivi per investire subito nella salute dell'intestino
 test su strada di un servizio di consegna a domicilio sibo
test su strada di un servizio di consegna a domicilio sibo
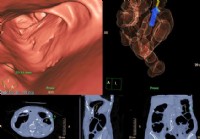 Colonscopia virtuale
Cosè una colonscopia? La colonscopia virtuale è una tecnica che utilizza una TAC per costruire immagini virtuali del colon. La colonscopia è una procedura che consente a un esaminatore (di solito u
Colonscopia virtuale
Cosè una colonscopia? La colonscopia virtuale è una tecnica che utilizza una TAC per costruire immagini virtuali del colon. La colonscopia è una procedura che consente a un esaminatore (di solito u
 9 modi per vivere più facilmente con la malattia da reflusso acido
Sebbene ti sia stata diagnosticata una malattia da reflusso gastroesofageo (GERD), ciò non significa che non puoi goderti la vita al massimo. Ci sono modi per controllare la GERD che non faranno neces
9 modi per vivere più facilmente con la malattia da reflusso acido
Sebbene ti sia stata diagnosticata una malattia da reflusso gastroesofageo (GERD), ciò non significa che non puoi goderti la vita al massimo. Ci sono modi per controllare la GERD che non faranno neces
 La dieta occidentale può aumentare il rischio di "sepsi mortale",
avvertire gli esperti Una nuova ricerca condotta presso la Portland State University suggerisce che la dieta occidentale può aumentare il rischio di sepsi grave e mortalità per infezione.
La dieta occidentale può aumentare il rischio di "sepsi mortale",
avvertire gli esperti Una nuova ricerca condotta presso la Portland State University suggerisce che la dieta occidentale può aumentare il rischio di sepsi grave e mortalità per infezione.