LIM homeodominio factor de transcripción Isl1 dirige el desarrollo normal de pilórica por la orientación Gata3
Resumen Antecedentes
Las anormalidades en el desarrollo del píloro o de la función contráctil de la causa de reflujo de píloro duodenal contenido en el estómago y aumentan el riesgo de metaplasia gástrica y cáncer. Las anormalidades de la región pilórica también están vinculados a defectos congénitos, como la estenosis relativamente común neonatal hipertrófica del píloro, y el reflujo duodenogástrico primaria. Por lo tanto, la comprensión del desarrollo del píloro es de gran relevancia clínica. Aquí, hemos investigado el papel del factor de transcripción LIM homeodominio Isl1 en el desarrollo del píloro.
Resultados
Examen de expresión Isl1 en el desarrollo de estómago de ratón mediante técnicas de inmunohistoquímica, todo el montaje in situ
hibridación y PCR en tiempo real cuantitativa demostraron que Isl1 es altamente expresado en el desarrollo de estómago de ratón, principalmente en la capa de músculo liso del píloro. Isl1 expresión también fue examinado por inmunofluorescencia en la estenosis pilórica hipertrófica humana, donde se encuentra la mayoría de las células del músculo liso de expresar Isl1. Isl1
función en el desarrollo embrionario del estómago se investigó la utilización de una tamoxifeno inducible Isl1
modelo de ratón knockout. Isl1
deficiencia llevó a casi completa ausencia de la capa muscular longitudinal exterior pilórica en el día embrionario 18,5, lo cual es consistente con Gata3
fenotipo del ratón nulo. inmunoprecipitación de la cromatina, ensayos de luciferasa, y ensayos de cambio de movilidad electroforética revelaron que Isl1 garantiza el desarrollo normal de pilórica atacando directamente a Gata3
.
Conclusiones
Este estudio demuestra que la Isl1-Gata3
eje regulador de la transcripción es esencial para desarrollo pilórica normal. Estos hallazgos son clínicamente muy relevante y pueden ayudar a comprender mejor las vías que conducen a la enfermedad del píloro.
Palabras clave
músculo liso α-actina de fondo Gata3 Isl1 píloro Francia El intestino de los vertebrados es una estructura notable que ingiere y digiere los alimentos, absorbe nutrientes, y elimina los productos de desecho. El intestino se origina a partir de una estructura tubular sencilla compuesta de tres capas germinales incluyendo un endodermo subyacente, un mesodermo esplácnico circundante, y un ectodermo [1-3]. En embriones de ratón, el intestino se vuelve modelado a lo largo de la anterior-posterior, dorsal-ventral, derecha-izquierda, y los ejes radiales. El tubo intestinal consta de intestino anterior, intestino medio, y el intestino posterior a lo largo de su eje anterior-posterior [4, 5]. Como el desarrollo progresa, el intestino anterior da lugar a la esófago, el estómago, el hígado, los pulmones y el páncreas. El intestino medio forma el intestino delgado y el intestino grueso se convierte en el intestino grueso [1, 5-8]. Comentario El estómago se deriva del intestino anterior posterior. El estómago se diferencia morfológicamente desde el tubo de intestino anterior alrededor del día embrionario 9,5 (E9.5) y la expansión del mesénquima pre-gástrica permite que el dominio del estómago para ser principio visible en E10.5 [9]. Las células mesenquimales de estómago se diferencian en cuatro capas concéntricas distintas, incluyendo lámina propia, muscular de la mucosa, y el músculo liso circular y longitudinal en diferentes etapas del desarrollo embrionario [10]. Por E11.5, el estómago está claramente aumentada. El músculo liso del estómago diferencia en E13, con una capa distinta de la actina α-músculo liso (α-SMA) células positivas que aparecen y una capa de músculo circular que forma en todo el estómago [11]. La capa de músculo liso se espesa en la región esfínter pilórico prospectivo constreñido a alrededor de E14.5 [2, 9]. En E18.5, el esfínter pilórico comienza a funcionar en la prevención de la de reflujo del contenido duodenal en el estómago [9].
El posterior o parte píloro del estómago es la unión anatómica entre el estómago y el duodeno. En el extremo del píloro, las aletas valvulares distintas del esfínter pilórico se pueden ver [2]. En condiciones fisiológicas normales, el estómago depende de su contracción peristáltica para moler y empuje el alimento parcialmente digerido, y el píloro se basa en su esfínter pilórico engrosada para controlar el flujo de los alimentos en el intestino delgado. Las anormalidades en el desarrollo pilórica o en la función contráctil del píloro causan reflujo del contenido duodenal en el estómago y aumentan el riesgo de metaplasia gástrica y cáncer [12, 13]. Anomalías del píloro están relacionados con defectos congénitos [14-16]. Por lo tanto, se ha prestado mucha atención a los elementos de regulación y las vías de desarrollo de estómago, especialmente píloro y el desarrollo del esfínter pilórico.
Los datos anteriores en chick sugirió que la proteína morfogenética ósea (BMP) de señalización regula la expresión mesenquimales de Nkx2.5
y Sox9
, que afecta el carácter del epitelio pilórica pero no tiene efecto sobre el músculo pilórico suave [5, 17], lo que sugiere que mesenquimales señalización por factores desconocidos afecta el fenotipo epitelial pilórica. En el ratón, los mecanismos moleculares de la formación del píloro son poco comprendidos, con relativamente pocos de los factores necesarios para el desarrollo normal de pilórica haber sido identificado. Aquellos que han sido incluyen Sox9
[17], SIX2
[9], Bapx1
[18], Nkx2.5
[3, 17], Gremlin
[9], y GATA3
[19, 20]. La ablación del factor de transcripción de homeodominio, SIX2
, expresada en el estómago posterior, interrumpe el engrosamiento de la capa de músculo liso pilórico y atenúa la constricción del esfínter píloro. Además, la pérdida de SIX2
elimina Sox9
expresión, y reduce Nkx2.5
y Gremlin
expresión en el píloro, aunque esta expresión se recupera más tarde [9], lo que sugiere que SIX2
, sox9
, Nkx2.5
, y Gremlin
se requieren para el desarrollo del píloro. Además, Nkx2.5
, Sox9
, y Gata3 ¿Cuáles son co-expresados en la capa muscular longitudinal externa del píloro (OLM) dorsal que madura entre E14.5 y E16.5. Tras la eliminación de Nkx2.5
o Gata3
, el OLM pilórica dorsal es casi ausente y la constricción del esfínter píloro se atenúa [20]. Francia El homeodominio LIM (LIM-HD) factor de transcripción Isl1 fue originalmente encontrado para funcionar como una proteína de unión del gen de insulina potenciador [21]. Isl1 se compone de dos dominios LIM tándem y un homeodominio. El homeodominio, con su estructura de hélice-giro-hélice, se une a secuencias de ADN reguladoras de los genes diana, mientras que los dominios LIM están implicadas principalmente en las interacciones proteína-proteína que regulan la actividad de la LIM-HD [22]. Isl1 juega un papel crítico en la determinación de células, la proliferación y la diferenciación en el sistema nervioso [23, 24], el corazón [25], y la glándula pituitaria [26]. Además, la expresión Isl1 se ha detectado en mesénquima gástrico [27, 28] y el epitelio gastrointestinal tanto en ratones embrionarias y adultas [29]. Sin embargo, el papel de Isl1 en el desarrollo de estómago aún no se ha explorado. En el presente estudio, se examinó la expresión Isl1 en el estómago. Isl1 fue altamente expresado en el estómago posterior en las primeras etapas de desarrollo y se limita principalmente a las células musculares lisas de la píloro. Para examinar Isl1
función en el desarrollo del estómago, se utilizó un modelo de ratón knockout tamoxifeno inducible. Se necesitaba un modelo inducible porque Isl1 - /-
mutantes mueren aproximadamente a E10.5 debido a defectos en la formación del corazón. Nuestros resultados muestran que Isl1 es vital para la formación de la capa OLM pilórica del estómago durante la organogénesis.
Resultados
Isl1 se expresa en el estómago de embriones de ratón
Examinamos Isl1
niveles de mRNA en el estómago de embriones de ratón mediante real PCR cuantitativa en tiempo (RT-qPCR) y todo el montaje hibridación in situ gratis (desea). Isl1
mRNA se detectó inicialmente en E11.5 por RT-qPCR. Isl1
alcanzado el nivel más alto en E13.5, seguido de una fuerte caída en E14.5, y no tuvieron cambios significativos en la edad adulta (archivo adicional 1: Figura S1a). Este resultado fue similar a un informe anterior [29]. La localización de Isl1
expresión de ARNm se investigó el uso de WISH. Se realizó Isl1
deseo en el estómago de embriones en E11.5, E13.5, E14.5 y. En E11.5, Isl1
fue localizado en la mitad posterior del estómago (archivo adicional 1: Figura S1b). Sin embargo, la Isl1
señal deseo era mucho más fuerte y se condensa en el píloro por E13.5 (Figura 1A). A medida que avanzaba el desarrollo de estómago, el píloro continuaron expresando Isl1
y expresión de Isl1
extiende al esfínter pilórico prospectivo en E14.5 (archivo adicional 1: Figura S1b). Sin embargo, la señal de WISH Isl1
debilitó considerablemente de E13.5 a E14.5. Estos resultados Isl1
DESEO coincidieron con los resultados Isl1
RT-qPCR. Figura 1 Isl1 se expresa en el desarrollo del estómago del ratón. (A) análisis de WISH estómago embrionario demostró que Isl1
expresión se limitaba al píloro en E13.5 (flecha). (B) tinción inmunohistoquímica para Isl1 en el estómago. Las secciones de embriones fueron dispuestos en rostral a caudal secuencia en E11.5 y E13.5, y la expresión Isl1 fue localizada principalmente en el mesénquima del estómago posterior. En E14.5 y E18.5, Isl1 fue altamente expresado en las células del músculo liso del píloro, aunque hubo algunas células Isl1-positivo en la lámina propia (puntas de flecha). imágenes ampliadas en regiones encuadradas A continuación se muestran las imágenes originales. D, el duodeno; Liv, el hígado; Pa, páncreas; St, estómago. Las barras de escala de las imágenes originales: 100 m; barras de escala de las imágenes ampliadas:. 50 micras
A continuación, evaluaron la expresión de proteínas mediante técnicas de inmunohistoquímica Isl1. Los resultados demostraron que Isl1 fue localizada principalmente en el mesénquima posterior del estómago de E11.5 a E13.5, entonces Isl1 se expresó en células de músculo liso del píloro (Figura 1B y archivo adicional 1: Figura S1c), y también fue detectable en el la lámina propia (Figura 1B, puntas de flecha). En el estómago de ratón adulto, sólo se observaron unas pocas células Isl1-positivas en la capa de músculo liso. (Archivo adicional 1: Figura S1c) ¿Cuáles son co-expresaron células Isl1-positiva con α-actina de músculo liso en el estómago de embriones de ratón
para ver si la expresión Isl1 estaba relacionado con un desarrollo sin problemas del músculo del píloro, se analizó la expresión de Isl1 y el medio de inmunofluorescencia primeros marcadores de músculo liso α-SMA. Los resultados demostraron que la proporción de células que expresan Isl1-positivo α-SMA aumenta gradualmente (Figura 2). En E11.5, no se detectaron células α-SMA-positivas, aunque Isl1 fue altamente expresado en el mesénquima del estómago posterior. En E14.5, un subconjunto de células Isl1-positivas, principalmente aquellos en el músculo circular interna (ICM) de la píloro, eran α-SMA positivos. Por E16.5, pilórica OLM ya ha sido objeto de diferenciación [20]. En E18.5, la mayoría de las células Isl1-positivas en el píloro eran α-SMA positivos. Isl1 expresión persistió en ICM pilórica maduro y OLM, y las células de la lámina propia también expresó Isl1 (archivo adicional 1: Figura S2). Además, la expresión Isl1 se examinó en muestras humanas de la estenosis pilórica hipertrófica por inmunofluorescencia, y los resultados demostraron que Isl1 también se expresó en células de músculo liso humanas del píloro (archivo adicional 1: Figura S3). Por lo tanto, estos resultados sugieren que Isl1 puede participar en la formación de esfínter pilórico. Figura 2 inmunotinción doble para Isl1 y actina de músculo liso α-ratón en células del músculo liso del píloro dorsal. Isl1 y α-SMA co-expresión en células de músculo liso en E11.5, E14.5, y E18.5. líneas de puntos amarillos marcan la membrana basal epitelial, gruesas líneas de puntos blancos indican la ICM y la OLM límites y líneas de puntos blancos indican OLM y cobertura de páncreas. tinción con rojo es Isl1, tinción con verde se alfa-SMA, y DAPI contratinción nuclear (ADN) es de color azul. α-SMA, α-actina de músculo liso; ICM, músculo circular interior; OLM, músculo longitudinal exterior; Pa, páncreas; St, estómago. Las barras de escala:. 50 micras
expresión Isl1 se realiza la ablación efectiva en Isl1
MCM /F
ratones knockout inducible
Para investigar los efectos de la ablación Isl1
sobre el desarrollo del estómago, que utilizan Isl1
MCM /F
inducible Cre (Isl1
MCM /Del
) ratones (Figura 3A) y Isl1
F /+ m
hielo eran utilizado como controles [30, 31]. Los embriones fueron genotipo por PCR en E18.5 (archivo adicional 1: Figura S4) e intacto o mutante Isl1
ARNm se distinguió por PCR semi-cuantitativa (Figura 3B). Figura 3 La eficiencia de la ablación Isl1 en los estómagos de Isl1 MCM /Del estómagos de ratón mutante en E18.5. El tamoxifeno inducible Cre recombinasa extirpado secuencias de ADN (A) flanqueado por dos sitios loxP
. (B) Isl1
niveles de ARN fueron extirpadas en Isl1
MCM /Del
estómagos mutantes como se ve por PCR semi-cuantitativa. Isl1
F /+ m
hielo mostró un par producto de base 592 mientras que Isl1
MCM /Del
ratones genera un producto de 303 pares de base. (C) Isl1 fue significativamente las reguladas en los niveles de proteína en Isl1
MCM /Del
estómagos mutantes como se muestra por Western blot. Expresión de embriones en E11.5 se utilizó como control positivo. (D) la expresión de proteínas en Isl1 Isl1
F /+ a
nd Isl1
MCM /Del
píloro embrionario. Isl1 expresión se redujo significativamente en Isl1
MCM /Del
estómagos embrionarias, como se ve por inmunofluorescencia. Imágenes en Isl1
F /+ a
nd Isl1
MCM /Del
se procesa en la misma diapositiva y fotografiado en la misma exposición. imágenes ampliadas de las áreas en caja se muestran en el lado derecho de las imágenes fusionadas. puntas de flechas amarillas muestran células Isl1-positivas representativas, y puntas de flechas blancas muestran células Isl1-negativos representativos. líneas de puntos amarillos marcan la membrana basal del epitelio. Las barras de escala:. analiza 50 micras
Western blot mostraron que los niveles de proteína en el estómago Isl1 embrionario de Isl1
MCM /Del
ratones fueron significativamente inferiores a los de Isl1
F /+ m
hielo (Figura 3C). resultados de inmunofluorescencia demostrado reducir significativamente la tinción Isl1 en píloro de Isl1
MCM /Del
ratones en comparación con los controles (Figura 3D). Estos datos demuestran que Isl1
expresión era efectivamente las reguladas en Isl1
MCM /Del
estómagos mutantes.
Anomalías del píloro en Isl1
MCM /F
mutantes
Para investigar los efectos de la ablación Isl1
en el desarrollo del estómago, se compararon las diferencias morfológicas e histológicas entre Isl1
MCM /del Opiniones y Isl1
F /+ s
tomachs en E18.5. En E18.5, se observó líquido amarillo en Isl1
MCM /Del
estómagos, pero no en los estómagos de Isl1
F /+ l
ittermates (Figura 4A, asterisco) . El examen histológico demostró que la capa de músculo liso pilórico dorsal era mucho más delgada en el píloro de Isl1
MCM /Del
ratones en comparación con los controles (Figura 4B). Se examinó la expresión y distribución de α-SMA en tanto Isl1
MCM /Del
mutantes y Isl1
F /+ p
ylorus. Los resultados demostraron que la deficiencia de inmunofluorescencia Isl1 llevó a casi completa ausencia de la capa OLM pilórica en E18.5, y las células restantes fueron débilmente organizados (Figura 5A, asteriscos). Además, la constricción del esfínter pilórico se atenuó en Isl1
MCM /Del
estómagos mutantes en comparación con constricción en Isl1
F /+ s
tomachs (Figura 5B) . Además, analizamos la expresión de la proteína específica del músculo liso calponin-1 en E18.5, e inmunofluorescencia resultados demostraron que la pérdida de Isl1 también dio lugar a casi ausencia de calponin-1 de expresión en la capa OLM pilórica dorsal, similar al resultado con α- SMA (archivo adicional 1: Figura S5). Sox9 se expresa tanto en el epitelio y el mesénquima [9] y es necesario para el desarrollo de OLM y la formación de constricción esfínter pilórico [20]. Nuestros resultados mostraron que la inmunofluorescencia Sox9 se mantuvo en niveles normales en el epitelio del estómago de Isl1
MCM /Del
ratones E14.5 y E18.5 en la (Figura 6, puntas de flecha), pero el área de la musculatura lisa del píloro que expresan Sox9 se redujo significativamente en Isl1
MCM /Del
mutantes en E14.5 (Figura 6A, asteriscos) y ausentes en E18.5 (Figura 6B, asteriscos). Por lo tanto, se requiere Isl1 Opiniones de Sox9
expresión en células OLM pilórico dorsales. Estos resultados indican que Isl1 es crítica para la regulación del desarrollo de ratón músculo liso pilórico. Figura 4 morfológica y cambios histológicos en el desarrollo de estómago Isl1 MCM /Del mutantes. (A) Gross y evidencia microscópica de los defectos de estómago en Isl1
MCM /Del
ratones. Todo el montaje vistas en E18.5 en Isl1
F /+ a
nd Isl1
MCM /Del
estómagos de ratón. Isl1
MCM /Del
estómagos mutante carecía de un esfínter pilórico funcional (punta de flecha), lo que permite el reflujo de líquido como se observa en los embriones mutantes. líquido amarillo se denota con asterisco. (B) con hematoxilina y eosina de Isl1
F /+ a
nd Isl1
MCM /Del
píloro ratón en E18.5. El dorsal del músculo liso del píloro (negro región en caja) era prominente en Isl1
F /+ e
mbryos, pero era mucho más delgada en Isl1
MCM /Del
embriones. El resto de la píloro era histológicamente normal. líneas de puntos verdes marcan la membrana basal del epitelio. imágenes ampliadas en regiones encuadradas A continuación se muestran las fotos originales. Las barras de escala de las fotos originales: 200 micras; barras de escala de las imágenes ampliadas: 50 m. H &. E, hematoxilina y eosina
Figura 5 Pérdida de Isl1 interrumpe la formación de la dorsal del píloro muscular longitudinal externa. (A) La inmunofluorescencia de Isl1 y α-SMA en Isl1
F /+ a
nd Isl1
MCM /Del
píloro embriones en E18.5. La pérdida de Isl1 dio como resultado la pérdida de casi completa de las células alfa-SMA-positivas en el OLM pilórica dorsal (asteriscos). líneas de puntos amarillos marcan la membrana basal epitelial y líneas de puntos blancos indican la ICM y la OLM límite. tinción con rojo es Isl1, tinción con verde se alfa-SMA, y DAPI contratinción nuclear (ADN) es de color azul. Barras de escala: 50 m. (B) α-SMA inmunofluorescencia de Isl1
F /+ a
nd Isl1
MCM /Del
píloro embriones en E18.5. En comparación con Isl1
F /+ e
mbryos, la constricción esfínter pilórico era más amplia en Isl1
MCM /Del
animales. se muestran las mediciones de constricción del esfínter. Las barras amarillas demarcan el píloro y ponen de manifiesto la marcada diferencia en anchura entre + /
nd Isl1
MCM /Del
muestras Isl1
F a. Los datos son la media ± SEM (n =
6 ratones por grupo), * P Hotel < 0,05 (t de Student
-test). Barras de escala: 50 m. α-SMA, α-actina de músculo liso; ICM, músculo circular interior; OLM, muscular longitudinal externa.
Figura 6 Pérdida de Isl1 elimina la expresión Sox9 músculo dorsal del píloro longitudinal externa. (A) inmunotinción doble para Isl1 y Sox9 en el píloro dorsal en E14.5. En ausencia de Isl1, el dominio de las células mesodérmicas (asteriscos) que expresa Sox9 era más pequeña. (B) inmunotinción doble para Isl1 y Sox9 en el píloro dorsal en E18.5. Inducible por nocaut Isl1 elimina efectivamente la expresión Isl1, con la pérdida concomitante de Sox9 expresión en las células dorsal OLM (asteriscos). sox9 expresión era normal en el epitelio del estómago en ambas etapas (puntas de flecha). líneas de puntos amarillos marcan la membrana basal epitelial y líneas de puntos blancos ICM independiente y OLM. tinción con rojo es Isl1, tinción verde es Sox9, y DAPI contratinción nuclear (ADN) es de color azul. Barras de escala: 50 m. ICM, músculo circular interior; OLM, muscular longitudinal externa.
Expresión y distribución de producto del gen de proteína 9,5 (PGP9.5), un marcador del sistema nervioso entérico [32], estaba intacto en E18.5 en Isl1
MCM /Del
estómagos mutantes (archivo adicional 1: Figura S6). Pancreático y duodenal gen homeobox 1 (Pdx1) se expresa en las células epiteliales del segmento antral-pilórico y el duodeno rostral [33]. Nuestros resultados mostraron que la expresión de inmunofluorescencia Pdx1 fue similar en Isl1
MCM /Del
ratones en comparación con los controles en E18.5 (archivo adicional 1: Figura S7). Además, se estableció el estómago del ratón y límite epitelial duodenal entre E14.5 y E16.5 [34], este periodo que coincide con el desarrollo de la capa de OLM [20]. Pusimos a prueba la integridad de la frontera del píloro epiteliales del intestino, el estómago pequeño en E18.5 mediante el examen de la expresión de un marcador de Cdx2 epitelio-delgado específica [19]. Nuestros resultados de inmunohistoquímica demostraron que la posición de la frontera del píloro epitelial en Isl1
MCM /Del
ratones fue similar a la de los controles (archivo adicional 1: Figura S8). Estos resultados indican que la pérdida de Isl1 no afecta la inervación o desarrollo epitelial del píloro.
Pérdida de Isl1 no afecta a la proliferación o la apoptosis de pilórica músculo circular interior y células musculares longitudinales exteriores
Para ver si la expresión Isl1 fue relacionada a la proliferación celular del píloro, examinamos co-localización de Isl1 y la bromodesoxiuridina proliferativa marcador (BrdU) mediante inmunofluorescencia en Isl1
F /+ m
hielo. Nuestros resultados mostraron que las células BrdU-positivas eran densos en E11.5 y esparcidos por las regiones de ICM y OLM en E14.5 y E18.5 (archivo adicional 1: Figura S9a). Además, la proporción de la proliferación de células ICM y OLM no fue significativamente diferente entre Isl1
MCM /Del Opiniones y Isl1
F /+ m
hielo en E18.5 ( archivo adicional 1:. Figura S9b): perfil Para evaluar el potencial de influir en la apoptosis, se examinaron escinde la caspasa 3 de expresión en E18.5, y nuestros resultados de inmunofluorescencia mostró que no había caspasa 3-positivo en las células ICM pilórica o la capa OLM de Isl1
MCM /del Opiniones y Isl1
F /+ m
hielo (archivo adicional 1: Figura S10). Estos datos indican que Isl1 ablación no afecta a la proliferación o la apoptosis de las ICM pilórico y células OLM.
La pérdida de Isl1 en disminución de la expresión de Gata3
, Gremlin
, y Nkx2.5
Dado que un número de factores, incluyendo Bapx1
[18], Barx1
[35], Gata3
[19, 36], Gremlin
[5], Nkx2.5
[2] y SIX2
[9], se han notificado a estar involucrados en la regulación del desarrollo del píloro, se examinaron los efectos de la pérdida de Isl1 en la expresión de estos genes en E14.5 y E18.5. RT-qPCR mostraron que la pérdida de Isl1
no afectó a la expresión de Bapx1
o Barx1
en cualquiera E14.5 o E18.5 (Figura 7A, B). En E18.5, α-SMA Opiniones y SIX2
niveles de mRNA fueron significativamente inferiores en Isl1
MCM /Del
ratones en comparación con los controles (Figura 7B). En tanto E14.5 y E18.5, Nkx2.5
, Gata3
, y Gremlin
niveles de mRNA en el estómago de Isl1
MCM /Del
ratones fueron inferiores a los controles (Figura 7A, B). GATA3
niveles de mRNA fueron de aproximadamente 70% disminuyó en ambas etapas examinados (Figura 7A, B). Sobre la base de estos resultados, se determinó Isl1
, Gata3
, Gremlin
, y Nkx2.5
expresión en Isl1
MCM /F
mutante y Isl1
F /+ s
tomachs usando deseen. Los resultados demostraron que la expresión de cada uno de estos genes se limita principalmente a la región pilórica, como se esperaba; Gata3
expresión fue más reducida en los estómagos de mutantes; y Gremlin Opiniones y Nkx2.5
sólo tenía cambios sutiles (Figura 7E, F). Isl1 Opiniones y Gata3
expresión fueron los más fuertemente afectados (Figura 7C, D). Estos resultados fueron consistentes con los datos de RT-qPCR y sugieren que Isl1 regula la expresión de Gata3
, Gremlin
, y Nkx2.5
. Figura 7 la expresión génica aberrante en hindstomach en Isl1 MCM /Del mutantes. (A) Análisis de RT-qPCR de los niveles de mRNA de factores de transcripción hindstomach enriquecida en E14.5 indica una reducción significativa de la α-SMA
, SIX2 Nkx2.5
, Gata3
, y Gremlin
ARNm en Isl1
MCM /Del
estómagos mutantes (n = 4
). Todos los resultados se normalizaron a niveles de Gapdh
mRNA. (B) Análisis de RT-qPCR de niveles de ARNm de los factores de transcripción hindstomach enriquecida en E18.5 indica una reducción significativa de Nkx2.5
, Gata3
, y Gremlin
mRNA en el Isl1
MCM /Del
estómagos mutantes (n = 4
). Todos los resultados se normalizaron a niveles de Gapdh
mRNA. Los datos son la media ± SEM (n = 6
ratones por grupo). * P Hotel < 0,05 frente a Isl1
F /+
; ** P Hotel < 0,01 frente Isl1
F /+ gratis (t-test de Student
). (CF) Análisis de ARNm de WISH confirmó la pérdida de Isl1
, Gata3
, Gremlin
, y la expresión de ARNm Nkx2.5
en E14.5 en el Isl1
MCM /Del
mutante estómagos. Isl1 Opiniones y Gata3
ARNm fueron severamente regulados hacia abajo en Isl1
MCM /Del
ratones, mientras que Gremlin Opiniones y Nkx2.5
expresión se reducen ligeramente. Las flechas apuntan a que el esfínter pilórico.
Isl1 dirige Gata3
y activa su transcripción
Gata3
se expresa selectivamente en el píloro del embrión de ratón en desarrollo [19, 20]. Se observó la expresión de ambos Isl1 Opiniones y Gata3
mRNA en el píloro a E14.5, pero si Gata3 y Isl1 se expresan en las mismas células no ha sido explorado. Por lo tanto, hemos examinado la expresión de Isl1 y Gata3 por análisis de inmunofluorescencia. Los resultados demostraron que las proteínas Isl1 y GATA3 se co-expresada dentro de las mismas células del píloro en E14.5 y E18.5 en Isl1
F /+ c ontrol
estómagos (Figura 8). Además, el área que expresa Gata3 fue significativamente menor en Isl1
MCM /Del
capa de músculo liso del píloro mutante en E14.5 (Figura 8A) y se perdió en E18.5 en la capa OLM pilórica (Figura 8B). Por lo tanto, se requiere Isl1 Opiniones de Gata3
expresión en la capa OLM pilórica dorsal. Para investigar si Isl1 regula el desarrollo del píloro mediante la regulación directa Gata3
, se realizó un análisis de la bioinformática locus genómico Gata3. El ratón Gata3
gen contiene varios elementos de respuesta putativos Isl1 (ATTA /Taat) a -2,832 pares de bases (pb) a 1.002 pb de los sitios de iniciación de la transcripción [37]. Se identificaron 10 áreas que contenían un Isl1 putativo sitio (Figura 9A) de unión, y 10 pares de cebadores correspondientes fueron diseñados para amplificar estas regiones siguientes inmunoprecipitación de la cromatina (CHIP), los estudios que utilizan anticuerpos a Isl1. Se obtuvo ADN genómico inmunoprecipitó a partir de regiones pilórico de embriones de ratón en E14.5. De las 10 zonas de unión Isl1 putativos, dos regiones discretas, en el -2.558 -2.303 pb pb a (región P1) y -1081 pb a -855 pb (región P6), fueron ocupados por la proteína Isl1. Este resultado fue confirmado por PCR semi-cuantitativa (Figura 9B) y el método de enriquecimiento de veces (Figura 9C). Figura 8 Pérdida de Isl1 elimina el músculo longitudinal exterior expresión pilórica dorsal Gata3. (A) inmunotinción doble para Isl1 y Gata3 en el píloro dorsal en E14.5. La región de las células del mesodermo (asteriscos) que expresa Gata3 fue menor en el Isl1
MCM /Del
píloro que Isl1 hotels, FL +
. (B) inmunotinción doble para Isl1 y Gata3 en el píloro dorsal en E18.5. Inducible por nocaut Isl1 elimina efectivamente la expresión Isl1, con la consiguiente pérdida de expresión en las células Gata3 dorsal OLM (asteriscos). líneas de puntos amarillos marcan la membrana basal epitelial y líneas de puntos blancos indican el límite ICM y OLM. punta de flecha blanca indica tinción no específica. Todos los autores leído y aprobado el manuscrito final.
 Las bacterias intestinales pueden predecir el riesgo de hipertensión pulmonar
Las bacterias intestinales pueden predecir el riesgo de hipertensión pulmonar
 La codificación de metabarras de ADN podría mejorar el análisis de la dieta humana
La codificación de metabarras de ADN podría mejorar el análisis de la dieta humana
 Probióticos como terapia adyuvante para pacientes con COVID-19
Probióticos como terapia adyuvante para pacientes con COVID-19
 La dieta y la nutrición influyen en el microbioma de la mucosa del colon
La dieta y la nutrición influyen en el microbioma de la mucosa del colon
 Científicos resuelven misterioso caso de síndrome de cervecería automática
Científicos resuelven misterioso caso de síndrome de cervecería automática
 Los mensajeros de proteínas del microbioma humano arrojan luz sobre la salud humana
Los mensajeros de proteínas del microbioma humano arrojan luz sobre la salud humana
 Cómo estimular su sistema inmunológico para combatir el coronavirus
Mientras el mundo lucha contra la pandemia de coronavirus, cada vez más personas intentan protegerse contra el virus por todos los medios posibles. Actualmente no existen vacunas contra el síndrome re
Cómo estimular su sistema inmunológico para combatir el coronavirus
Mientras el mundo lucha contra la pandemia de coronavirus, cada vez más personas intentan protegerse contra el virus por todos los medios posibles. Actualmente no existen vacunas contra el síndrome re
 Estudio vincula el consumo de vegetales fermentados con una baja mortalidad por COVID-19
Un nuevo e intrigante estudio realizado por investigadores en Europa sugiere que es probable que las tasas de mortalidad por coronavirus 2019 (COVID-19) sean más bajas en países donde las dietas son r
Estudio vincula el consumo de vegetales fermentados con una baja mortalidad por COVID-19
Un nuevo e intrigante estudio realizado por investigadores en Europa sugiere que es probable que las tasas de mortalidad por coronavirus 2019 (COVID-19) sean más bajas en países donde las dietas son r
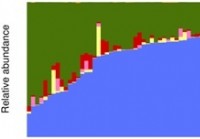 El estudio describe la base de datos inicial del microbioma intestinal sano y el perfil de abundancia
En un estudio publicado el 11 de septiembre, se describe una base de datos inicial de microbioma intestinal sano y un perfil de abundancia. 2019 en la revista de acceso abierto PLOS ONE de Charles Had
El estudio describe la base de datos inicial del microbioma intestinal sano y el perfil de abundancia
En un estudio publicado el 11 de septiembre, se describe una base de datos inicial de microbioma intestinal sano y un perfil de abundancia. 2019 en la revista de acceso abierto PLOS ONE de Charles Had