Für das Studium, veröffentlicht 26. Mai in Zelle , internationale Ermittler sammelten fast 5, 000 Proben über einen Zeitraum von drei Jahren in 60 Städten in 32 Ländern und sechs Kontinenten. Die Forscher analysierten die Proben mit einer genomischen Sequenzierungstechnik namens Shotgun-Sequenzierung, um das Vorhandensein verschiedener Mikroben zu erkennen. einschließlich Bakterien, Archaeen (einzellige Organismen, die sich von Bakterien unterscheiden), und Viren, die DNA als genetisches Material verwenden. (Andere Arten von Viren, die RNA als genetisches Material verwenden, wie SARS-CoV-2, das Virus, das COVID-19 verursacht, mit den DNA-Analysemethoden, die in dieser Prä-Pandemie-Studie verwendet wurden, nicht nachgewiesen worden wären.)
Dieses Forschungsgebiet hat wichtige Auswirkungen auf die Erkennung von Ausbrüchen bekannter und unbekannter Infektionen und auf die Untersuchung der Prävalenz antibiotikaresistenter Mikroben in verschiedenen städtischen Umgebungen.
„Jedes Mal, wenn du dich in die U-Bahn setzt, Sie pendeln wahrscheinlich mit einer völlig neuen Spezies, " sagte Senior-Autor Dr. Christopher Mason, Co-Direktor der WorldQuant Initiative for Quantitative Prediction und Professor für Physiologie und Biophysik an der Weill Cornell Medicine. Dr. Mason ist auch Mitbegründer und bezahlter Berater von Biotia und Onegevity Health. und ein bezahlter Redner für WorldQuant LLC.
Die aktuelle Studie führte zur Entdeckung von 10, 928 Viren und 748 Bakterien, die in keiner Referenzdatenbank vorhanden sind.
Dr. Mason gründete 2015 MetaSUB (kurz für Metagenomics and Metadesign of Subways and Urban Biomes), zusammen mit Dr. Evan Afshin, der damals ein Undergraduate-Student am Macaulay Honors College am Queens College war und jetzt Clinical Fellow in Physiologie und Biophysik bei Weill Cornell Medicine und bezahlter Berater für Onegevity Health ist.
Die neu veröffentlichte Studie wurde von Drs. Mason, David Danko, ein Doktorand der Weill Cornell Graduate School in Dr. Masons Labor während der Studie, und Daniela Bezdan, der zu dieser Zeit wissenschaftlicher Mitarbeiter im Bereich Computational Biomedicine bei Weill Cornell Medicine war.
Durch das Sammeln von Proben von Mikroben und die Analyse ihrer Gene – allgemein als Mikrobiom bekannt – hoffen die Forscher, mehr über die Bakterien zu erfahren, Viren und andere Mikroorganismen, die unter Menschen leben. Zum Beispiel, die Forschung kann helfen, das Auftreten antibiotikaresistenter Stämme zu identifizieren.
Die Vorhersage von Antibiotikaresistenzen allein aus genetischen Sequenzen ist eine Herausforderung, aber die Forscher konnten einige Gene kartieren, von denen bekannt ist, dass sie mit Resistenzen in Verbindung stehen. quantifizieren ihre Häufigkeit und bestätigen die Fähigkeit der genetischen Marker, Resistenz zu verleihen. Sie fanden heraus, dass einige Städte mehr Resistenzgene hatten als andere. und dass es für einige dieser Gene stadtspezifische Signaturen geben könnte.
Antibiotikaresistenzen bleiben eine große globale Herausforderung für die Gesundheit. „Während weitere Forschung erforderlich ist, dieser Datensatz zeigt den Wert und das Potenzial der Mikrobiomkartierung und -überwachung, und die Erkenntnisse, die es Ärzten liefern kann, Wissenschaftler und Beamte des öffentlichen Gesundheitswesens, " sagte Dr. Afshin.
Außerdem, Das Erlernen der kleinen Moleküle und Proteine, die von Mikroben hergestellt werden, könnte auch zur Entdeckung neuer Antibiotika sowie anderer Moleküle führen, die das Potenzial haben, als Medikamente entwickelt zu werden. Viele Antibiotika und Medikamente, die derzeit verwendet werden, stammen aus mikrobiellen Quellen.
Entdeckungen über neue mikrobielle Arten könnten auch zu neuen Laborinstrumenten und -ansätzen führen, B. neuartige Möglichkeiten zur Verwendung des molekularen Bearbeitungswerkzeugs CRISPR. In dieser Studie, die Forscher fanden 838, 532 neuartige CRISPR-Arrays – Schnipsel viraler DNA, die in Bakterien gefunden wurden – und 4,3 Millionen neue Peptide (kleine Proteine).
Aufgrund dieser Probenahmebemühungen Dr. Mason sagte, dass er mit etwa 90-prozentiger Genauigkeit vorhersagen kann, wo eine Person lebt, einfach durch Sequenzieren der DNA auf ihren Schuhen. Es wurde festgestellt, dass viele Faktoren das Mikrobiom einer Stadt beeinflussen, einschließlich Gesamtbevölkerung und Bevölkerungsdichte, Elevation, Nähe zum Meer und zum Klima. Die Erkenntnisse über diese unterschiedlichen Signaturen könnten zukünftige forensische Studien ermöglichen.
Ein Mikrobiom enthält molekulare Echos des Ortes, an dem es gesammelt wurde. Eine Küstenprobe kann salzliebende Mikroben enthalten, während eine Probe aus einer dicht besiedelten Stadt eine bemerkenswerte Artenvielfalt aufweisen kann. ", sagte Dr. Danko.
Dr. David Danko, Student der Wirtschaftswissenschaften, Weill Cornell Graduiertenschule.
Drs. Mason und Afshin begannen 2013 mit dem Sammeln und Analysieren von Mikrobenproben im New Yorker U-Bahn-System. Nachdem sie ihre ersten Ergebnisse veröffentlicht hatten, genannt PathoMap, Sie wurden von Forschern aus der ganzen Welt kontaktiert, die ähnliche Studien für ihre eigenen Städte durchführen wollten.
Das internationale Interesse inspirierte Dr. Masons Labor dazu, MetaSUB zu gründen und Daniela Bezdan als Forschungsleiterin zu gewinnen. „Wir brauchten international anerkannte Protokolle, Logistik- und Kooperationsvereinbarungen mit Wissenschaftlern, Anbieter, Regierungsstellen und philanthropische Stiftungen für potenziell 100 Städte in 20 Ländern, “ sagte Bezdan.
Heute wächst MetaSUB weiter und hat sich auf das Sammeln von RNA- und DNA-Proben aus der Luft ausgeweitet, Wasser und Abwasser, zusätzlich zu harten Oberflächen. Dies hat in drei Bundesstaaten (Florida, New York und Wisconsin), und das Teil des neuen National Wastewater Surveillance System (NWSS) des Center for Disease Control and Prevention ist.
Die Gruppe betreut auch Projekte wie den Global City Sampling Day (gCSD), findet jedes Jahr am 21. und hat bereits weitreichende Studien durchgeführt, darunter eine umfassende mikrobielle Analyse von Rio de Janeiro, während und nach den Olympischen Sommerspielen 2016. Viele der in der aktuellen Studie analysierten Proben wurden am Global City Sampling Day in den Jahren 2016 und 2017 gesammelt.
Die Probenahme in New York City wurde mit Unterstützung des Weill Cornell Medicine Clinical and Translational Science Center (CTSC) durchgeführt. in Zusammenarbeit mit dem leitenden CTSC-Programmmanager Jeff Zhu. Dr. Mason und seine Kollegen bereiten sich derzeit auf die diesjährige Veranstaltung vor.
„Als wir 2015 anfingen, das Konsortium bestand aus 16 Städten; sechs Jahre später haben wir mehr als 100 Städte. Es ist toll, diese Gruppe von neugierigen, selbststartende und begeisterte Mitermittler, " sagte Dr. Mason, der auch Professor für Computational Genomics in Computational Biomedicine am HRH Prince Alwaleed Bin Talal Bin Abdulaziz Al-Saud Institute for Computational Biomedicine bei Weill Cornell Medicine ist.
"Obwohl Proben auf der ganzen Welt gesammelt werden, Ein Großteil der Analysen wird hier in New York City bei Weill Cornell Medicine durchgeführt. “ sagte Dr. Mason. Die Analyse und Zusammenstellung von Sequenzen nutzte auch Bridges und Bridges-2, Extreme Science and Engineering Discovery Environment (XSEDE)-Supercomputer am Pittsburgh Supercomputing Center.
MetaSUB-Forscher in der Schweiz (Dr. Andre Kahles und Gunnar Rätsch) nutzten diese Baugruppen und Rohdaten, um eine durchsuchbare, globales DNA-Sequenzportal (MetaGraph), das alle bekannten genetischen Sequenzen (einschließlich MetaSUB-Daten) indiziert. Das Portal ordnet alle bekannten oder neu entdeckten genetischen Elemente ihrem Standort auf der Erde zu und kann bei der Entdeckung neuer mikrobieller Interaktionen und mutmaßlicher Funktionen helfen.
Die DNA-Isolierung aus Proben wurde größtenteils mit Unterstützung von Zymo Research und Promega durchgeführt. und sequenziert in Zusammenarbeit mit Dr. Shawn Levy am HudsonAlpha Institute for Biotechnology, Dr. Klas Udekwu von der Universität Stockholm und dem New York Genome Center. Zukünftige und laufende Studien werden RNA und DNA mit Long Reads und räumlichen Bildgebungsmethoden untersuchen. sowie die Metaboliten von den globalen Standorten zu verfolgen, und die genetische Karte im planetaren Maßstab weiter zu aktualisieren.
 Wie Proctalgia Fugax diagnostiziert wird
Wie Proctalgia Fugax diagnostiziert wird
 Was ist laparoskopische ventrale Netzrektopexie?
Was ist laparoskopische ventrale Netzrektopexie?
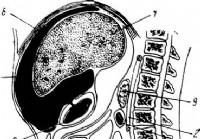 Verdeckte Perforation eines perforierten Geschwürs - Diagnose eines akuten Abdomens
Verdeckte Perforation eines perforierten Geschwürs - Diagnose eines akuten Abdomens
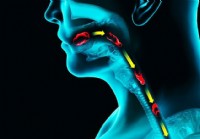 Was ist der Unterschied zwischen Odynophagie und Dysphagie?
Was ist der Unterschied zwischen Odynophagie und Dysphagie?
 Machen Fehler im Lebensmittelgeschäft Sie krank?
Machen Fehler im Lebensmittelgeschäft Sie krank?
 So bereiten Sie Bohnen richtig zu (damit sie gut für den Darm sind)
So bereiten Sie Bohnen richtig zu (damit sie gut für den Darm sind)
 Gelbsucht bei Erwachsenen (Hyperbilirubinämie)
Gelbsucht Definition und Fakten Bild von Gelbsucht im Gesicht und in den Augen Mit freundlicher Genehmigung der CDC Gelbsucht ist eine gelbliche Verfärbung der Haut, der Schleimhäute und des Aug
Gelbsucht bei Erwachsenen (Hyperbilirubinämie)
Gelbsucht Definition und Fakten Bild von Gelbsucht im Gesicht und in den Augen Mit freundlicher Genehmigung der CDC Gelbsucht ist eine gelbliche Verfärbung der Haut, der Schleimhäute und des Aug
 Psychotherapien können langfristigen Nutzen für Reizdarmpatienten haben
Neueste Verdauungsnachrichten Wenn eine Nierentransplantation fehlschlägt, ist es am besten, es noch einmal zu versuchen Lassen Sie sich Ihr Weihnachtsfest nicht von Sodbrennen ruinieren Nachfrage na
Psychotherapien können langfristigen Nutzen für Reizdarmpatienten haben
Neueste Verdauungsnachrichten Wenn eine Nierentransplantation fehlschlägt, ist es am besten, es noch einmal zu versuchen Lassen Sie sich Ihr Weihnachtsfest nicht von Sodbrennen ruinieren Nachfrage na
 Mundkrebs bewältigen
Der Umgang mit Mundkrebs kann eine Herausforderung sein, nicht nur wegen der Härte der Krebsbehandlung, sondern weil wir uns beim Essen, Trinken und Trinken auf unseren Mund, unsere Zunge und unseren
Mundkrebs bewältigen
Der Umgang mit Mundkrebs kann eine Herausforderung sein, nicht nur wegen der Härte der Krebsbehandlung, sondern weil wir uns beim Essen, Trinken und Trinken auf unseren Mund, unsere Zunge und unseren