In einer Studie, die diese Woche in . veröffentlicht wurde mBio , Forscher verwendeten Deep Sequencing, um Bakterienfamilien zu identifizieren, die mit den hochgradigen Läsionen assoziiert sind, die mit höherer Wahrscheinlichkeit krebsartig werden. Die Forscher fanden in hochgradigen Läsionen eine reichhaltigere und vielfältigere mikrobielle Mischung als in minderwertigen Läsionen oder gesunden Zervixen. Zu den am häufigsten vorkommenden Taxa, die mit den hochgradigsten Läsionen assoziiert waren, gehörten Mycoplasmatales, Pseudomonaden, und Staphylokokken.
Die Daten deuten darauf hin, dass Mycoplasma-Bakterien, bestimmtes, kann das Wachstum von HPV-bedingten Läsionen fördern, sagt der Virologe und Studienleiter Peter C. Angeletti von der University of Nebraska-Lincoln.
Die neue Studie fand eine klare Korrelation zwischen den Bakterien und den Läsionen, sagt Angeletti, aber es ist nicht klar, welche die Verbindung steuert. „Die Daten sprechen wirklich für die Idee, dass diese Bakterien an den Läsionen vorhanden sind. " sagt er. "Aber wie ist die Beziehung, und sind sie vor dem HPV da?"
Zu verstehen, dass dieser Zusammenhang klinische Auswirkungen auf Gebärmutterhalskrebs hat, die zweithäufigste Krebstodesursache bei Frauen weltweit. "Es führt uns auf einen Weg, der zur Behandlung spricht, “ sagt Angeletti. Wenn Forscher feststellen, dass Bakterien das Wachstum der Läsionen fördern, zum Beispiel, dann kann eine Vorbehandlung mit bestimmten Antibiotika die Entwicklung von Krebs verhindern.
Angeletti und seine Kollegen sequenzierten 16s-RNA-Gene in Proben, die direkt aus zervikalen Läsionen von 144 Frauen in Tansania entnommen wurden. die eine der höchsten Sterblichkeitsraten bei Gebärmutterhalskrebs weltweit hat. Die Forscher verglichen zunächst Bakterien in Proben von HIV-negativen Frauen mit Proben von Frauen, die HIV-positiv waren. Frühere Studien haben gezeigt, dass eine HIV-Infektion eine Person einem höheren Risiko für eine HPV-Infektion aussetzt.
Die Analyse zeigte eine größere Vielfalt unterschiedlicher Taxa in Proben von HIV-positiven Frauen als in Proben von Frauen, die HIV-negativ waren. Zusätzlich, In der Untergruppe der Frauen, die HIV-positiv waren und Zervixläsionen hatten, Die Forscher fanden heraus, dass höhergradige Läsionen vielfältigere Bakterienpopulationen beherbergten.
Von den identifizierten Bakterien diejenigen, die zur Mycoplasmatales-Familie gehörten, korrelierten am deutlichsten mit der Schwere der Läsionen. Im Durchschnitt, diese Bakterien waren häufiger in Gegenwart von höhergradigen Läsionen, und weniger häufig für niedriggradige Läsionen. Die Forscher fanden die gleichen relativen Beziehungen sowohl bei HIV-positiven als auch bei HIV-negativen Frauen.
Angeletti sagt, dass seine nächste Herausforderung darin besteht, die mechanistische Beziehung zwischen der Bakterienfauna und HPV herauszufinden. Die Bakterien könnten eine HPV-Infektion direkt oder indirekt fördern – indem sie chronische Entzündungen verursachen, die HPV fördert.
Gebärmutterhalskrebs ist die häufigste durch HPV verursachte Krebserkrankung. aber die Infektion erhöht auch das Risiko einer oralen, anal, Penis, und andere Krebsarten. "Es wirkt, indem es die Schleimhaut überall dort beeinflusst, wo Schleimhautgewebe vorhanden ist, " sagt Angeletti. An denselben Orten, er sagt, das Mikrobiom könnte eine Rolle bei der Förderung von Krebs spielen – und könnte in ähnlicher Weise einen Weg für zukünftige präventive Behandlungen vorschlagen.
 Wie Essen mein Leben verändert hat:Erdrückende Müdigkeit, Depressionen und Verdauungsprobleme überwinden
Wie Essen mein Leben verändert hat:Erdrückende Müdigkeit, Depressionen und Verdauungsprobleme überwinden
 Andreas TEDx Talk ist online!
Andreas TEDx Talk ist online!
 Weltgesundheitstag – Reden wir über SIBO
Weltgesundheitstag – Reden wir über SIBO
 Die Forschung befasst sich mit Aerobiomen,
Die Forschung befasst sich mit Aerobiomen,
 Wissenschaftler entwickeln Peptide, die das Gleichgewicht der Darmbakterien wiederherstellen und Arteriosklerose umkehren
Wissenschaftler entwickeln Peptide, die das Gleichgewicht der Darmbakterien wiederherstellen und Arteriosklerose umkehren
 Was sind die besten Nahrungsmittel bei Morbus Crohn?
Was sind die besten Nahrungsmittel bei Morbus Crohn?
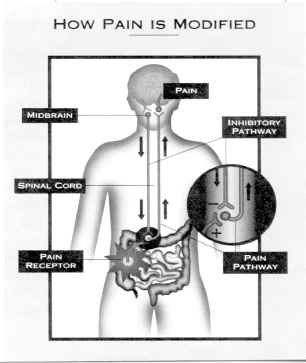 Zentral vermitteltes Bauchschmerzsyndrom
Menschen mit funktionellen Magen-Darm-Erkrankungen (GI) können eine Vielzahl von Symptomen haben, die von schmerzlosem Durchfall oder Verstopfung bis hin zu Schmerzen im Zusammenhang mit Durchfall und
Zentral vermitteltes Bauchschmerzsyndrom
Menschen mit funktionellen Magen-Darm-Erkrankungen (GI) können eine Vielzahl von Symptomen haben, die von schmerzlosem Durchfall oder Verstopfung bis hin zu Schmerzen im Zusammenhang mit Durchfall und
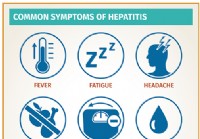 28. Juli:Welt-Hepatitis-Tag
Welt-Hepatitis-Tag Am 28. Juli wird der Welt-Hepatitis-Tag begangen. Jedes Jahr bemühen sich Ärzte für Verdauungsmedizin und viele andere Spezialisten, das Bewusstsein für Hepatitis weltweit zu schär
28. Juli:Welt-Hepatitis-Tag
Welt-Hepatitis-Tag Am 28. Juli wird der Welt-Hepatitis-Tag begangen. Jedes Jahr bemühen sich Ärzte für Verdauungsmedizin und viele andere Spezialisten, das Bewusstsein für Hepatitis weltweit zu schär
 7 Hormone, die jede Frau verstehen sollte
Sind Sie eine Frau mit Hormonproblemen? Oder kennst du eine Frau, die sie hat? Sie könnten das an Dingen erkennen wie … Wenig Energie, schläft viel, fühlt sich nie ausgeruht Hitzewallungen, geringer
7 Hormone, die jede Frau verstehen sollte
Sind Sie eine Frau mit Hormonproblemen? Oder kennst du eine Frau, die sie hat? Sie könnten das an Dingen erkennen wie … Wenig Energie, schläft viel, fühlt sich nie ausgeruht Hitzewallungen, geringer