Epstein-Barr-Virus-spezifische Methylierung von menschlichen Genen in Magenkrebszellen
Zusammenfassung
Hintergrund
Epstein-Barr-Virus (EBV) ist in 10% aller Magen-Adenokarzinome aber seine Rolle bei der Tumorentwicklung und Wartung Überreste gefunden unklar. Das Ziel dieser Studie war, EBV-vermittelte Fehlregulation der zellulären Faktoren in Magen Karzinogenese beteiligt zu untersuchen.
Methods
Genexpressionsmuster in EBV-negativen sucht wurden und EBV-positive AGS Magenepithelzellen eine geringe Dichte Mikroarray verwenden, reverse Transkription PCR, histochemische Färbungen und methylierungsspezifische DNA-Sequenzierung. Die Expression von PTGS2 (COX2) wurde in AGS-Zellen und in primären Adenokarzinom des Magens Gewebe gemessen.
Ergebnisse
Array Studien, fast die Hälfte der 96 menschlichen Gene getestet, die 15 verschiedene krebsbedingte Signaltransduktionswege wurden dysreguliert nach EBV-Infektion. Die reverse Transkription PCR bestätigten signifikanten Einfluss auf die Faktoren, die vielfältigen Funktionen wie Zellzyklusregulation (IGFBP3
, CDKN2A, CCND1, HSP70, ID2, ID4)
, DNA-Reparatur (BRCA1, TFF1
), Zelladhäsion ( ICAM1
), Entzündung (COX2
) und der Angiogenese (HIF1A
). Demethylierung unter Verwendung von 5-Aza-2'-Desoxycytidin umgekehrt die EBV-vermittelte Dysregulation für alle hier aufgeführten 11 Gene. Für einige Promotor-Sequenzen, CpG-Insel-Methylierung und Demethylierung trat in einem EBV-spezifischen Muster, wie durch Bisulfit-DNA-Sequenzierung gezeigt. Immunhistochemie war weniger empfindlich als die Herunterregulierung von COX2 Western-Blot war Infektion bei EBV zur Detektion. Virus-bezogenen Dysregulation von COX2 Ebenen in vitro
wurde nicht rekapituliert in vivo
unter natürlich Magenkrebs Gewebe infiziert.
Schlussfolgerungen
EBV verändert die menschliche Gen-Expression in einer Weise, die zu dem einzigartigen Pathobiologie des Virus beitragen könnten -assoziierten Krebs. Darüber hinaus deuten darauf hin, die Häufigkeit und Umkehrbarkeit der Methylierung bezogenen Transkriptions Veränderungen, dass Demethylierungsagenzien für die Verwaltung von EBV-verwandten Karzinom therapeutisches Potenzial haben.
Hintergrund
Magenkrebs die vierthäufigste Krebsart und die zweithäufigste Ursache für Krebs Tod weltweit [1]. Eine Vielzahl von genetischen Veränderungen sowie Infektions- und andere Umweltfaktoren scheinen Faktoren in Magen-Krebsentstehung zu sein. Epstein-Barr-Virus (EBV), ein doppelsträngiges DNA-Gammaherpesvirus, ist innerhalb der malignen Zellen in 10% der Magen-Adenokarzinome gefunden und Infektion scheint maligne Transformation vorausgehen [2]. Grundlagen- und klinische Beobachtungen legen nahe, dass EBV-assoziierten Magenkrebs eine andere Pathobiologie von EBV-negativen Magenkrebs haben [3-8]. Rationale Design von Virus-gerichtete Therapie erfordert ein besseres Verständnis der pathogene Rolle von EBV in Magen Karzinogenese.
Frühere Studien haben gezeigt, Verlust von drei kritischen Tumorsuppressor-Genprodukten, CDH1 (E-Cadherin), p73 und CDKN2A (p16 ), Magenkrebs in EBV-infizierten [18.09]. Virus-assoziierter Methylierung dieser Gene, zusammen mit dem Nachweis der globalen DNA Methylierung in EBV-positiven Tumoren legt nahe, dass EBV-bezogenen Magenkarzinome eine Teilmenge von CpG Insel Methylator Phänotyp (CIMP) Krebsarten sind [4, 11, 19-26]. Ein möglicher Mediator ist DNA-Methyltransferase 1 (DNMT1)
dass in natürlich infizierten Magenkrebs hochreguliert wird und könnte Methylierungsmuster Tochterzellen bei der Zellteilung [21, 27-29] propagiert helfen etablieren. Laufende Studien werden zum Verständnis der Rolle von EBV und Helicobacter-pylori-Infektion gerichtet in der Entstehung von Entzündungen und die damit verbundenen globalen Hyper bei Magenkrebs Entwicklung [22].
Zelllinie Modelle, DNMT1 Überexpression von EBV LMP1 und LMP2 vermittelt wird [21, 28 -31]. EBV scheint epigenetische Mechanismen anzuwenden, um die Host-Transkriptom zu steuern und auch Expression seiner eigenen viral kodierten Gene zu steuern, [11, 12, 14, 15, 19, 21, 24, 29, 32, 33]. Bei anfänglicher Infektion einer Zelle kann das unmethylierte virale Genom virale Replikation mit neuen Virionproduktion zu unterziehen, während eine Untergruppe infizierter Zellen eine stark methylierten viralen Genoms erwerben, die Expression von Fremdproteinen zermalmt und vermittelt langfristige Viruspersistenz haft latenten Infektion [23, 34]. Infizierte Tumoren sind in der Regel hoch methylierte DNA EBV und Methylierung bezogenen Silencing von viralen Genen zu haben, hilft zu erklären, wie infizierte Tumorimmun Zerstörung entziehen.
Während Methylierung von Gen-Promotoren typischerweise mit Transkriptionsherabregulation
über selektive Bindung von Repressor Proteinen assoziiert ist , das erste Protein jemals einen methylierten Promotor zu binden und aktivieren
gezeigt wurde EBV BZLF1, der Schlüsselfaktor für den Wechsel von latent zu replikativen Formen der viralen Infektion [35] steuern. Es scheint, dass das Virus geschickt ein Mittel zur Überwindung der Promotor-Methylierung zu seinem Vorteil entwickelt [34, 35]. Die antivirale Strategien werden auf ihre antineoplastischen Potenzial untersucht. Interessanterweise sind die am häufigsten verwendeten antivirale Mittel, Aciclovir und Ganciclovir, sind wirksam bei der viralen Replikation heruntergefahren, aber sie beseitigen nicht Ausdruck von latenten und frühen lytischen viralen Genen wie LMP1, LMP2 und BZLF1., Die klinischen Implikationen der EBV- im Zusammenhang mit Methylierung des Genoms Magenkrebs sind immens. Erstens Beweise Schwellen zeigt das Potenzial für eine verbesserte Diagnose von Magenkrebs durch Magen Wäschen für Krebs-spezifische Methylierungsmuster zu testen, vielleicht zusammen mit Tests für EBV den Virus-infizierten Untergruppe von Krebserkrankungen zu identifizieren [36-40]. Unterschiedliche Muster der Promotormethylierung in Virus-positiv gegenüber Virus-negative Zellen [11, 21, 24] unterstreichen die Notwendigkeit Methylierungsmuster in einer Art und Weise zu charakterisieren, dass die Empfindlichkeit des Assays für die Krebserkennung maximiert. Sowohl Infektion und veränderte DNA-Methylierung erscheinen frühe Ereignisse in der Karzinogenese zu sein [2, 41], die möglicherweise zu erleichtern Nachweis von präkanzerösen Läsionen im Magen-Saft.
Eine zweite klinische Implikation ist das Potenzial für eine verbesserte Behandlung von Magenkrebs unter Verwendung von Medikamenten, die die Reverse Wirkung von Promotor-Hypermethylierung [42, 43]. Insbesondere Demethylierungsagenzien, die DNA-Methyltransferase hemmen und Tumor-Suppressor-Gen-Silencing oder Onkogen-Aktivierung sind potentielle antineoplastischen Strategien [43] rückgängig machen. Zu berücksichtigen ist in der Wirkung von Demethylierungsagenzien in Virus-positiv gegenüber
Virus-negativen Tumoren auf mögliche Unterschiede gegeben werden [43-45]. Wir und andere haben gezeigt, dass natürlich infizierten Magenkrebs haben gezeigt untere CDKN2A (p16) Ausdruck [14, 15]. In einer klinischen Studie mit Fluorouracil (5FU) für Magenkrebs, CDKN2A
Promotor Methylierungsstatus war ein unabhängiger Prädiktor für das Überleben [46]. Der Grund für die Verwendung Demethylierungsagenzien wie 5-Aza-2'-Desoxycytidin in klinischen Studien beruht auf wissenschaftlichen Erkenntnissen, dass die Therapie Demethylierung die tumorigenen Eigenschaften von Krebszellen modifiziert.
Mehrere Forscher erfolgreich infizierten Epithelzelllinien mit EBV in
haben vitro
[47, 48]. unter Verwendung von Low-Density-Microarray-Analyse In der aktuellen Studie, EBV-positiven und EBV-negativen AGS-Zellen Magenkrebs wurden für Unterschiede in der Genexpression Muster untersucht und Transkription-Polymerase-Kettenreaktion (rtPCR) umkehren. AGS ist eine Zelllinie, die ursprünglich von Adenokarzinom des Magens Gewebe gezüchtet wurde und heute allgemein als Modell für Magenkrebs verwendet wird. Die Rolle der DNA-Methylierung in ausgewählten Effekte vermittelnde wurde durch Bisulfit DNA-Sequenzierung untersucht und durch die Fähigkeit eines Demethylierungsmittel Testen der Wirkung von EBV on gene silencing umkehren. Die Ergebnisse zeigten, umfangreiche Gen Dysregulation auf EBV-Infektion in AGS-Zellen mit dem Nachweis, dass Promotor-Methylierung verantwortlich ist, zumindest teilweise. Umkehrung des Virus-assoziierten Transkriptions Effekte legt nahe, dass Demethylierungsagenzien sollten auf ihr Potenzial untersucht werden Malignitäten zu steuern Wachstum von EBV-bezogenen.
Methoden
Magenkrebszelllinien
Die AGS Magenkrebs-Zelllinie (ATCC CRL- 1739) wurde in Dulbeccos modifiziertem Eagle-Medium kultiviert, das 10% fötales Rinderserum (hitzeinaktiviert für 20 Minuten bei 65 ° C) und 1% Penicillin-Streptomycin (10.000 Einheiten Penicillin, 10.000 &mgr; g /ml Streptomycin, Gibco, Carlsbad, CA) . Die Zellen wurden mit einem rekombinanten EBV-Stamm (ein Geschenk von Dr. Henri J. Delecluse) infiziert [33, 49, 50], die dazu entworfen wurde, grün fluoreszierendes Protein (GFP) und Hygromycin B-Resistenz auszudrücken, indem sie diese Gene in das prototypische B95 Klonen 0,8 Stamm von EBV, wo die zweite Kopie von oriLyt normalerweise befindet. Vor der Infektion wurden AGS Zellen mit 1 ug eines kodierenden Expressionsvektor transfiziert CD21 (der EBV-Rezeptor) und ein Puromycin-Resistenzgen von Fugene 6 unter Verwendung von (Roche, Indianapolis, IN), wie zuvor beschrieben [51]. 48 Stunden nach der Transfektion wurden unter Verwendung von 0,5 &mgr; g /ml Puromycin-HCl (Roche) CD21 Zellen enthält, positiv selektiert. Virusbestände von rekombinantem EBV wurden in Nieren-293-Zellen, eine humane embryonale epithelialen Zellinie, die durch lytische Replikation induzieren 20 ng /ml Phorbol-12-tetradecanoat-13-acetat und 3 mM Butyrat verwenden. Überstände wurden 3 Tage nach der Induktion geerntet, filtriert (0,8 &mgr; M) und bei -80 ° C bis zur Verwendung eingefroren. Puromycin-resistente AGS-Zellen wurden bei 50% der vollen Dichte in 60-mm-Gewebekulturschalen und Co-Inkubation mit 1 ml Lager Virionen plattiert. Vier Tage später wurden die EBV-infizierten Zellen AGS (heute AGS-B95-HygB) wurden positiv mit 100 ug /ml Hygromycin B (Roche) ausgewählt.
DNA-Fingerprinting bestätigt, dass AGS-Zellen in dieser Studie verwendeten entsprach dem Genotyp AGS-Zellen in der American Type Culture Collection. Fingerabdrucks durchgeführt wurde mit der PowerPlex 1.2 STR-Kit (Promega), gefolgt von Elektrophorese auf einem ABI 310 Kapillargelelektrophorese Instrument (Applied Biosystems).
Gene Expression durch histochemische Stains
Paraffinblöcke wurden aus AGS und AGS-B95- HygB Zelllinien und Paraffinblöcke primären Adenokarzinom des Magens wurden aus klinischen Archiv abgerufen. Die restlichen klinischen Proben repräsentieren alle verfügbaren EBV positiven Magenkrebs (n = 9) und eine zufällige Auswahl von EBV-negativen Magen-Krebs (n = 9). Die histochemische Flecken wurden auf Paraffinschnitten angewendet Infektion zu bestätigen und die Genexpression zu bewerten. Zur Herstellung von Blöcken, wurden gezüchtete Zellen zuerst in Dulbeccos phosphatgepufferter Salzlösung (Gibco, Invitrogen), geerntet mit 0,25% Trypsin (Gibco, Invitrogen) gespült, verstrickt in einem Gerinnsel unter Verwendung Dade Ci-Trol Coagulation Control (Citrat) -Level 1 (Dade Behring, Marburg, Deutschland) und Thrombin 200 (Pacific Hämostase, Middle VA), in 10% gepuffertem Formalin, in Paraffin eingebettet und geschnitten auf beschichtete Objektträger.
EBER
in situ Hybridisierung
durchgeführt wurde fluoreszenzmarkierte mit EBER
Sonde und Oligo (d) T-Sonde auf einer Benchmark-in-situ-Hybridisierung
System (Ventana Medical Systems, Tucson, AZ). Immunhistochemische Färbungen für EBV LMP1 und LMP2-Proteine wurden wie zuvor beschrieben durchgeführt [52] Citrat-Antigen-Retrieval verwendet und die CS1-4 Cocktail von monoklonalen Maus-Antikörpern gegen LMP1 (1: 100, Dako, capinteria, CA) und die E411 monoklonalen Ratten-Antikörpers gegen LMP2 (1 mg /ml, Asencion, München, Deutschland). Paraffinschnitte von EBV-verwandten Hodgkin-Lymphom und nach der Transplantation lymphoproliferative Störung diente als positive Kontrollen
immunhistochemischen Analyse der EBV replikativen Proteine BMRF1 und BZLF1 wurde durchgeführt unter Verwendung von Anti-BMRF1 Klon G3-E31 (1:. 200 Verdünnung, Forschung Diagnostics , Inc., Flanders, NJ) und Anti-BZLF1 Klon BZ.1 (01.25 Verdünnung, Dako, Carpinteria, CA), während menschliche PTGS2 (umgangssprachlich Cyclooxygenase-2 genannt, COX2) wurde anti-COX2 monoklonalen Antikörper untersucht unter Verwendung von ( 1: 200 Verdünnung, Cayman Chemical). Die Schnitte wurden bei 37 ° C für die EBV Targets mit primärem Antikörper für 30 Minuten inkubiert oder bei 4 ° C über Nacht für COX2, die Sperr- und Detektionsprotokolle in der Super-Sensitive Non Biotin HRP Detection Kit (Biogenex) verwendet wird. Gebundener Antikörper wurde unter Verwendung von Diaminobenzidin Chromogen (Biogenex) nachgewiesen, und die Zellen wurden mit Hämatoxylin gegengefärbt (Dako). Paraffinschnitte von oralen Haarleukoplakie diente als positive Kontrolle für die lytische EBV, während reaktives Tonsillen und Nasopharynxkarzinom Geweben als Kontrollen für COX2 Flecken diente. Die Ergebnisse wurden durch mikroskopische scoring maligner Zellen in ≥10 Felder bei 400facher Vergrößerung ergibt einen durchschnittlichen Anteil score (0 = keine, 1 = interpretiert < 1%, 2 = 1-10%, 3 = 10-33%, 4 = 33 -66%, 5 = > 66% der Zellen) und Intensität Score (0 = keine, 1 = schwach, 2 = mittel, 3 = starke Färbung). Insgesamt Scores wurden in EBV Vergleich positiv gegenüber
negativen Tumoren ein Mann-Whitney ungepaarten t-Test.
Nachweis des EBV-Genoms
Eine Batterie der quantitativen real-time PCR (Q-PCR) Assays Targeting sechs disparate Regionen des EBV-Genoms, dass die virale Infektion von AGS-Zellen war erfolgreich zu demonstrieren, wurde verwendet. Amplifizierte Produkte entdeckt wurden die ABI Prism 7500 Real-Time-PCR-Instrument mit Sequence Detection System-Software (Applied Biosystems) unter Verwendung von wie zuvor beschrieben, unter Verwendung von Primern Targeting des BamH1W
, EBNA1
, LMP1
, LMP2
, und BZLF1
Regionen des EBV-Genoms [52] oder EBER1
DNA [53]. Um zu überprüfen, für die Amplikonkontamination, enthielt jeder Lauf mindestens zwei "keine Vorlage" Kontrollen, in denen Nuklease-freies H2O für Vorlage ersetzt wurde.
Low-Density-cDNA-Microarray-Analyse
RNA wurde isoliert von AGS und AGS-B95- HygB Zellen das RNeasy RNA Mini Kit (Qiagen) nach erstmaligem QIAshredder ™ Spin-Säule (Qiagen) unter Verwendung der Zellen zu lysieren. RNA-Integrität mit Hilfe eines Agilent Bioanalyzer, Ausdruck Microarray-Analyse wurde von SABiosciences Corporation (Frederick, MD) unter Verwendung ihrer GEArray Q-Serie Menschen Signal Transduction in Krebs-Gen-Array Nach der Bestätigung. Diese Low-Density-Microarray besteht aus 96 Sonden, die in der Onkogenese beteiligt Aktivierung von 15 Signaltransduktionswege testen. (Target-Transkripte werden in Ergebnisse aufgeführt.) Biotin-markierter cDNA aus 10 &mgr; g jeder RNA-Probe, hergestellt unter Verwendung des AmpoLabeling-LPR-Methode (SABiosciences) wurde zu dem Array hybridisiert und Chemilumineszenznachweis wurde unter Verwendung einer CCD-Kamera durchgeführt. Integrierte rohe Intensitätswerte für jeden Punkt wurden von GEArray Analysis Suite-Software (SABiosciences) erzeugt, und eine weitere Analyse und Normalisierung wurde mit Microsoft Excel durchgeführt. Der tiefste Punkt Intensitätswert für jedes Array wurde als Hintergrund zu sein und wurde von jedem rohen Intensitätswert für jede Sonde abgezogen, und dann waren Spotintensitäten normiert auf die des Housekeeping-Gens, glyceraldehydephosphate (GAPDH
). Ein paarweisen Vergleich der Genexpressionsniveaus wurde zwischen den EBV-positiven Zellen (AGS-B95-HygB) und die elterliche EBV-negativen AGS-Zellen hergestellt. Wenn das Verhältnis ≥2 oder ≤0.5 war, wurde das Gen als in infizierten Zellen nach oben reguliert oder nach unten reguliert werden.
SYBR Green Semi-quantitative rtPCR Produkte ausgewählt Microarray-Genexpressionsergebnisse zu bestätigen, wurde rtPCR ausgeführt Real-Time RT mit
2-Gen-spezifischen PCR-Primer (SABiosciences). Die ReactionReady ™ First Strand cDNA Synthesis Kit (SABiosciences) wurde verwendet, um zu konvertieren 3 ug RNA in cDNA und die cDNA wurde 01.10 vor der PCR-Analyse verdünnt. Die folgenden 38 cDNAs wurden gezielt: GAPDH, A2M, ABCB1, BCL2L1, BIRC1, BIRC2, BIRC3, EN1, GADD45, HIF1A, ID2, IGFBP3, BRCA1, TMEPAI, IRF1, BCL2, BMP-4, CDKN2A (p16), FN1, HK1, ICAM1, IL2, CCND1, MDM2, COX2 (PTGS2), TFRC, WISP1, TRAF1, CDK2, VCAM1, CDKN2C (p18), CDKN1A (p21), DUSP1, HSP70, NFKB1, TNFSF10, TRIM25
und FOSL1
. Die Reaktionen wurden in einem Gesamtvolumen von 25 &mgr; l, enthaltend 1X TaqMan ® Universal-Mix (ABI) durchgeführt wird, RT 2-Gen-spezifischen Primer-Set-Mix (10 &mgr; M von jedem Primer, SABiosciences), 2,5 ul 5X SYBR grüne Lösung ( Molecular Probes, Eugene, OR) und 5 ul cDNA-Matrize. Thermocycling-Bedingungen waren: 50 ° C für 2 Minuten, 95 ° C für 10 Minuten, und 40 Zyklen von 95 ° C für 15 Sekunden und 60 ° C für 1 Minute auf einem ABI 7500-Instrument mit Sequence Detection System-Software (Applied Biosystems). Das gleiche Schwellenwert wurde für jedes Gen und Platte durch die "Relative Quantifizierung Plate" Protokoll ausgewertet verwendet. Jede PCR-Reaktion wurde in dreifacher Ausfertigung für jede Probe auf zwei getrennten Platten mit 96 Vertiefungen ausgeführt und die Ergebnisse gemittelt wurden in jedem Gen des Expressionsniveaus in der EBV-positive Vergleich
EBV-negativen Zellinie relative Differenzen zu bestimmen.
Minor Nut Bindungs Probe semi~~POS=TRUNC quantitative~~POS=HEADCOMP rtPCR
Expression von fünf ausgewählten Genen zu bewerten, die nicht auf dem Mikroarray oben beschrieben wurden, semi-quantitative rtPCR Assays wurden durchgeführt (Assays-on-Demand, Applied Biosystems) unter Verwendung der kleinen Furche bindenden Sonden Targeting-Helicase-like Transkriptions Faktor (HLTF
), trefoil factor-1 (TFF1
), Basis-Leucin-zipper-ATF-ähnlichen Transkriptionsfaktor (BATF
), Inhibitor der DNA-Bindungsprotein-4 (ID4
) und nucleostemin (NU
). Diese fünf wurden ausgewählt, weil sie angeblich in einem erheblichen Anteil der Magen-Adenokarzinome oder EBV-Krebsarten dysreguliert sind [32, 54-59]. GAPDH
diente als endogene Kontrolle zur relativen Quantifizierung Zwecke. RNA wurde in cDNA unter Verwendung des High Capacity cDNA Archive Kit (Applied Biosystems) umgewandelt, und die cDNA wurde 1:10 mit Nuklease-freiem Wasser verdünnt. Jede 50 &mgr; l PCR-Reaktion enthielt: 1X TaqMan ® Universal-Master-Mix, 1X Ziel Gene Expression Assay oder GAPDH
Endogene Kontrollmischung und 10 &mgr; l cDNA. Um Amplikonkontamination überprüfen, wurde für eine Vorlage ersetzt jedes Expressionstest auf jeder Platte mindestens zwei "keine Vorlage" Kontrollen, in denen nukleasefreiem Wasser enthielt. Thermozyklisierung Bedingungen und Datenanalyse oben für die SYBR Green rtPCR.
Demethylierung Behandlung und Sequenzierung von Bisulfit-modifizierten DNA-
wie beschrieben waren, waren Zur Untersuchung der Wirkung von Demethylierung, die AGS und AGS-B95-HygB Magenkrebszelllinien gewachsen auf 75% Konfluenz und dann für drei aufeinander folgende Tage 1 uM frischer 5-Aza-2'-desoxycytidin (5aza; Sigma) wurde täglich zugegeben. Am vierten Tag wurden die RNA und DNA aus behandelten und unbehandelten Kulturen geerntet. RNA wurde für die Genexpression Ebenen ausgewertet, und die DNA wurde für die Methylierung untersucht, nachdem Natrium Bisulfit-Behandlung (EZ DNA-Methylierungs-Kit, Zymo Research, Orange, CA) zu unmethylierte Cytosine in Uracil umwandeln, während methylierte Cytosine unverändert [60] zu halten. Die positive Kontrolle war CpGenome Universal-methylierter DNA (Chemicon) auf denselben Bisulfit-Umwandlung unterzogen, und die Steuerung der Primer C8orf4
zellulären Genpromotor bestätigt erfolgreiche Bisulfit-Umwandlung von jeder DNA-Probe [61] Targeting. CpG-Inseln wurden mit CpG Plot für jede der fünf menschlichen genes-- ICAM1
(RefSeq # NM_00201), CDKN2A (p16)
(RefSeq # NM_000077.3), ID4
(RefSeq # NM_001546) identifiziert, COX2
(RefSeq # NM_000963) und TFF1
(RefSeq�.225,2) -, für die Promotorsequenzen (3000 Basenpaare stromaufwärts der Transkriptionsstartstelle durch Exon 1) wurden von GenBank heruntergeladen. Folgende Parameter für die CpG-Insel Identifizierung wurden verwendet: Länge von mindestens 200 bp, minimale durchschnittliche Prozentsatz von C + G von 50% und minimalen durchschnittlichen Verhältnis der beobachteten C + G von 0,6 [62] zu erwarten. Zur Identifizierung wurde CpG dichte Bereiche für COX2
und TFF1
, die minimale Länge Parameter auf 50 bp reduziert [61]. Primersequenzen sind in Tabelle 1 Jede 50 &mgr; l PCR-Reaktion gezeigt, enthalten: 1X PCR-Puffer, 2 mM MgCl 2, 1 Einheit Platinum Taq DNA Polymerase (Invitrogen, Carlsbad, CA), 0,2 mM dNTPs (ABI) und 30 pmol von jedem Primer. Thermocycling-Bedingungen waren: 94 ° C für 2 Minuten, 40 Zyklen von 94 ° C für 30 Sekunden, 55 ° C für 30 Sekunden und 72 ° C für 1 Minute, 72 ° C für 10 Minuten. Um Amplikonkontamination überwachen, enthielt jeder Lauf ein "keine Vorlage" Steuerung, bei der Nuklease-freies Wasser für templateTable substituierten 1 Bisulfit-konvertierter genspezifischen PCR-Primer-Sequenzen
ICAM1
Weiterleiten
5'-TGG GGG TTG TGG TTT TAG TT-3 '
Rückwärts
5'-CTC CCT CCA CTA AAA AC-3'
Amplicongröße
412 bp
CDKN2A (p16)
Vorwärts
5'-AGA TGT TTT GTG GTT GTT GTG A-3 '
5'-CAA AAA TCT TCC ATT CTT CAA AC Rückwärts
-3 '
Amplicongröße
418 bp
ID4
Vorwärts
5'-TTT TTT GGG TAT ATA TTA GTT TGG-3'
5'-TAT CCT Rückwärts
AAT CAC TCC CTT C-3 '
Amplicongröße
477 bp
COX2
Vorwärts
5'-TAT GTG TTG TAT ATA GAG TAG A-3'
Rückwärts
5'-AAA AAA TAA TCC CCA CTC TC -3 '
Amplicongröße
399 bp
TFF1
Vorwärts 5'-TTA GGT TGG AGT GTA GTA GG-3
'
Rückwärts
5'-CCT ACT CAT ATC TAA AAA ACC C-3 '
Amplicongröße
489 bp
C8orf4 Kontrolle
Vorwärts
5'-GAA TTA AAA TAT AAG GAG AGT TTT-3 '
Rückwärts
5'-AAC ATT ACC CAA ACA TAA AAC AA-3'
Amplicongröße
328 bp
Die Sequenzierung wurde auf Amplikons von Bisulfit behandelten Vorlagen durchgeführt zu identifizieren die methylierten und nicht-methylierten CpG mit oder ohne 5aza Behandlung. Zunächst wurde jedes PCR-Produkt kloniert in pGEM-T-Vektor den pGEM-T Einfache Vector System II (Promega) und transformiert in JM109 hohe Effizienz kompetente Zellen verwendet wird. Weiße Kolonien Inserts enthielten, wurden ausgewählt und über Nacht kultiviert und Plasmid-DNA wurde unter Verwendung des QIAprep Spin Miniprep Kit (Qiagen) extrahiert. Die Sequenzierung wurde auf einem ABI 3100 Genetic Analyzer getan, um die ABI PRISM mit ™ BigDye ™ Version 1.1 Terminator Cycle Ready Reaction Kit mit AmpliTaq-DNA-Polymerase und einem M13R3 Primer. Die Ergebnisse wurden in Sequencher Software (Gene Codes, Ann Arbor, MI) heruntergeladen umgekehrt Kompliment jeder Sequenz zu erhalten, und beide Vorwärts- und Rückwärtssequenzen wurden ausgerichtet und analysiert nicht methylierten Cytosine von methylierten Cytosine zu unterscheiden. Western Blot auf dem AGS
Zelllinie
Aufgrund des Potenzials für COX2-Hemmer-Therapie COX2 Effekte zu überwinden, wurden die RNA-basierte Ergebnisse für COX2 gewählt für Studie Follow-up auf Proteinebene. Konfluente AGS-Zellen mit oder ohne EBV wurden mit 0,25% Trypsin geerntet, zweimal in phosphatgepufferter Kochsalzlösung gewaschen und durch Zentrifugation pelletiert. Zellen wurden in 500 ul NP-40 Zell-Lyse-Puffer (50 mM Tris-HCl, 150 mM NaCl, 1% NP-40, pH 8,0), inkubiert auf Eis für 30 Minuten resuspendiert und bei 12.000 rpm für 15 Minuten bei 4 versponnen ° C. Aliquots des Lysats (bei 50, 100, 150 ug Protein pro Well) wurden SDS-PAGE auf einem Tris-Glycin 4-20% Gradientengel (Invitrogen) und auf eine Nitrocellulosemembran unter Verwendung gelöst. COX2 wurde mit einer 1 festgestellt: 5000 Verdünnung des monoklonalen Antikörpers folgte eine 1: 10.000 Verdünnung des sekundären Antikörpers, konjugiert mit alkalischer Phosphatase (Amersham Biosciences) und Visualisierung mit einem Typhoon Phosphorlmager (Molecular Dynamics). Band Dichte gemessen semi-quantitativ und normiert auf Beta Aktin (ACTB) wurde im Vergleich zwischen infizierten und nicht infizierten Zellen AGS.
Ergebnisse
 Warum Sie ein Gesundheitsheld sind und was Sie mit dieser Energie anfangen können
Warum Sie ein Gesundheitsheld sind und was Sie mit dieser Energie anfangen können
 Was untergräbt wirklich die Darmgesundheit Ihrer Familie?
Was untergräbt wirklich die Darmgesundheit Ihrer Familie?
 Was sind die besten Lebensmittel bei Gallensteinen?
Was sind die besten Lebensmittel bei Gallensteinen?
 Pooping 101:4 Zeichen, dass Ihr Poop gesund ist
Pooping 101:4 Zeichen, dass Ihr Poop gesund ist
 Langfristig niedriger FODMAP | Ein Ernährungsberater erklärt
Langfristig niedriger FODMAP | Ein Ernährungsberater erklärt
 Darmbakterien können das Risiko einer pulmonalen Hypertonie vorhersagen
Darmbakterien können das Risiko einer pulmonalen Hypertonie vorhersagen
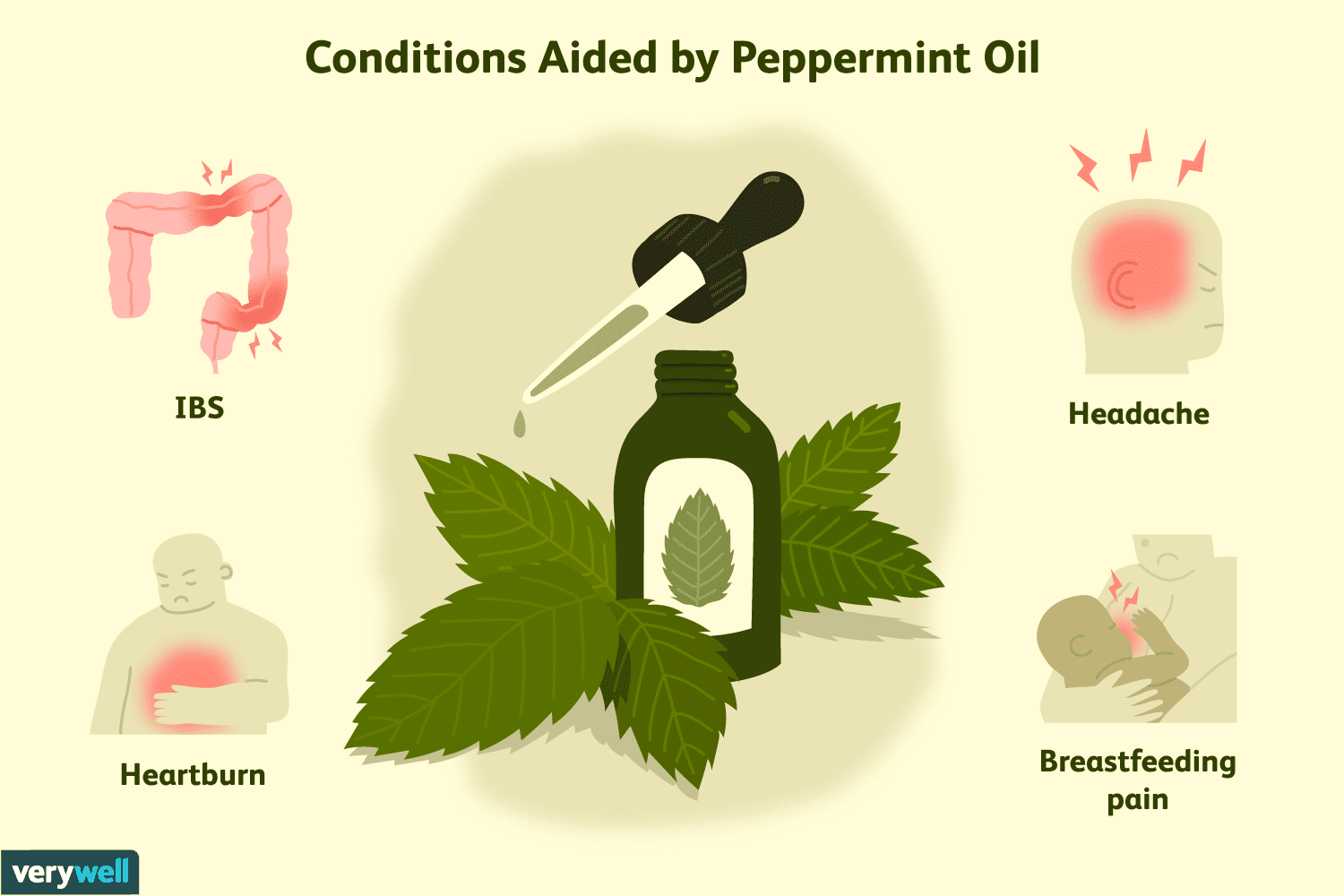 Die gesundheitlichen Vorteile von Pfefferminzöl
Pfefferminze (Mentha piperita ) wird häufig zum Würzen von Speisen verwendet, insbesondere von süßen Leckereien. Pfefferminzöl und -blätter wurden auch zur Behandlung einer Vielzahl von Erkrankungen v
Die gesundheitlichen Vorteile von Pfefferminzöl
Pfefferminze (Mentha piperita ) wird häufig zum Würzen von Speisen verwendet, insbesondere von süßen Leckereien. Pfefferminzöl und -blätter wurden auch zur Behandlung einer Vielzahl von Erkrankungen v
 Ray Manzarek stirbt an Gallengangskrebs
Venice Beach, Kalifornien, 1965. Durch Glück trifft Ray Manzarek auf Jim Morrison, einen ehemaligen Klassenkameraden der UCLA, und die Welt ist mit der Musik von The Doors gesegnet. Die Band verkauft
Ray Manzarek stirbt an Gallengangskrebs
Venice Beach, Kalifornien, 1965. Durch Glück trifft Ray Manzarek auf Jim Morrison, einen ehemaligen Klassenkameraden der UCLA, und die Welt ist mit der Musik von The Doors gesegnet. Die Band verkauft
 Ein Überblick über Achlorhydrie
Achlorhydrie ist das Fehlen von Salzsäure in den im Magen produzierten Magensäften. Der Zustand tritt im Allgemeinen nicht alleine auf, sondern ist eine sekundäre Folge eines anderen Zustand oder Um
Ein Überblick über Achlorhydrie
Achlorhydrie ist das Fehlen von Salzsäure in den im Magen produzierten Magensäften. Der Zustand tritt im Allgemeinen nicht alleine auf, sondern ist eine sekundäre Folge eines anderen Zustand oder Um