Titulo del articulo: Descubriendo múltiples tipos de metilación del ADN de bacterias y microbiomas individuales mediante secuenciación de nanoporos
Autor correspondiente: Colmillo de pandillas, Doctor
Línea de fondo:
Resultados: En este trabajo, Desarrollamos un nuevo método que permite la secuenciación de nanoporos para el descubrimiento de metilación de amplia aplicación. Lo aplicamos a bacterias individuales y al microbioma intestinal para un descubrimiento confiable de la metilación. Además, demostramos el uso de la metilación del ADN para el análisis de microbiomas de alta resolución, mapeo de elementos genéticos móviles con sus genomas de acogida directamente a partir de muestras de microbioma.
Cómo: Al examinar tres tipos de metilación del ADN en una gran diversidad de contextos de secuencia, observamos que la señal de secuenciación de nanoporos muestra una heterogeneidad compleja a través de eventos de metilación del mismo tipo. Para capturar esta complejidad y permitir la secuenciación de nanoporos para el descubrimiento de metilación de amplia aplicación, generamos un conjunto de datos de entrenamiento a partir de una variedad de especies bacterianas y desarrollamos un método novedoso que combina la identificación y el mapeo fino de las tres formas de metilación del ADN en un diseño de clasificación de múltiples etiquetas.
Evaluamos el método y luego lo aplicamos a bacterias individuales y microbioma intestinal de ratón para un descubrimiento confiable de la metilación. Además, Demostramos en el análisis del microbioma el uso de la metilación del ADN para agrupar contigs metagenómicos, asociar elementos genéticos móviles con sus genomas anfitriones, y por primera vez identificación de contigs metagenómicos mal ensamblados.
Dijo Gang Fang de Mount Sinai de la obra:
 ¿Qué detiene naturalmente la diarrea?
¿Qué detiene naturalmente la diarrea?
 ¡Cómo hacer que las zanahorias legales SCD sean más rápidas y nutritivas al mismo tiempo!
¡Cómo hacer que las zanahorias legales SCD sean más rápidas y nutritivas al mismo tiempo!
 Terapia conductual cognitiva (TCC) para aliviar los síntomas del SII
Terapia conductual cognitiva (TCC) para aliviar los síntomas del SII
 La presencia de ciertas bacterias intestinales en las madres podría proteger a los bebés de las alergias alimentarias
La presencia de ciertas bacterias intestinales en las madres podría proteger a los bebés de las alergias alimentarias
 La enfermedad de Crohn en niños puede comenzar a partir de bacterias
La enfermedad de Crohn en niños puede comenzar a partir de bacterias
 ¿Qué puede causar dolor pélvico en los hombres?
¿Qué puede causar dolor pélvico en los hombres?
 La verdad tóxica sobre los alimentos sin gluten y la enfermedad celíaca
“Lo que es comida para un hombre puede ser veneno feroz para otros.”—Lucretius Hoy en día, incluso las tiendas de comestibles más pequeñas de todo Estados Unidos están comenzando a vender alimentos
La verdad tóxica sobre los alimentos sin gluten y la enfermedad celíaca
“Lo que es comida para un hombre puede ser veneno feroz para otros.”—Lucretius Hoy en día, incluso las tiendas de comestibles más pequeñas de todo Estados Unidos están comenzando a vender alimentos
 ¿Por qué me duele el estómago por la noche?
¿Qué es el dolor de estómago? El dolor abdominal tipo cólico surge de la hiperactividad del peristaltismo intestinal normal, también conocido como contracciones musculares, y puede ser causado por
¿Por qué me duele el estómago por la noche?
¿Qué es el dolor de estómago? El dolor abdominal tipo cólico surge de la hiperactividad del peristaltismo intestinal normal, también conocido como contracciones musculares, y puede ser causado por
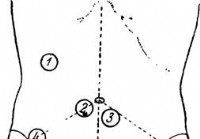 Investigación del estómago - Diagnóstico del abdomen agudo
Examinando, es necesario observar dos siguientes, aparentemente, las condiciones sin importancia:1) es bueno desnudar el vientre, habiendo levantado la camisa hasta los pezones y debajo la ropa blanca
Investigación del estómago - Diagnóstico del abdomen agudo
Examinando, es necesario observar dos siguientes, aparentemente, las condiciones sin importancia:1) es bueno desnudar el vientre, habiendo levantado la camisa hasta los pezones y debajo la ropa blanca