Genom szekvencia Helicobacter suis katalógusa támogatja annak szerepe a gyomor patológia
Abstract
Helicobacter
(H.
) suis
kapcsolatba hozták a krónikus gyomorhurut és fekélyek a pars oesophagea sertésekben , és a gyomorhurut, gyomorfekély-betegség és a gyomornyálkahártya-asszociált limfoid szövet limfóma emberben. Annak érdekében, hogy jobb betekintést részt vevő gének patogenitás és a specifikus alkalmazkodás a gyomor környezete H. suis katalógusa, egy genom analízist végeztünk két H. suis
izolált törzsek a gyomor nyálkahártya a sertések . Homológjait a túlnyomó többsége a gének kimutatták, hogy fontos a gyomor kolonizáció a humán patogén a H. pylori
detektáltunk a H. suis
genomban. H. suis
kódol több feltételezett külső membrán proteinek, amelyből a két hasonló a H. pylori
adhezinek HpaA és Horb. H. suis katalógusa kikötők szinte teljes fésű katalógusa IV-es típusú szekréciós rendszer tagjai és a IV-es típusú szekréciós rendszer 3, de hiányzik belőle a legtöbb gén jelen van a CAG katalógusa patogenitási sziget H. pylori katalógusa . Homológjait kódoló gének a H. pylori
neutrofil aktiváló fehérje és γ-glutamil transzpeptidáz azonosítottak H. suis katalógusa. H. suis
is van számos más feltételezett virulencia-asszociált gének, beleértve homológját mviN katalógusa, a H. pylori
flavodoxin gén, és egy homológját a H. pylori
vacuolating cytotoxin A gén. Arra a következtetésre jutottak, hogy bár a kódoló gének néhány fontos virulencia faktorok H. pylori katalógusa, mint például a citotoxin-asszociált protein (CagA), nem mutatható ki a H. suis
genom homológok más gének társuló kolonizáció és virulencia H. pylori katalógusa és más baktériumok is jelen vannak.
Bevezetés
Helicobacter
(H.
) suis
egy nagyon kényes, spirál alakú, Gram-negatív baktérium igénylő kétfázisú táptalaj pH = 5 dúsított borjúmagzatszérumot, és egy mikroaerob atmoszférában in vitro növekedését [1]. H. suis
colonizes a gyomorban több mint 60% vágósertés [1, 2]. Bár a pontos szerepe a H. suis
gyomor betegség sertésekben még mindig nem tisztázott, azt már társult krónikus gyomorhurut [3, 4] és fekélyek a pars oesophagea a gyomor [5-7]. Ez azt eredményezheti, jelentős gazdasági veszteségek miatt a hirtelen halál, csökkent takarmányfelvétel és csökkentett napi súlygyarapodás [8]. A csökkenés körülbelül 20 g /nap súlygyarapodást figyeltünk állatokban kísérletesen fertőzött H. suis katalógusa, míg a nem-fertőzött kontroll állatok [9].
Bakteriális gyomor rendellenességek emberekben elsősorban által okozott Helicobacter pylori
[10]. Azonban a nem-Helicobacter pylori
helicobacters (NHPh) szintén kapcsolatba hozták a humán gyomor betegség előfordulási közötti 0,2 és 6% [5]. H. suis
a leggyakoribb NHPh faj található az emberben, ahol eredetileg a neve "H. heilmannii
" Type 1 [11]. A jelek arra mutatnak, hogy a sertések szolgálhat a fertőzés forrása az emberre [5, 12]. Az emberi gazdaszervezetben, H. suis
kapcsolatba hozták a peptikus fekély [13], a gyomor nyálkahártyáját-asszociált limfoid szövet (MALT) lymphoma [14] és a krónikus gasztritisz [15]. Rágcsáló modell az emberi gyomor betegség, baktérium okoz súlyos gyulladást és MALT lymphoma-szerű elváltozások [16].
Eddig keveset tudunk patogenezisében H. suis katalógusa fertőzések. Jobb megértése érdekében a gének szerepet játszanak patogenitású, gyomor kolonizáció és fennmaradása H. suis katalógusa, a genomra kiterjedő összehasonlítása a jól vizsgált H. pylori katalógusa genom végeztük. Néhány virulencia faktorok valóban lehet hasonló mind a baktériumok. Ahogy ott is lehet különbség, eleve kommentárok a H. suis katalógusa genom végeztük is. Katalógusa Anyagok és módszerek katalógusa genomszekvenálási katalógusa A piroszekvenálás (454 Life Sciences Corporation, Branford, CT, USA) assay alkalmazták a genom típusú törzs H. suis katalógusa (HS1
T = LMG 23995 T = DSM 19735 T) és a H. suis
törzs 5 ( HS5) izolált gyomornyálkahártya két különböző sertés, módszer szerint által leírt Jan Baele et al. [1]. Minőségi szűrt szekvenciákat összeállítva contigs egy 454 Newbler assembler (Roche, Branford, CT, USA). Katalógusa Funkcionális annotáció katalógusa Annak érdekében, hogy a lehető legtöbb minőségi gén kommentárok, két különböző feliratozás megközelítést követtek: a határokon térképezés három Helicobacter pylori katalógusa törzsek (26695, Shi470, és G27 az NCBI számok NC_000915, NC_010698 és NC_011333-kal), és ab initio feliratozást. katalógusa Kereszt-mapping feliratozást
Egy egyedi BLAST [17 ] adatbázist hoztunk létre a HS1 T és HS5 genomi contigs. A H. pylori katalógusa proteomikai és nem kódoló RNS-eket igazodik (tblastn programja BLAST programcsomag, e küszöbérték beállítása, 10 -3) a H. suis katalógusa adatbázisban. Minden robbanás sújtotta a következő kiegészítő információkat elemezték: 1) (szekréció) szignál peptid hasítási helyet, ha jelen van, miként értékelik a SignaiP 3.0 programban [18, 19]; 2) előírásai transzmembránhélixek (szám, kezdő és véghelyzet feltételezett topológia tekintettel a sejtmembrán), ha vannak, miként értékelik a TMHMM programot [20]; 3) becslést a riboszóma kötési erőssége a mRNS régió megelőző legvalószínűbb startkodon. Riboszómakötő szilárdság becsülték alkalmazásával két megállapított tények: i) egy mRNS-szál, általában 20 nukleotid, mielőtt a tényleges start kodon, a reverz komplementere 5-7 nukleotidok közelében, a 16S rRNS 3'-végét működik, mint egy attraktor és pozícionáló a riboszomális kis alegység; ez a régió ismert, mint a Shine-Dalgarno-szekvencia [21, 22]; ii) Gram-negatív baktériumok egy AU-gazdag mRNS régió mintegy 16 nukleotid hosszú, és közvetlenül megelőző Shine-Dalgarno-szekvenciát is vonzó és helyzetét riboszómák, hogy segítsen kezdeményezi fordítását a helyes, biológiailag aktív génterméket [23, 24]. H. suis katalógusa, a Shine-Dalgarno-szekvenciát meghatároztuk, hogy egy alszekvenciája AGGAGGU (ami a fordított komplement, a 3 'végén a 16S rRNS), és a minimális AU-gazdagságát (egyenértékű a riboszóma kötési kapacitást ) az előző régió önkényesen állítva 10/16. Minden egyes elméleti ORF egy sor lehetséges startkodonok pontoztuk; Minél nagyobb a hasonlóság, hogy az ideális Shine-Dalgarno-szekvenciát, vagy az AU-Richer a megelőző régióban, vagy a jobb a kettő kombinációja, annál valószínűbb, hogy a potenciális startkodont az, hogy a tényleges startkodon.
Ab initio annotáció
az ab initio kommentár elméleti nyitott leolvasási keretet (ORF) elsőként határoztuk meg EMBOSS getorf szerszám (minimum ORF hosszát a 90 nukleotid, és az összes alternatív Start kodon véve) [25]. Minden ORF fordították később, és BLAST (bastp program) végeztük e küszöb 10 -15 ellen Uniprot-KB univerzális fehérje adatbázisban. A generalista algoritmusa getorf engedett durván tízszeres a várható természetes ORF, csökkentve annak kockázatát, hamis negatívok. Annak érdekében, hogy a fals pozitív arány alacsony, extra paramétereket figyelembe venni: 1) százalék közötti illesztés lekérdezés és a hit ORF; 2) százalékos hasonlóság vagy megőrzése között rendezi részei lekérdezés és hit ORF; 3) riboszóma kötő erő (további részleteket lásd fent). Jelenlétének meghatározására egy vagy több konzervált domént egy rpsblast keresés (alapértelmezett paraméter értékek) végeztük minden egyes elméleti ORF ellen a lefordított konzervált domén adatbázis, amely tartja fehérje domén nyomvonalakat több más adatbázis-források [26].
Eredmények
általános jellemzői H. suis katalógusa genom katalógusa a HS1 T genom összesen 1 635 292 bázispár és a HS5 genom 1 669 960 bp szekvenciáját, mind az átlagos GC tartalma 40%. Ezzel szemben a H. pylori katalógusa, csak egy példányt mind a 16S és 23S rRNS-gének volt kimutatható, de mint a H. pylori katalógusa, H. suis
három példányban a 5S rRNS gént. Harmincnyolc transzfer RNS azonosítottak. Az egész, 1266 ORF-ek HS1 T és 1257-re HS5 detektáltunk, amelyből 194 és 191 kódolt feltételezett proteineket rendre. A 98 és a 92 ORF-ek egy szignál peptid hasítási hely volt kimutatható, demonstrálva megjósolt szekretált fehérjék HS1 T és HS5 volt. A TMHMM programot megjósolt 210 és 206 fehérjék legalább egy transzmembrán hélix a HS1 T és HS5 volt. A sorrend frakció azonos HS1 T és HS5 a továbbiakban együtt írják le, mint a "H. suis katalógusa genom". Katalógusa gének esetleg részt gyomor kolonizáció és kitartás katalógusa homológjait H. pylori katalógusa részt vevő gének sav akklimatizáció, kemotaxist, tapadás gyomor hámsejtek, oxidatív stressz ellenállás (1. táblázat), és a motilitás detektáltunk a H. suis
genomban. Az utóbbi azonosították, mint egy ostoros Biosystem hasonló a H. pylori katalógusa [27]. Sőt, H. suis katalógusa tartalmazza fibrinonectin /fibrinogén-kötő proteint kódoló gén, de a megfelelő fehérje hiányzik a transzmembrán hélix vagy szignál peptid hasítási hely szerint a bioinformatikai eszközöket korábban említett. Homológjait kódoló CMP-N-acetil-neuraminsav-szintetázt (Neua) (HSUHS1_0474, HSUHS5_0481), sziálsav szintetáz (NeuB) (HSUHS1_0477, HSUHS5_0478) és UDP-N-acetil-glükózamin-2-epimeráz (WecB) (HSUHS1_1107, HSUHS5_0784) voltak megfigyelt well.Table 1 kapcsolatos gének pH homeosztázis, a kemotaxist, tapadás hámsejtek, és az oxidatív stressz ellenállást a genomjában H. suis katalógusa típusú törzs 1 (HS1T) és H. suis
törzs 5 (HS5 ).
Csoport Matton Gene kimutatható HS1T Matton Gene kimutatható HS5 Matton leírása homológ Matton aránya sorrendben rendezi (ebből% konzervált) a leírt homolog1 Matton pH homeosztázis katalógusa HSUHS1_0708 katalógusa HSUHS5_0286 katalógusa ureáz alegység alfa (karbamid katalógusa) H. heilmannii
100 (94) hotelben HSUHS1_0707 katalógusa HSUHS5_0285 katalógusa ureáz alegység béta (ureB katalógusa) H. heilmannii Matton 100 (94) hotelben HSUHS1_0706
HSUHS5_0284 katalógusa ureáz transzporter (UREI katalógusa) H. felis Matton 100 (89) hotelben HSUHS1_0705 katalógusa HSUHS5_0283 katalógusa ureáz kapcsolódó fehérje (ureE katalógusa) H . bizzozeronnii Matton 100 (84) hotelben HSUHS1_0704 katalógusa HSUHS5_0282 katalógusa ureáz kapcsolódó fehérje (ureF katalógusa) H. bizzozeronnii Matton 100 (84) hotelben HSUHS1_0702
HSUHS5_0280 katalógusa ureáz kapcsolódó fehérje (ureH katalógusa) H. bizzozeronnii Matton 96 (84) hotelben HSUHS1_0703 katalógusa HSUHS5_0281 katalógusa ureáz kapcsolódó fehérje (ureG katalógusa) H. bizzozeronnii Matton 100 (95) hotelben HSUHS1_0133 katalógusa HSUHS5_0547 katalógusa hidrogenáz kifejezés /formáció fehérje (hypA katalógusa) a H. pylori Matton 98 (83)
HSUHS1_0615
HSUHS5_0817 katalógusa hidrogenáz kifejezés /formáció fehérje (hypB katalógusa) a H. pylori Matton 99 (91) hotelben HSUHS1_0616 katalógusa HSUHS5_0816 katalógusa hidrogenáz kifejezés /formáció fehérje (hypC katalógusa) a H. pylori Matton 98 (89) hotelben HSUHS1_0617 katalógusa HSUHS5_0815 katalógusa hidrogenáz kifejezés /formáció fehérje (hypD katalógusa) H. achinonychis
katalógusa 98 (80) hotelben HSUHS1_0081 katalógusa HSUHS5_1197 katalógusa l-aszparagináz II (ansB katalógusa) a H. pylori Matton 98 (64) hotelben HSUHS1_0230 katalógusa HSUHS5_1130 katalógusa argináz (rocF katalógusa) a H. pylori Matton 99 (75) hotelben HSUHS1_0888 katalógusa HSUHS5_0231 katalógusa akrilamid amidohydrolase (Amie katalógusa) a H. pylori
katalógusa 100 (93) hotelben HSUHS1_0680 katalógusa HSUHS5_0265 katalógusa Formamidase (amiF katalógusa) a H. pylori Matton 100 (98) hotelben HSUHS1_0161 katalógusa HSUHS5_1077
α-karboanhidráz H. pylori Matton 92 (69) hotelben HSUHS1_0391 katalógusa HSUHS5_0874 katalógusa aszpartáz (aspA katalógusa) H. acinonychis
100 (89 ) hotelben chemotaxis katalógusa HSUHS1_1004 katalógusa HSUHS5_0649 katalógusa CHEA-MCP kölcsönhatás modulátorok H. pylori Matton 99 (79) hotelben HSUHS1_1003
- Kétfunkciós kemotaxis protein ( Chef katalógusa) a H. pylori Matton 82 (86) hotelben HSUHS1_1002 katalógusa HSUHS5_0775 katalógusa purin-kötő kemotaxis portein (Chew katalógusa) a H. pylori katalógusa
98 (91) hotelben HSUHS1_0538 katalógusa HSUHS5_0706 katalógusa Chemotaxis protein (Chev katalógusa) a H. pylori Matton 100 (92) hotelben HSUHS1_0846 katalógusa HSUHS5_0081 katalógusa vélt CHIPS-fehérje H. pylori Matton 100 (79) hotelben HSUHS1_0299 katalógusa HSUHS5_0250 katalógusa chemotaxis protein (CHEY katalógusa) a H. pylori Matton 100 (95)
HSUHS1_1001 katalógusa HSUHS5_0774 katalógusa Metil-elfogadó kemotaxis protein (tlpA katalógusa) a H. pylori Matton 100 (60) hotelben HSUHS1_0286 katalógusa HSUHS5_0256 katalógusa Metil-elfogadó kemotaxis protein (tlpB katalógusa) a H. pylori Matton 98 (63) hotelben HSUHS1_0479 katalógusa HSUHS5_0476 katalógusa Methyl- elfogadó CHIPS-fehérje H. acinonychis
100 (66 ) hotelben HSUHS1_0196 katalógusa HSUHS5_0122 katalógusa Methyl- elfogadó CHIPS-fehérje Campylobacter Upsaliensis katalógusa 2
 Az irritábilis bél szindróma (IBS) római kritériumai
Az irritábilis bél szindróma (IBS) római kritériumai
 Mi az a laparoszkópos bal oldali kolektómia/hemikolektómia?
Mi az a laparoszkópos bal oldali kolektómia/hemikolektómia?
 Milyen ételeket érdemes kerülni, ha divertikulitisze van?
Milyen ételeket érdemes kerülni, ha divertikulitisze van?
 Honnan tudhatja, hogy vesefertőzése van?
Honnan tudhatja, hogy vesefertőzése van?
 Zellerlé:jó a bélrendszerünknek vagy „Gotta Go”?
Zellerlé:jó a bélrendszerünknek vagy „Gotta Go”?
 Amit a gyógynövény-stimuláló hashajtókról tudni kell?
Amit a gyógynövény-stimuláló hashajtókról tudni kell?
 Szövetségesei a bélmikrobióta gondozásában:változatos étrend magas rosttartalommal
„Az öt szuperétel, amelyet meg kell enni az egészség megőrzéséhez”, „A kávé jót tesz a bélmikrobiótának. Ez az oka. A Kombucha tea lenyűgöző egészségügyi előnyei a bélrendszer egészségére. „Ez a diéta
Szövetségesei a bélmikrobióta gondozásában:változatos étrend magas rosttartalommal
„Az öt szuperétel, amelyet meg kell enni az egészség megőrzéséhez”, „A kávé jót tesz a bélmikrobiótának. Ez az oka. A Kombucha tea lenyűgöző egészségügyi előnyei a bélrendszer egészségére. „Ez a diéta
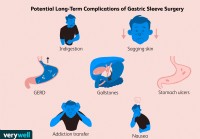 Hosszú távú szövődmények gyomorhüvely műtét után
Gastric sleeve műtét, más néven sleeve gastrectomia, egy súlycsökkentő műtét. A műtét során a sebész eltávolítja a gyomor körülbelül 80%-át. Ezt a műtétet általában laparoszkóposan végzik. Ez azt jele
Hosszú távú szövődmények gyomorhüvely műtét után
Gastric sleeve műtét, más néven sleeve gastrectomia, egy súlycsökkentő műtét. A műtét során a sebész eltávolítja a gyomor körülbelül 80%-át. Ezt a műtétet általában laparoszkóposan végzik. Ez azt jele
 A Zika könnyek között, izzadságon keresztül terjedhet:Jelentés
A fertőző betegségekről szóló legfrissebb hírek Az ókorban még a gazdagoknak is voltak parazitái A CDC a denevérekkel kapcsolatos veszettség növekedésére figyelmeztet E. Coli járvány 6 államban csoma
A Zika könnyek között, izzadságon keresztül terjedhet:Jelentés
A fertőző betegségekről szóló legfrissebb hírek Az ókorban még a gazdagoknak is voltak parazitái A CDC a denevérekkel kapcsolatos veszettség növekedésére figyelmeztet E. Coli járvány 6 államban csoma