gástrica in silico
análisis y verificación de la expresión génica en el cáncer gástrico S100
Resumen Antecedentes
la familia de proteínas S100 se compone de 22 miembros cuyas secuencias de proteínas abarcar al menos una mano EF Ca
2 + motivo de unión. Que estaban involucrados en la regulación de una serie de procesos celulares tales como progresión del ciclo celular y la diferenciación. Sin embargo, el estado de la expresión de los miembros de la familia S100 en el cáncer gástrico no se conoce todavía.
Métodos
combinado con el análisis de análisis de serie de la expresión génica, datos de transferencia de Northern y microarrays virtuales, los niveles de expresión de los miembros de la familia S100 en la normalidad y los tejidos malignos de estómago se investigaron sistemáticamente. La expresión de S100A3 se evaluó mediante RT-PCR cuantitativa.
: Resultados de la Al menos se encontraron 5 genes S100 que se upregulated en cado gástrica mediante el análisis in silico. Entre ellos, cuatro genes, incluyendo S100A2, S100A4, S100A7 y S100A10, se informó que se sobreexpresa en el cáncer gástrico anteriormente. se examinó la expresión de S100A3 en ochenta pacientes de cáncer gástrico. Los resultados mostraron que la media de niveles de expresión de S100A3 en los tejidos de cáncer gástrico fueron 2,5 veces tan alto como en los tejidos no tumorales adyacentes. S100A3 expresión se correlaciona con la diferenciación del tumor y TNM (tumor-nódulo-metástasis) etapa del cáncer gástrico, el cual fue relativamente altamente expresado en tejidos de cáncer gástrico pobremente diferenciados y avanzados (P Hotel < 0,05).
Conclusión
a nuestro entender este es el primer informe de evaluación sistemática de S100 expresiones de genes en el cáncer gástrico por múltiples análisis in silico. Los resultados indicaron que la sobreexpresión de genes de la familia S100 eran características de los cánceres gástricos y S100A3 podrían desempeñar un papel importante en la diferenciación y la progresión del cáncer gástrico.
Antecedentes
El cáncer gástrico es la segunda causa más común de muerte por cáncer en todo el mundo. Los factores ambientales y genéticos son importantes en la carcinogénesis gástrica [1, 2]. En las últimas dos décadas, se ha avanzado mucho en la identificación de genes implicados en el desarrollo de cáncer gástrico. Estos genes identificados son útiles en la comprensión de la patogénesis del cáncer gástrico y la definición de su firma molecular. También pueden servir como biomarcadores para el diagnóstico precoz y los objetivos para el desarrollo de fármacos.
Recientemente, la expresión génica a gran escala análisis han surgido como herramientas importantes para el cribado de genes relacionados con el cáncer [3]. Las dos tecnologías experimentales disponibles para el análisis de la expresión génica a gran escala son: análisis en serie 1) basada en la secuenciación del ADN de la expresión génica (SAGE) y enfoques etiqueta de secuencia expresada (EST) y 2) el análisis de microarrays basada dot-blot. infraestructuras de bioinformática múltiples se han establecido para compilar los datos derivados de estas técnicas. Entre ellos, el Proyecto del Genoma del Cáncer Anatomía (CGAP) y Gene Expression Omnibus (GEO) son dos importantes redes [4, 5]. aplicaciones anteriores de minería de datos utilizando el CGAP y recursos GEO han conducido a la identificación de varios genes novedosos o conocidos relacionados con el cáncer [6, 7].
La familia de proteínas S100 comprende 22 miembros cuyas secuencias de proteínas abarcar al menos una mano EF Ca2 + vinculante motivo [8]. proteínas S100 se localizan en el citoplasma y /o núcleo de una amplia gama de células, y participan en la regulación de una serie de procesos celulares tales como progresión del ciclo celular y la diferenciación. Diecisiete miembros de la familia S100 se encuentran como un grupo en el cromosoma 1q21-22, una región con frecuencia reorganizado en varios tumores. Además, el análisis molecular ha revelado que varios S100S, incluyendo S100A2, S100A4, S100A7 y S100A10, exhiben niveles de expresión alterados en el cáncer gástrico [9, 10]. Por lo tanto, es interesante investigar sistemáticamente la expresión de otros miembros de la familia S100 en tejidos normales y de cáncer gástrico.
En este estudio, hemos utilizado las bases de datos y enfoques analíticos disponibles de la Expresión Génica Omnibus y Anatomía del Proyecto del Genoma del Cáncer para analizar sistemáticamente la expresión de 22 genes S100 en tejidos normales y cancerosas de estómago. Los SAGE y microarrays de datos independientes fueron seleccionados para la S100 patrones de expresión génica. Nos presentó pruebas de que al menos cinco genes S100 se upregulated en el cáncer gástrico y además verificado experimentalmente la regulación al alza de S100A3 por RT-PCR cuantitativa.
Métodos de Análisis FODA SAGE SAGE
mide el número de etiquetas que representan la transcripción productos de un gen. Los datos producidos por la tecnología SAGE es una lista de variables con sus valores de recuento correspondientes. Todos los datos de SAGE recopilados y publicados en la página web GEO hasta enero de 2008, se utilizaron para el análisis de la expresión génica S100. Ambas etiquetas NlaIII y Sau3A de SAGEmap http:.... //Www NCBI NLM NIH gov /SAGE /fueron asignadas a UniGene http:... //Www NCBI NLM NIH . gov /UniGene /. Se adoptaron los grupos UniGene fiables adaptados a las etiquetas S100. Estas etiquetas de secuencias se utilizaron para determinar los niveles de expresión de 22 genes S100 en 2 normal y 8 bibliotecas de cáncer gástrico. Se proporcionó una lista de las etiquetas que se utilizan para el análisis en la Tabla 1 y la información detallada acerca de estas bibliotecas se encuentran disponibles en la página web GEO. Los análisis se realizaron mediante la comparación del número medio de etiquetas S100 en las bibliotecas de mucosa normal con el de las bibliotecas de cáncer gástrico. Diferencia de > 3 veces el cambio será considerado como positive.Table 1 SAGE análisis de los genes S100 expresión en bibliotecas normales y de cáncer gástrico
Gen
Unigene grupo
etiqueta SAGE
normal (TPM *) guía empresas cáncer (tpm*)
S100A2
516484
GATCTCTTGG
0.0
98.1
S100A3
433168
TCTCCCACAC
0.0
2.8
S100A4
81256
ATGTGTAACG
0.0
207.8
S100A6
275243
CCCCCTGGAT
245.9
865.9
S100A7
112408
GAGCAGCGCC
0.0
143.8
S100A8
416073
TACCTGCAGA
0.0
549.2
S100A9
112405
GTGGCCACGG
0.0
637.0
S100A10
143873
AGCAGATCAG
263.6
1890.5
S100A12
19413
GATTTTTAAA
0.0
13.9
S100A16
515714
AGCAGGAGCA
0.0
130.6
Sólo S100S que muestran las coincidencias positivas se muestran en esta tabla. * Etiquetas por millón;
virtual
del Norte En la base de datos del CGAP, análisis de transferencia Northern virtual permite a los investigadores ver la expresión de un gen específico en todas las bibliotecas de EST y SAGE [4]. A través de la herramienta de Gene http Finder:... //CGAP NCI NIH gov /Genes /GeneFinder, alguna información organizada sobre un gen en particular se puede encontrar mediante la consulta sea el identificador único gen o una palabra clave. Se incluyen en la información disponible son los patrones expresivos EST y SAGE vNorthern en todas las bibliotecas disponibles clasificadas de acuerdo con sus orígenes tisulares. Los datos recogidos en SAGE CGAP incluyen 5 bibliotecas de tejidos y 2 bibliotecas de xenoinjerto de cáncer gástrico, así como 3 bibliotecas normales de estómago, que eran parcialmente diferente de la recogida en GEO se ha mencionado anteriormente. S100 genes cuya expresión en las bibliotecas EST y SAGE de cáncer gástrico era a la vez > 3 veces tanto como los de estómago normal fueron designados como los positivos
Análisis de microarrays
En la actualidad, cinco conjuntos de datos de microarrays (Tabla 2) que contienen. los datos de los tejidos normales y de cáncer gástrico estaban disponibles en el sitio web GEO http: //www. NCBI NLM NIH gov /proyectos /geo /.... El conjunto de datos GSE2669, GSE2701 y GSE3438 aportado por Boussioutas A [11], Chen X [12] y Kim S [13], respectivamente, fueron elegidos para llevar a cabo el análisis de microarrays debido a que estos tres conjuntos de datos contienen relativamente más casos de cáncer normal y gástrica (10 normales y 64 cánceres de Boussioutas A et al; 22 normales y 90 cánceres de Chen X; 50 normales y 50 cánceres de Kim S). Más detalles acerca de las muestras y el análisis de microarrays se pueden encontrar en el sitio web del GEO. Diferencia se consideró significativa cuando p Hotel < 0.05. S100 genes cuya expresión se vio altamente en tres grupos de tejidos de cáncer gástrico fueron designados como positivos ones.Table 2 conjuntos de datos de microarrays de cáncer gástrico recogidos en la expresión génica página web ómnibus.
Casos
matriz
punto
Proveedor
|
normal
cáncer
|
|
|
GSE2637 página 3
55
ADNc página 13 k /17 K
Aggarwal A et al
GSE2685 página 8
22
oligo- nucleótido
~ 7.2 K
Hippo Y et al
GSE2669
10
64
ADNc
~ 7,4 K
Boussioutas A et al
GSE2701
22
90
ADNc
44 K
Chen X et al
GSE3438
50
50
ADNc página 14 K
Kim S et al
Recogida de tejido
Ochenta pacientes con cáncer gástrico que se sometió a una cirugía en nuestro hospital a partir de septiembre de 2004 a marzo de 2007 se inscribieron en este estudio. El tumor resecado y muestras de tejidos no tumorales adyacentes se congelaron inmediatamente en nitrógeno líquido y se mantuvieron a 70 ° C hasta la extracción de ARN. El diagnóstico tanto de cáncer gástrico y mucosa gástrica normal se demostró clínica y patológicamente. el sexo del paciente, la edad, el tamaño del tumor, el estadio TNM, la profundidad de la invasión de la pared, subtipo microscópica, y el estado de la metástasis de los ganglios linfáticos se obtuvieron de los registros quirúrgicos y patológicos. Los protocolos utilizados en este estudio fueron aprobados por el Comité de Protección de Sujetos Humanos del Hospital. Los pacientes que proporcionan tejido quirúrgico fresco para el estudio firmaron el consentimiento informado.
RT-PCR cuantitativa
Total RNA (mRNA) se extrajo de cáncer gástrico y los tejidos no tumorales adyacentes de acuerdo con las recomendaciones del fabricante del reactivo TRIzol (Invitrogen, Carlsbad , CA). 1 g de la muestra de ARN total se transcribió de forma inversa a ADN complementario (ADNc) con cebadores de oligo (dT). Los cebadores sentido y antisentido para S100A3 fueron diseñados de acuerdo con la secuencia de ARNm (número de acceso GenBank NM_002960.1). Se utilizó amplificado fragmentos de PCR que abarca los exones diferentes para evitar la amplificación de ADN genómico contaminado. El cebador sentido era 5'-GACCATCTGGTTCAGGTTCC-3 'y el cebador antisentido era 5'-ACATTCCCGAAACTCAGTCG-3'. Los productos de PCR fueron 200 pb de tamaño. La limpieza gen GAPDH se utilizó como control interno. El cebador sentido era 5'-CCAGGTGGTCTCCTCTGACTT-3 'y el cebador antisentido era 5'-GTTGCTGTAGCCAAATTCGTTGT-3'. Los productos de PCR fueron 130 pb de tamaño. México La curva estándar fue producido por la medición de la punto de cruce de cada valor estándar (6 veces ADNc diluidos en serie de músculo cardíaco, en los que el contenido de S100A3 estaba relativamente abundante) y trazado contra el valor logarítmico de las concentraciones. muestras de la curva estándar se incluyeron en cada ejecución. Cuantitativa en tiempo real RT-PCR se realizó utilizando un ABI PRISM 7000 Sequence Detection System (Applied Biosystems, Foster City, CA). La RT-PCR se llevó a cabo en un volumen total de 30 mL. La mezcla de reacción incluyó 1 x tampón, 200 mmol /L de trifosfatos de desoxi-ribonucleósido (dNTPs) (Invitrogen), 0,3 mol /L de cebadores sentido y antisentido, 1 U de Takara ExTaq Hotstart Taq (Takara Biotecnología), 0,6 l de 5 carboxi-x-rodamina (ROX) colorante de referencia, y 2 l de cDNA. El ciclo de PCR se realiza en 2 minutos a 95 ° C seguido de 40 ciclos de amplificación de desnaturalización a 94 ° C durante 30 segundos, hibridación a 58 ° C (para la detección de GAPDH) o 55 ° C (para la detección de S100A3) durante 30 segundos, y elongación a 72 ° C durante 1 minuto. La cuantificación relativa de ambos S100A3 y GAPDH se determinó por el método comparativo CT (ciclo térmico). Los valores de expresión S100A3 ARNm se normalizaron de acuerdo con la expresión de GAPDH. Cada ensayo se repitió tres veces para verificar los resultados, y se utilizó la relación entre el valor de la expresión del ARNm de tejidos de cáncer gástrico a los tejidos no tumorales adyacentes para su posterior análisis.
El análisis estadístico
Para las variables continuas, se expresaron los datos como el medio +/- SD. Los datos de expresión de genes S100 en microarrays de datos se recuperan y las diferencias en los niveles de expresión entre tejidos normales y de cáncer gástrico se determinaron por el estudiante de prueba T. La asociación entre la expresión ratios relativos de S100A3 y características clínicas se analizó mediante la prueba de Mann-Whitney. Se analizaron todos los datos utilizando el paquete de software SPSS11.0 (SPSS, Chicago, EE.UU.) y la diferencia se consideró significativa cuando p <
. 0.05: Resultados de la 1. SAGE y análisis virtual de transferencia Northern de la expresión de genes S100 en el cáncer gástrico
Hay 2 SAGE bibliotecas de mucosa gástrica normal y 8 SAGE bibliotecas de cáncer gástrico tejidos disponibles en el sitio web GEO (GSE545 y GSE14). Estas bibliotecas fueron proporcionados por dos laboratorios diferentes [14, 15]. Las etiquetas fiables de 20 genes S100 fueron extraídos de la página web SAGEmap y se utilizan para buscar los datos de SAGE. Se encontraron 10 genes para ser altamente expresado en tejidos de cáncer gástrico de acuerdo con los criterios de ajuste de > 3 veces la diferencia (Tabla 1). En estos 10 genes, sólo se S100A6 y S100A10 podrían ser detectables en los tejidos normales de la mucosa gástrica, que también tenían densidad media superior en el cáncer gástrico (865,9 y 1.890,5 TPM TPM, respectivamente). Los otros 8 genes, incluyendo S100A2, S100A3, S100A4, S100A7, S100A8, S100A9, S100A12 y S100A16, tenían diferente densidad media que van desde 2,8 a 637,0 TPM TPM, ninguno de los cuales se expresa en los tejidos normales de la mucosa gástrica. No hay diferencia significativa en la expresión entre los tejidos normales y cancerosos se puede conocer en s100a11, S100A14 y S100P (datos no mostrados). A continuación, utiliza transferencia de Northern virtual para analizar la expresión de genes S100 (Tabla 3). Seis genes fueron confirmados para ser aumentada en tejidos de cáncer gástrico. El positivo S100A6, S100A8, S100A16 y identificado por análisis de SAGE mostraron tener ninguna diferencia significativa en la expresión entre las bibliotecas normales y de cáncer gástrico cuando se realizan EST virtual Northern Blot. S100A13, no es detectable en las bibliotecas SAGE, ha demostrado ser altamente expresado en tejidos de cáncer gástrico por EST transferencia de Northern virtual. S100A3 y S100A12 no pudieron ser detectados por Northern blot.Table 3 análisis de Northern blot virtual Virtual de S100 genes de expresión en bibliotecas normales y de cáncer gástrico
|
etiquetas EST (TPM *)
etiquetas SAGE (TPM *) guía empresas |
Normal
Cancer
Normal
Cancer
S100A2
0.0
25.9
0.0
200.5
S100A3
0.0
0.0
0.0
0.0
S100A4
52.3
293.7
0.0
168.4
S100A7
0.0
0.0
0.0
304.8
S100A9
0.0
8.6
0.0
1339.4
S100A10
0.0
181.4
272.3
1483.7
S100A12
0
0
0
0
S100A13
0.0
43.2
0.0
0.0
Sólo S100S que muestran las coincidencias positivas se muestran en esta tabla. * Etiquetas por millón; se llevó a cabo el análisis
2. microarrays de expresión S100 genes en el cáncer gástrico
Análisis de microarrays para verificar aún más la sobreexpresión de los genes en el cáncer gástrico S100. 8, 11 y 7 S100 genes podrían ser encontrados en GSE2669, GSE2701 y GSE3438 conjuntos de datos, respectivamente (Tabla 4). Ambos genes S100A2 y S100A10, se mostró a ser upregulated en el cáncer gástrico de los tres conjuntos de datos. S100A3 se demostró que se sobreexpresa en el cáncer gástrico en un conjunto de datos GSE2669 y GSE2701, que no existía en el conjunto de datos GSE3438. S100A4, S100A6 y S100A7, se mostró a ser upregulated en el cáncer gástrico en un único conjunto de datos, pero no existen en los otros dos conjuntos de datos. Sin embargo, la expresión de S100A8 y S100A9 tenía ninguna diferencia significativa entre los tejidos normales y cancerosas en GSE2701 conjunto de datos. La tendencia expresión diferencial de S100A12 en GSE2701 era contraria a la del análisis SAGE (Tabla 1) .Tabla 4 Análisis de microarrays de genes S100 expresión en tejidos normales y de cáncer gástrico
|
GSE3438
GSE2669
GSE2701
|
normal *
cáncer *
p
normal *
cáncer *
p
normal *
cáncer *
P
S100A2
-0.22
0.00
0.00
1.03
1.35
0.01
-0.95
-0.36
0.00
S100A3
#
#
#
0.84
1.55
0.03
-0.14
0.05
0.03
S100A4
-0.23
0.30
0.00
#
#
#
#
#
#
S100A6
-0.43
0.27
0.00
#
#
#
#
#
#
S100A7
#
#
#
#
#
#
-1.00
-0.35
0.00
S100A8
#
#
#
0.32
0.69
0.01
0.66
0.70
0.81
S100A9
#
#
#
1.25
3.48
0.00
0.42
0.79
0.13
S100A10
-0.50
0.42
0.00
0.46
1.36
0.00
0.19
1.22
0.00
S100A12
#
#
#
#
#
#
0.50
0.21
0.01
Sólo S100S que muestran las coincidencias positivas se muestran en la tabla. * datos extraídos de microarrays de datos; No existen datos # en el conjunto de datos.
Tomados en conjunto, 5 genes se demostraron ser altamente expresado en el cáncer gástrico por los tres en los enfoques de análisis in silico. Eran S100A2, S100A3, S100A4, S100A7 y S100A10. Entre estos 5 genes, S100A3 fue el único que aún no se informó que estar relacionado con el cáncer gástrico anteriormente. A continuación se evaluó la expresión de S100A3 en tejidos de cáncer gástrico por RT-PCR cuantitativa.
3. Verificación de S100A3 sobreexpresión en cáncer gástrico por RT-PCR cuantitativa
Para investigar si S100A3 se sobreexpresa en el cáncer gástrico, se analizó la la expresión de ARNm de S100A3 en tejidos de cáncer gástrico y los correspondientes tejidos no tumorales adyacentes de 80 pacientes por RT-PCR cuantitativa. El nivel de expresión relativamente media de S100A3 en el cáncer gástrico fue 2,52 ± 1,45 en comparación con los tejidos no tumorales adyacentes (p
= 0,01). Se analizaron las correlaciones más de las expresiones S100A3 ARNm con las características clínicas. Los resultados mostraron que la expresión de S100A3 ARNm no se correlacionó con el género, la edad, el tamaño del tumor, la profundidad de invasión de la pared, los subtipos microscópicas o metástasis de ganglios linfáticos con una estadística de p > 0,05 en cada parámetro (Tabla 5). Sin embargo, se encontró que la expresión de S100A3 ARNm se correlaciona con la diferenciación del tumor y el estadio del tumor-nódulo-metástasis. Los niveles de expresión S100A3 en los tejidos tumorales bienestar y diferenciado moderados fueron significativamente menor que en los pobremente diferenciados (p
y lt; 0,05). expresión S100A3 en el estadio TNM I y II también fue menor que en la etapa III y IV (p
= 0,04). Todos estos datos demuestran que S100A3 se sobreexpresa en las muestras de cáncer gástrico y podría estar relacionado con la diferenciación y el desarrollo de gástrico cancer.Table 5 Correlación de la expresión de S100A3 ARNm con parámetros clinicalpathological en pacientes con carcinoma gástrico.
variable
la expresión del ARNm
P
|
Número
N /T > 4
La N /T 2-4
N /T 1-2
N /T < 1 |
|
Sexo Masculino
61 página 11
34 página 9 página 8
0,17
Mujer página 19 página 3
5 página 8
2 Edad (a) Hotel < 65
54
10
31 página 6 página 7
0,08
> 65
26
4 de 8 página 11 página 3
tumor diferenciación
Well1
3
0
0
1
2
0.22a
Moderate2
33
8
7
12
6
0.03b
Poor3
44
6
32
4
2
0.01c
El tamaño del tumor (cm) Hotel < 5.0
41 página 6
23 página 5 página 7
0,96 Hotel > 5.0
39 página 8
16 página 12 página 3
La profundidad de la invasión de la pared mucosa
, submucosa1 página 6
2 1
2 1 | 0.45A
Muscular propria2
18 página 6 página 8
4 de 0
0.33b
subserosa, serosa3
56
10
34
6 página 6
0.72c
Etapa
I + II página 19
2 7 página 5 página 5
0,04
III + IV
61 página 12
32 página 12 página 5
subtipos microscópicos
Intestinal1
57
10
29
11
7
0.87a
Diffuse2
18
4
8
4
2
0.26b
Atypical3
5
0
2
2
1
0.21c
Metástasis ganglionares
Sí
64 página 11
35 página 11 página 7
0,16
Sin
16 página 3 página 4
6 página 3
aP
: 1 VS página 2; bp
: 2 VS Estrellas: 3; cP
: 1 VS
3. Discusión
Aunque miembros de la familia S100 tienen una estructura común y se localizan principalmente en una región específica del cromosoma 1, todos ellos tienen patrones de expresión muy únicos en normal o patológico tejidos. Hemos investigado sistemáticamente la expresión de miembros de la familia S100 en tejidos de cáncer gástrico mediante la combinación de análisis de SAGE, datos de transferencia de Northern y microarrays virtuales. Se ha informado de que los errores de hibridación cruzada pueden ocurrir en el análisis de microarrays, cuando la similitud de secuencia superior a 75%. Sin embargo, el mRNA de similitud de secuencias de miembros de la familia S100 fue del 4% -67%, por lo que la posibilidad de hibridación cruzada de genes S100 en todos los tres chips analizados en el presente estudio sería relativamente pequeño. Por otra parte, Tanto la tecnología SAGE y EST transferencia de Northern virtuales se basan en la secuenciación del ADN y se cree que son métodos fiables de evaluación de la expresión génica. La combinación de múltiples bases de datos y enfoques analíticos mencionados anteriormente, la posibilidad de artefactos se reduciría en gran medida. En el presente estudio, 5 genes S100 se demostraron ser upregulated en el cáncer gástrico mediante la combinación de análisis, entre los que se había informado anteriormente 4 genes, lo que indica la validez de la estrategia de análisis in silico se utilizó en este trabajo y la posible importancia del papel de los genes S100 en desarrollo y progresión de cáncer gástrico.
en los últimos años, muchos miembros de la familia S100 se ha demostrado que ser regulados diferencialmente en diversos tumores malignos. Aunque los mecanismos de acción de S100S y las implicaciones funcionales de su expresión alterada se mantuvieron a determinar, varios estudios han demostrado que la sobreexpresión de proteínas S100 muestra grandes implicaciones clínicas para el diagnóstico y la estadificación de los tumores humanos, así como para la predicción de pronóstico.
se ha informado de que S100A2 fue altamente expresado en el cáncer no microcítico de pulmón, carcinoma de células escamosas de esófago, carcinoma de células escamosas de laringe, ovario carcinomas papilares serosos, así como cáncer gástrico. S100A2 también ha demostrado ser un predictor de metástasis a distancia y la tasa de supervivencia en las primeras etapas del cáncer no microcítico de pulmón de células [16]. S100A4 se encontró que se sobreexpresa en el cáncer de vejiga y podría ser servido como un predictor de la progresión del tumor [17]. Muchos estudios han demostrado que S100A7 tenía una expresión aumentada en el cáncer de mama. La sobreexpresión de S100A7 estaba relacionado con estadios TNM más altas de cáncer de mama y de cáncer de mama invasivo del receptor de estrógeno negativo, la expresión de S100A7 estaba asociada con un peor pronóstico [18]. S100A7 sobreexpresión también se asoció con un aumento de la malignidad del cáncer de mama, lo que puede ocurrir a través de la estimulación de la actividad Jab1. Además, S100A10 fue identificado como un gen upregulated en no pequeñas de cáncer de pulmón de células escamosas y carcinoma esofágico de células escamosas de la tecnología de microarrays. Todos estos 4 genes S100 también mostraron ser upregulaetd en los cánceres gástricos anteriormente [9, 10], lo que fue confirmado en el presente trabajo.
S100 genes pueden ser regulados a la baja en algunos otros tipos de cáncer. Por ejemplo, S100A6 se expresó humilde de los cánceres de próstata, que podrían estar relacionados con la hipermetilación del promotor S100A6. Otro ejemplo es S100A2. Tenía una expresión reducida en la próstata y el cáncer oral y fue considerado como un gen supresor tumoral potencial. S100A2 puede interactuar con C terminal de p53 y luego mejorar la actividad transcripcional de p53. Esta interacción p53-S100A2 dependiente del ciclo celular podría mediar en el efecto inhibidor de S100A2 en el cáncer. Estos resultados sugieren que genes S100 pueden desempeñar diferentes funciones durante el desarrollo de diferentes tipos de cáncer.
S100A3, una proteína correlacionado con el desarrollo de folículo piloso, se había demostrado que se sobreexpresa en los tumores. Por ejemplo, los niveles de expresión de las proteínas S100A3 diferían notablemente en el tejido del tumor astrocítico en relación con los tipos de tumores y grados [19]. El presente trabajo se confirmó por primera vez que S100A3 se reguló en el cáncer gástrico y asociado a la diferenciación de los pobres y superior etapa TNM de células de cáncer gástrico. Sin embargo, el papel de S100A3 en la diferenciación y la progresión del cáncer gástrico es necesario investigar más a fondo.
Conclusión
Nuestro trabajo sugiere que el análisis in silico es una estrategia válida para el descubrimiento de genes expresados diferencialmente en el cáncer gástrico, y era un S100A3 nuevo gen sobreexpresado en células de cáncer gástrico y podría desempeñar un papel importante durante la diferenciación y la progresión del cáncer gástrico.
Notas
Ji Liu, Li Xue, Guang Dong-Long contribuyeron igualmente a este trabajo.
abreviaciones
SAGE:
serie Análisis de la Expresión génica
EST:
etiqueta de secuencia expresada
CGAP:
Proyecto del Genoma del cáncer Anatomía
GEO:
de gene Expression Omnibus
RT-PCR: Read Transcripción inversa reacción de polimerasa en cadena .
Declaraciones
Agradecimientos
Este trabajo fue apoyado por la Fundación Nacional de Ciencias Naturales de China (Grant No. 30670970). Estamos muy agradecidos a Yanglin Pan para una excelente guía en el proceso de experimento.
Conflicto de intereses
Los autores declaran que no tienen intereses en competencia.
|  Aspectos destacados y conclusiones clave de la reunión de bacterias de Boston de 2019 (BBM)
Aspectos destacados y conclusiones clave de la reunión de bacterias de Boston de 2019 (BBM)
 Los investigadores manipulan especies bacterianas en el intestino utilizando la dieta
Los investigadores manipulan especies bacterianas en el intestino utilizando la dieta
 Modificar genéticamente las bacterias intestinales reduce el riesgo de cáncer colorrectal en ratones, según un estudio
Modificar genéticamente las bacterias intestinales reduce el riesgo de cáncer colorrectal en ratones, según un estudio
 Los científicos convierten la sangre tipo A en tipo O universal,
Los científicos convierten la sangre tipo A en tipo O universal,
 La investigación dice que en la infección canina SARS-CoV-2,
La investigación dice que en la infección canina SARS-CoV-2,
 Científico demuestra el papel del microbioma en la obesidad
Científico demuestra el papel del microbioma en la obesidad
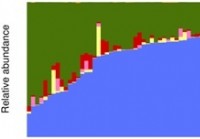 El estudio describe la base de datos inicial del microbioma intestinal sano y el perfil de abundancia
En un estudio publicado el 11 de septiembre, se describe una base de datos inicial de microbioma intestinal sano y un perfil de abundancia. 2019 en la revista de acceso abierto PLOS ONE de Charles Had
El estudio describe la base de datos inicial del microbioma intestinal sano y el perfil de abundancia
En un estudio publicado el 11 de septiembre, se describe una base de datos inicial de microbioma intestinal sano y un perfil de abundancia. 2019 en la revista de acceso abierto PLOS ONE de Charles Had
 La sangre joven devuelve la vitalidad a los ancianos
El Drácula de Bram Stoker sobrevivió gracias a la sangre de jóvenes doncellas. ¡Ahora los investigadores han descubierto que puede haber algo de verdad en esta extraña teoría! Según un genetista del
La sangre joven devuelve la vitalidad a los ancianos
El Drácula de Bram Stoker sobrevivió gracias a la sangre de jóvenes doncellas. ¡Ahora los investigadores han descubierto que puede haber algo de verdad en esta extraña teoría! Según un genetista del
 Nanotecnología y diagnóstico y tratamiento de COVID-19
El desarrollo de nanobiosensores y vacunas y medicamentos basados en nanopartículas ha abierto un nuevo camino hacia una mejor gestión de la pandemia de la enfermedad por coronavirus 2019 (COVID-19)
Nanotecnología y diagnóstico y tratamiento de COVID-19
El desarrollo de nanobiosensores y vacunas y medicamentos basados en nanopartículas ha abierto un nuevo camino hacia una mejor gestión de la pandemia de la enfermedad por coronavirus 2019 (COVID-19)