Os pesquisadores compararam o microbioma nasal de pacientes com doença coronavírus 2019 (COVID-19), indivíduos saudáveis, e profissionais de saúde. Esses estudos indicaram um aumento no patógeno
Pseudomonas aeruginosa presente no microbioma nasal de pacientes COVID-19, que pode ser responsável por outras infecções secundárias.
 Estudo:A infecção aguda por SARS-CoV-2 está associada a uma expansão de bactérias patogênicas no nariz, incluindo Pseudomonas Aeruginosa. Crédito da imagem:Christoph Burgstedt / Shutterstock
Estudo:A infecção aguda por SARS-CoV-2 está associada a uma expansão de bactérias patogênicas no nariz, incluindo Pseudomonas Aeruginosa. Crédito da imagem:Christoph Burgstedt / Shutterstock Introdução
p A síndrome respiratória aguda grave coronavírus 2 (SARS-CoV-2) se espalha principalmente por inalação de partículas virais transportadas pelo ar. Acredita-se que o principal local de replicação viral inicial seja o nariz, ao invés da boca, devido à maior expressão do receptor da enzima conversora de angiotensina 2 (ACE2) no nariz.
p A rápida replicação viral no trato respiratório superior pode causar infecção no trato respiratório inferior e doença grave. Contudo, muitos estudos não mostraram nenhuma correlação entre a gravidade da doença e a carga viral no nariz, sugerindo que pode haver outros fatores que contribuem para a gravidade da doença.
p Vários estudos sugeriram que o microbioma no nariz e na garganta pode desempenhar um papel nas infecções virais. A infecção viral pode perturbar a microbiota e levar à coinfecção, que pode piorar a inflamação e interromper a resposta imunológica. A infecção grave também pode levar à perda do olfato e do paladar. Contudo, existem apenas alguns estudos que investigaram o microbioma respiratório em pacientes com COVID-19.
p Em um novo estudo publicado no
bioRxiv * servidor de pré-impressão, pesquisadores da Universidade da Califórnia em Irvine investigaram o efeito da infecção por SARS-CoV-2 no microbioma nasal.
Comparando microbiomas nasais
p A equipe de pesquisadores usou o sequenciamento de amplicon do gene do ácido ribossômico ribossômico 16S (rRNA) para determinar se a infecção por SARS-CoV-2 levou a uma mudança na composição do microbioma nasal. Este experimento descobriu que o microbioma nasal de pacientes COVID-19 era, na verdade, diferente em comparação com aqueles que não tinham a doença, bem como o que estava presente no nariz dos trabalhadores da saúde. O microbioma nasal de pacientes com COVID-19 e profissionais de saúde era mais rico do que não-pacientes; Contudo, os microbiomas de todos os três tipos de amostra foram dominados por alguns micróbios selecionados.
p O microbioma nasal de uma pessoa saudável é composto principalmente de
Corynebacterium, Staphylococcus, Streptococcus, Dolosigranulum, e
Moraxella. Os autores descobriram que muitas bactérias patogênicas, tal como
Rothia ,
Acinetobacter , e
Pseudomonas, eram comuns em pacientes COVID-19, com níveis particularmente altos de
Pseudomonas aeruginosa relatados nesses pacientes.
p Estudos têm demonstrado que infecções virais agudas podem alterar o microbioma nasal e favorecer o aumento de bactérias patogênicas. Um aumento em
Pseudomonas também foi relatado em casos de gripe, o que sugere que sua presença pode não ser específica para COVID-19, mas sim uma resposta geral a processos inflamatórios.
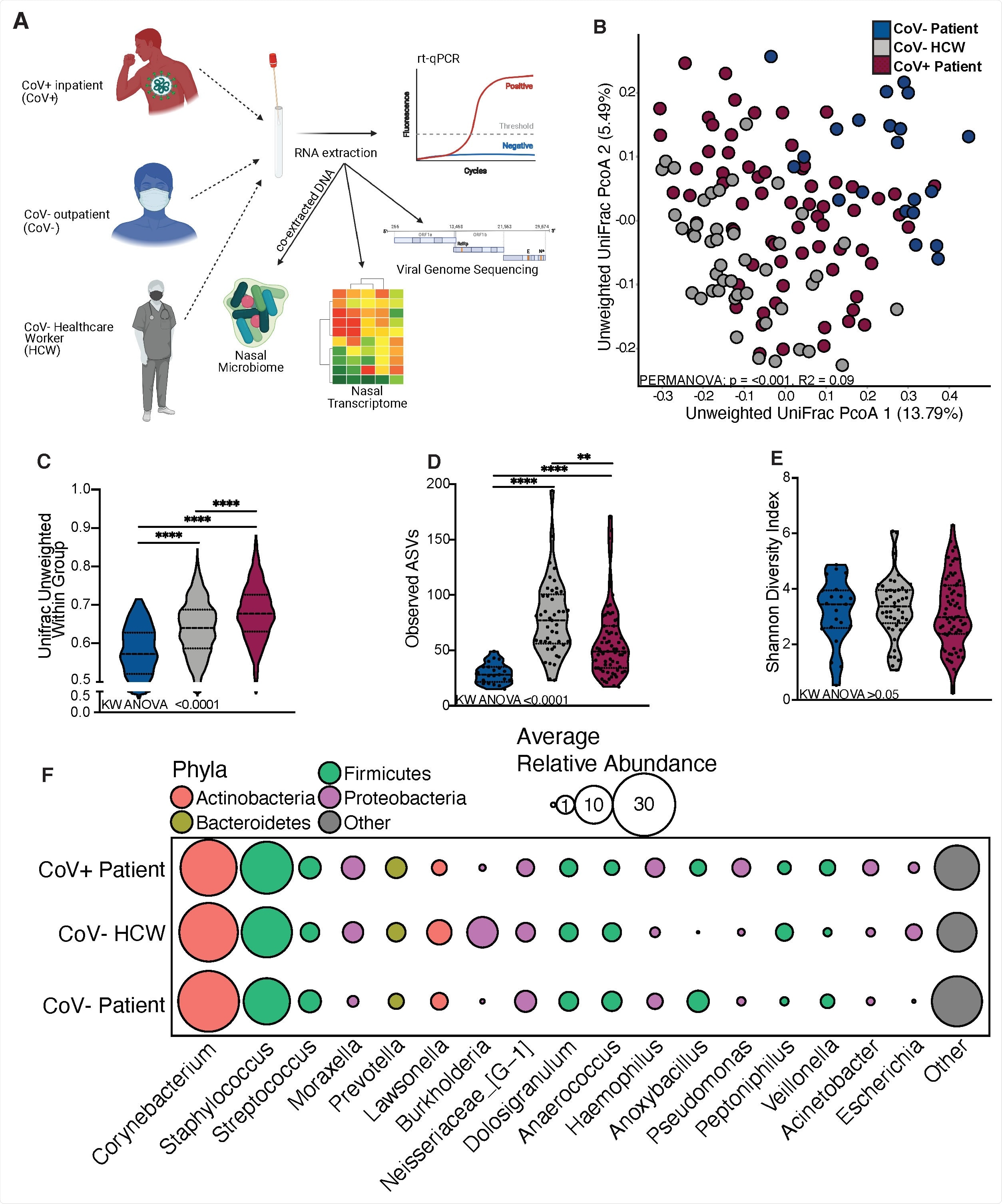 O microbioma nasal de pacientes infectados com SARS-CoV-2 é distinto. (A) Esquema do projeto do estudo. (B) Análise de coordenadas principais de comunidades microbianas nasais, distância UniFrac não ponderada colorida pelo status do hospedeiro. A contribuição do status do hospedeiro para a variância total nas matrizes de dissimilaridade UniFrac não ponderadas foi medida usando PERMANOVA (Adonis com 10, 000 permutações). (C, D, E) Gráfico de violino ilustrando (D) distâncias UniFrac médias não ponderadas (C) número de variantes de sequenciamento de amplicons observadas e (D) diversidade de shannon (E) dividido por status de hospedeiro. A significância para os painéis C-E foi determinada usando ANOVA não paramétrica de Kruskal Wallis (valores p inseridos na parte inferior de cada painel), com comparação múltipla de Dunn * =p <0,05, ** =p <0,01, *** =p <0,001, **** =p <0,0001. (F) Gráficos de bolhas de gêneros bacterianos encontrados em abundância média superior a 1% em toda a população de estudo ordenada da esquerda para a direita em abundância média descendente. O tamanho de cada círculo indica a abundância relativa média para cada taxa no grupo denotado e a cor de cada círculo indica a qual Fila bacteriana cada gênero pertence.
O microbioma nasal de pacientes infectados com SARS-CoV-2 é distinto. (A) Esquema do projeto do estudo. (B) Análise de coordenadas principais de comunidades microbianas nasais, distância UniFrac não ponderada colorida pelo status do hospedeiro. A contribuição do status do hospedeiro para a variância total nas matrizes de dissimilaridade UniFrac não ponderadas foi medida usando PERMANOVA (Adonis com 10, 000 permutações). (C, D, E) Gráfico de violino ilustrando (D) distâncias UniFrac médias não ponderadas (C) número de variantes de sequenciamento de amplicons observadas e (D) diversidade de shannon (E) dividido por status de hospedeiro. A significância para os painéis C-E foi determinada usando ANOVA não paramétrica de Kruskal Wallis (valores p inseridos na parte inferior de cada painel), com comparação múltipla de Dunn * =p <0,05, ** =p <0,01, *** =p <0,001, **** =p <0,0001. (F) Gráficos de bolhas de gêneros bacterianos encontrados em abundância média superior a 1% em toda a população de estudo ordenada da esquerda para a direita em abundância média descendente. O tamanho de cada círculo indica a abundância relativa média para cada taxa no grupo denotado e a cor de cada círculo indica a qual Fila bacteriana cada gênero pertence. p Esses resultados correspondem aos achados de infecções bacterianas secundárias em pacientes com COVID-19. A composição do microbioma nasal pode, portanto, funcionar como o tipo de amostra primária para avaliar o risco de uma pessoa de infecções secundárias após sua recuperação de certas infecções virais.
p Adicionalmente, o grupo de pesquisa com sede na Califórnia também descobriu que as cargas virais no nariz não afetaram a diversidade do microbioma nasal. Pacientes com COVID-19 com cargas virais baixas apresentaram uma quantidade mais significativa de
Estreptococo em seu microbioma nasal
, enquanto os pacientes COVID-19 com cargas virais moderadas tiveram uma quantidade maior de
Corynebacterium . Em terceiro lugar, Pacientes com COVID-19 com altas cargas virais apresentaram maiores níveis de
Cutibacterium, Neisseria, e Pseudomonas espécies presentes em seus microbiomas nasais.
p Os autores então compararam os transcriptomas nasais dos sujeitos de teste e encontraram 692 genes expressos de forma diferente entre pacientes com COVID-19 e profissionais de saúde. Os genes regulados positivamente em indivíduos COVID-19-positivos foram associados às vias de defesa do hospedeiro. Os genes que estão associados à morte celular, bem como aqueles que codificam para receptores inibitórios, também foram regulados positivamente em pacientes COVID-19.
p Comparativamente, alguns dos genes em COVID-19 que foram considerados regulados negativamente incluíram aqueles que estão associados à manutenção de condições internas consistentes, organização celular, e processamento de tecido neuronal. Além disso, os genes responsáveis pela produção de mucina nas vias nasais, bem como aqueles que influenciam os órgãos sensoriais, também foram considerados regulados para baixo. Tomados em conjunto, esses achados podem explicar a perda de olfato e paladar amplamente relatada em pacientes com COVID-19.
Aumento de bactérias patogênicas
p As infecções adquiridas em hospitais continuam sendo um grande desafio no ambiente clínico. Essas infecções geralmente surgem devido a ambientes hospitalares e profissionais de saúde contaminados. Por causa de sua longa exposição em hospitais, profissionais de saúde são portadores comuns de micróbios patogênicos.
p Os autores encontraram níveis mais elevados de patógenos como
Escerichia, Klebsiella, e
Burkholderia no microbioma nasal de profissionais de saúde. A abundância de
Acinetobacter foi considerado maior em pacientes com COVID-19 e profissionais de saúde, que pode ser devido a uma possível transferência entre os dois.
Conclusão
p Os resultados do presente estudo indicam uma mudança no microbioma nasal de pessoas infectadas com SARS-CoV-2, com um aumento de patógenos como
Pseudomonas aeruginosa . Uma limitação deste estudo foi o fato de que apenas o momento foi avaliado; Portanto, estudos futuros que comparem a composição do microbioma nasal de pacientes com COVID-19, indivíduos saudáveis, e o hospital ao longo do tempo pode melhorar a compreensão de como o microbioma nasal muda.
*Notícia importante
p
bioRxiv publica relatórios científicos preliminares que não são revisados por pares e, Portanto, não deve ser considerado conclusivo, orientar a prática clínica / comportamento relacionado à saúde, ou tratadas como informações estabelecidas.
 Estudo:A infecção aguda por SARS-CoV-2 está associada a uma expansão de bactérias patogênicas no nariz, incluindo Pseudomonas Aeruginosa. Crédito da imagem:Christoph Burgstedt / Shutterstock
Estudo:A infecção aguda por SARS-CoV-2 está associada a uma expansão de bactérias patogênicas no nariz, incluindo Pseudomonas Aeruginosa. Crédito da imagem:Christoph Burgstedt / Shutterstock  O microbioma nasal de pacientes infectados com SARS-CoV-2 é distinto. (A) Esquema do projeto do estudo. (B) Análise de coordenadas principais de comunidades microbianas nasais, distância UniFrac não ponderada colorida pelo status do hospedeiro. A contribuição do status do hospedeiro para a variância total nas matrizes de dissimilaridade UniFrac não ponderadas foi medida usando PERMANOVA (Adonis com 10, 000 permutações). (C, D, E) Gráfico de violino ilustrando (D) distâncias UniFrac médias não ponderadas (C) número de variantes de sequenciamento de amplicons observadas e (D) diversidade de shannon (E) dividido por status de hospedeiro. A significância para os painéis C-E foi determinada usando ANOVA não paramétrica de Kruskal Wallis (valores p inseridos na parte inferior de cada painel), com comparação múltipla de Dunn * =p <0,05, ** =p <0,01, *** =p <0,001, **** =p <0,0001. (F) Gráficos de bolhas de gêneros bacterianos encontrados em abundância média superior a 1% em toda a população de estudo ordenada da esquerda para a direita em abundância média descendente. O tamanho de cada círculo indica a abundância relativa média para cada taxa no grupo denotado e a cor de cada círculo indica a qual Fila bacteriana cada gênero pertence. p Esses resultados correspondem aos achados de infecções bacterianas secundárias em pacientes com COVID-19. A composição do microbioma nasal pode, portanto, funcionar como o tipo de amostra primária para avaliar o risco de uma pessoa de infecções secundárias após sua recuperação de certas infecções virais.
p Adicionalmente, o grupo de pesquisa com sede na Califórnia também descobriu que as cargas virais no nariz não afetaram a diversidade do microbioma nasal. Pacientes com COVID-19 com cargas virais baixas apresentaram uma quantidade mais significativa de Estreptococo em seu microbioma nasal , enquanto os pacientes COVID-19 com cargas virais moderadas tiveram uma quantidade maior de Corynebacterium . Em terceiro lugar, Pacientes com COVID-19 com altas cargas virais apresentaram maiores níveis de Cutibacterium, Neisseria, e Pseudomonas espécies presentes em seus microbiomas nasais.
p Os autores então compararam os transcriptomas nasais dos sujeitos de teste e encontraram 692 genes expressos de forma diferente entre pacientes com COVID-19 e profissionais de saúde. Os genes regulados positivamente em indivíduos COVID-19-positivos foram associados às vias de defesa do hospedeiro. Os genes que estão associados à morte celular, bem como aqueles que codificam para receptores inibitórios, também foram regulados positivamente em pacientes COVID-19.
p Comparativamente, alguns dos genes em COVID-19 que foram considerados regulados negativamente incluíram aqueles que estão associados à manutenção de condições internas consistentes, organização celular, e processamento de tecido neuronal. Além disso, os genes responsáveis pela produção de mucina nas vias nasais, bem como aqueles que influenciam os órgãos sensoriais, também foram considerados regulados para baixo. Tomados em conjunto, esses achados podem explicar a perda de olfato e paladar amplamente relatada em pacientes com COVID-19.
O microbioma nasal de pacientes infectados com SARS-CoV-2 é distinto. (A) Esquema do projeto do estudo. (B) Análise de coordenadas principais de comunidades microbianas nasais, distância UniFrac não ponderada colorida pelo status do hospedeiro. A contribuição do status do hospedeiro para a variância total nas matrizes de dissimilaridade UniFrac não ponderadas foi medida usando PERMANOVA (Adonis com 10, 000 permutações). (C, D, E) Gráfico de violino ilustrando (D) distâncias UniFrac médias não ponderadas (C) número de variantes de sequenciamento de amplicons observadas e (D) diversidade de shannon (E) dividido por status de hospedeiro. A significância para os painéis C-E foi determinada usando ANOVA não paramétrica de Kruskal Wallis (valores p inseridos na parte inferior de cada painel), com comparação múltipla de Dunn * =p <0,05, ** =p <0,01, *** =p <0,001, **** =p <0,0001. (F) Gráficos de bolhas de gêneros bacterianos encontrados em abundância média superior a 1% em toda a população de estudo ordenada da esquerda para a direita em abundância média descendente. O tamanho de cada círculo indica a abundância relativa média para cada taxa no grupo denotado e a cor de cada círculo indica a qual Fila bacteriana cada gênero pertence. p Esses resultados correspondem aos achados de infecções bacterianas secundárias em pacientes com COVID-19. A composição do microbioma nasal pode, portanto, funcionar como o tipo de amostra primária para avaliar o risco de uma pessoa de infecções secundárias após sua recuperação de certas infecções virais.
p Adicionalmente, o grupo de pesquisa com sede na Califórnia também descobriu que as cargas virais no nariz não afetaram a diversidade do microbioma nasal. Pacientes com COVID-19 com cargas virais baixas apresentaram uma quantidade mais significativa de Estreptococo em seu microbioma nasal , enquanto os pacientes COVID-19 com cargas virais moderadas tiveram uma quantidade maior de Corynebacterium . Em terceiro lugar, Pacientes com COVID-19 com altas cargas virais apresentaram maiores níveis de Cutibacterium, Neisseria, e Pseudomonas espécies presentes em seus microbiomas nasais.
p Os autores então compararam os transcriptomas nasais dos sujeitos de teste e encontraram 692 genes expressos de forma diferente entre pacientes com COVID-19 e profissionais de saúde. Os genes regulados positivamente em indivíduos COVID-19-positivos foram associados às vias de defesa do hospedeiro. Os genes que estão associados à morte celular, bem como aqueles que codificam para receptores inibitórios, também foram regulados positivamente em pacientes COVID-19.
p Comparativamente, alguns dos genes em COVID-19 que foram considerados regulados negativamente incluíram aqueles que estão associados à manutenção de condições internas consistentes, organização celular, e processamento de tecido neuronal. Além disso, os genes responsáveis pela produção de mucina nas vias nasais, bem como aqueles que influenciam os órgãos sensoriais, também foram considerados regulados para baixo. Tomados em conjunto, esses achados podem explicar a perda de olfato e paladar amplamente relatada em pacientes com COVID-19.
 Pesquisadores descobrem uma nova maneira de se proteger contra doenças no modelo de MS
Pesquisadores descobrem uma nova maneira de se proteger contra doenças no modelo de MS
 Pacientes com SII podem se beneficiar de suplementos de vitamina D,
Pacientes com SII podem se beneficiar de suplementos de vitamina D,
 A doença de Parkinson pode ser prevenida por micróbios intestinais
A doença de Parkinson pode ser prevenida por micróbios intestinais
 Usando FLUOstar Omega para estudar novas bactérias intestinais que podem influenciar nossa saúde
Usando FLUOstar Omega para estudar novas bactérias intestinais que podem influenciar nossa saúde
 O coronavírus é transmitido pelas fezes?
O coronavírus é transmitido pelas fezes?
 Perfil bacteriano intestinal pode prever danos intestinais após radioterapia
Perfil bacteriano intestinal pode prever danos intestinais após radioterapia
 O tipo de bactéria das vias aéreas superiores pode influenciar a gravidade da asma
Um novo estudo levanta a possibilidade de modular os sintomas da asma por meio de bactérias que comumente vivem nas vias aéreas superiores. O trabalho, que foi publicado em 16 de dezembro, 2019, no jo
O tipo de bactéria das vias aéreas superiores pode influenciar a gravidade da asma
Um novo estudo levanta a possibilidade de modular os sintomas da asma por meio de bactérias que comumente vivem nas vias aéreas superiores. O trabalho, que foi publicado em 16 de dezembro, 2019, no jo
 Dieta e nutrição influenciam o microbioma na mucosa do cólon
A dieta é importante para manter a saúde humana, mas seu mecanismo subjacente ainda não é totalmente compreendido. Agora, uma equipe de pesquisadores lança luz sobre a conexão entre dieta e saúde, e t
Dieta e nutrição influenciam o microbioma na mucosa do cólon
A dieta é importante para manter a saúde humana, mas seu mecanismo subjacente ainda não é totalmente compreendido. Agora, uma equipe de pesquisadores lança luz sobre a conexão entre dieta e saúde, e t
 Esse Pepto provavelmente não ajudará sua úlcera
A úlcera péptica é uma ferida ou lesão que se desenvolve no revestimento do esôfago, estômago, ou duodeno. Não faz muito tempo, presumia-se que as úlceras eram causadas por uma pessoa estressada ou si
Esse Pepto provavelmente não ajudará sua úlcera
A úlcera péptica é uma ferida ou lesão que se desenvolve no revestimento do esôfago, estômago, ou duodeno. Não faz muito tempo, presumia-se que as úlceras eram causadas por uma pessoa estressada ou si