 Étude :L'infection aiguë par le SRAS-CoV-2 est associée à une expansion de bactéries pathogènes dans le nez, notamment Pseudomonas Aeruginosa. Crédit d'image :Christoph Burgstedt/Shutterstock
Étude :L'infection aiguë par le SRAS-CoV-2 est associée à une expansion de bactéries pathogènes dans le nez, notamment Pseudomonas Aeruginosa. Crédit d'image :Christoph Burgstedt/Shutterstock Le coronavirus 2 du syndrome respiratoire aigu sévère (SRAS-CoV-2) se propage principalement par inhalation de particules virales en suspension dans l'air. On pense que le site principal de la réplication virale initiale est le nez, plutôt que la bouche, en raison de l'expression plus élevée du récepteur de l'enzyme de conversion de l'angiotensine 2 (ACE2) dans le nez.
La réplication virale rapide dans les voies respiratoires supérieures peut entraîner une infection des voies respiratoires inférieures et une maladie grave. Cependant, de nombreuses études n'ont montré aucune corrélation entre la gravité de la maladie et la charge virale dans le nez, suggérant qu'il peut y avoir d'autres facteurs qui contribuent à la gravité de la maladie.
Plusieurs études ont suggéré que le microbiome du nez et de la gorge pourrait jouer un rôle dans les infections virales. L'infection virale peut perturber le microbiote et conduire à une co-infection, ce qui peut aggraver l'inflammation et perturber la réponse immunitaire. Une infection grave peut également entraîner une perte de l'odorat et du goût. Cependant, il n'y a que quelques études qui ont étudié le microbiome respiratoire chez les patients COVID-19.
Dans une nouvelle étude publiée sur le bioRxiv* serveur de préimpression, des chercheurs de l'Université de Californie à Irvine ont étudié l'effet de l'infection par le SRAS-CoV-2 sur le microbiome nasal.
L'équipe de chercheurs a utilisé le séquençage de l'amplicon du gène de l'acide ribonucléique ribosomique (ARNr) 16S pour déterminer si l'infection par le SRAS-CoV-2 entraînait une modification de la composition du microbiome nasal. Cette expérience a révélé que le microbiome nasal des patients COVID-19 était, En réalité, différent par rapport à ceux qui n'avaient pas la maladie, ainsi que celui qui était présent dans le nez des travailleurs de la santé. Le microbiome nasal des patients COVID-19 et des travailleurs de la santé était plus riche que celui des non-patients ; cependant, les microbiomes des trois types d'échantillons étaient dominés par quelques microbes sélectionnés.
Le microbiome nasal d'une personne en bonne santé est principalement constitué de Corynébactérie, Staphylocoque, Streptocoque, Dolosigranule, et Moraxelle. Les auteurs ont découvert que de nombreuses bactéries pathogènes, tel que Rothia , Acinetobacter , et Pseudomonas, étaient fréquents chez les patients COVID-19, avec des niveaux particulièrement élevés de Pseudomonas aeruginosa rapportés chez ces patients.
Des études ont montré que les infections virales aiguës peuvent altérer le microbiome nasal et favoriser une augmentation des bactéries pathogènes. Une augmentation de Pseudomonas a également été signalé dans des cas de grippe, ce qui suggère que sa présence peut ne pas être spécifique au COVID-19 mais est plutôt une réponse générale aux processus inflammatoires.
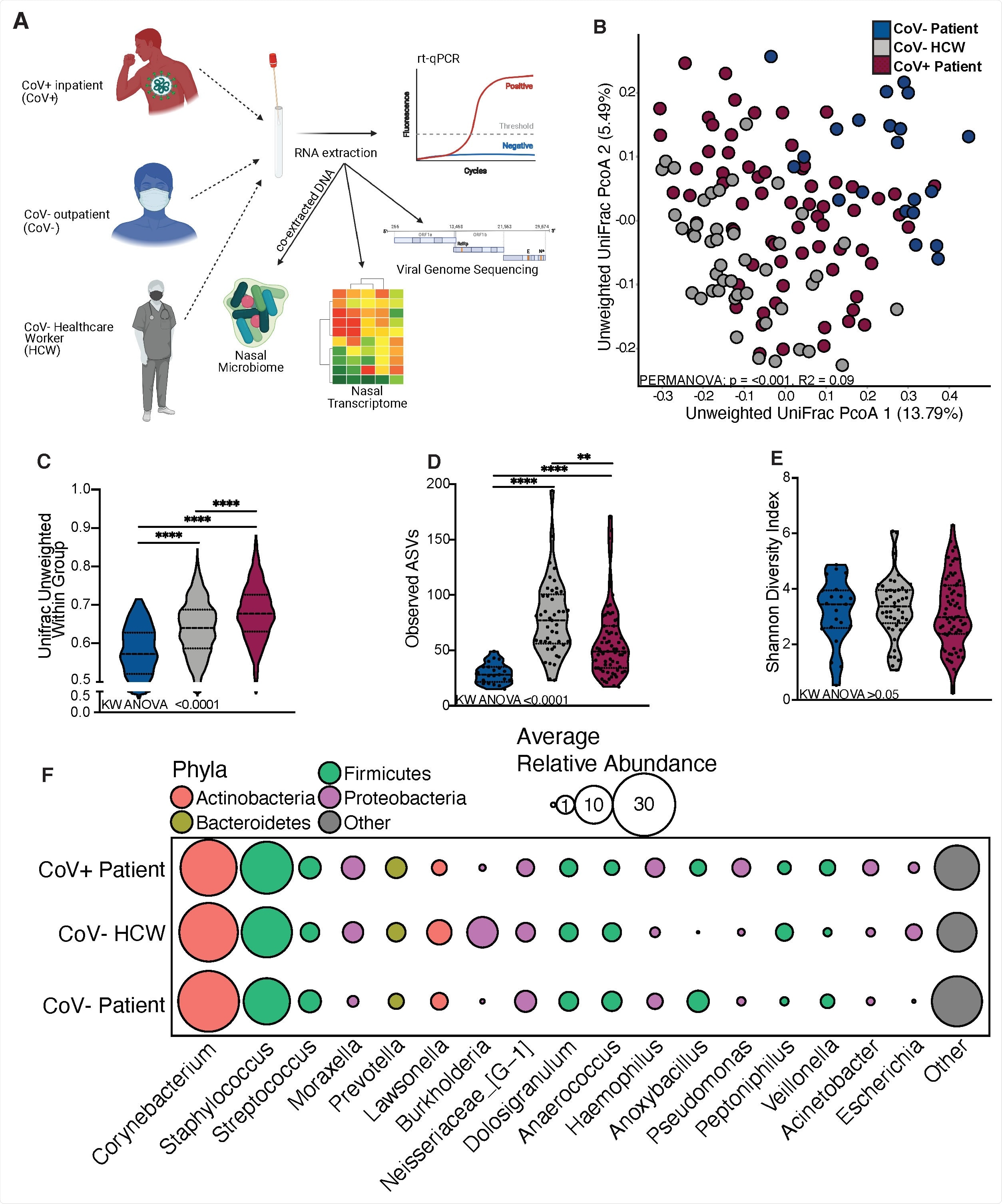 Le microbiome nasal des patients infectés par le SRAS-CoV-2 est distinct. (A) Schéma de conception de l'étude. (B) Analyse des coordonnées principales des communautés microbiennes nasales, distance UniFrac non pondérée colorée par le statut de l'hôte. La contribution du statut d'hôte à la variance totale dans les matrices de dissimilarité UniFrac non pondérées a été mesurée à l'aide de PERMANOVA (Adonis avec 10, 000 permutations). (C, RÉ, E) Graphique de violon illustrant (D) les distances UniFrac moyennes non pondérées (C) le nombre de variantes de séquençage d'amplicons observées et (D) la diversité de Shannon (E) divisée par le statut de l'hôte. La signification pour les panels C-E a été déterminée à l'aide de l'ANOVA non paramétrique de Kruskal Wallis (valeurs p incrustées au bas de chaque panel), avec la comparaison multiple de Dunn * =p <0,05, ** =p<0,01, *** =p <0,001, **** =p <0,0001. (F) Diagrammes à bulles de genres bactériens trouvés à une abondance moyenne supérieure à 1% dans l'ensemble de la population étudiée, classés de gauche à droite en abondance moyenne décroissante. La taille de chaque cercle indique l'abondance relative moyenne pour chaque taxon du groupe indiqué et la couleur de chaque cercle indique à quel phylum bactérien chaque genre appartient.
Le microbiome nasal des patients infectés par le SRAS-CoV-2 est distinct. (A) Schéma de conception de l'étude. (B) Analyse des coordonnées principales des communautés microbiennes nasales, distance UniFrac non pondérée colorée par le statut de l'hôte. La contribution du statut d'hôte à la variance totale dans les matrices de dissimilarité UniFrac non pondérées a été mesurée à l'aide de PERMANOVA (Adonis avec 10, 000 permutations). (C, RÉ, E) Graphique de violon illustrant (D) les distances UniFrac moyennes non pondérées (C) le nombre de variantes de séquençage d'amplicons observées et (D) la diversité de Shannon (E) divisée par le statut de l'hôte. La signification pour les panels C-E a été déterminée à l'aide de l'ANOVA non paramétrique de Kruskal Wallis (valeurs p incrustées au bas de chaque panel), avec la comparaison multiple de Dunn * =p <0,05, ** =p<0,01, *** =p <0,001, **** =p <0,0001. (F) Diagrammes à bulles de genres bactériens trouvés à une abondance moyenne supérieure à 1% dans l'ensemble de la population étudiée, classés de gauche à droite en abondance moyenne décroissante. La taille de chaque cercle indique l'abondance relative moyenne pour chaque taxon du groupe indiqué et la couleur de chaque cercle indique à quel phylum bactérien chaque genre appartient. Ces résultats correspondent aux découvertes d'infections bactériennes secondaires chez les patients COVID-19. La composition du microbiome nasal pourrait donc fonctionner comme le type d'échantillon principal pour évaluer le risque d'infections secondaires d'une personne après sa guérison de certaines infections virales.
En outre, le groupe de recherche basé en Californie a également découvert que les charges virales dans le nez n'affectaient pas la diversité du microbiome nasal. Les patients COVID-19 avec une faible charge virale se sont avérés avoir une quantité plus importante de Streptocoque dans leur microbiome nasal , alors que les patients COVID-19 avec des charges virales modérées avaient une plus grande quantité de Corynébactérie . Troisièmement, Les patients COVID-19 avec des charges virales élevées se sont avérés avoir des niveaux plus élevés de Cutibactérie, Neisseria, et Pseudomonas espèces présentes dans leurs microbiomes nasaux.
Les auteurs ont ensuite comparé les transcriptomes nasaux des sujets testés et ont trouvé 692 gènes exprimés différemment entre les patients COVID-19 et les travailleurs de la santé. Les gènes régulés à la hausse chez les individus positifs au COVID-19 étaient associés aux voies de défense de l'hôte. Les gènes associés à la mort cellulaire, ainsi que ceux qui codent pour les récepteurs inhibiteurs, ont également été régulés à la hausse chez les patients COVID-19.
Relativement, certains des gènes de COVID-19 qui se sont avérés être régulés à la baisse comprenaient ceux qui sont associés au maintien de conditions internes cohérentes, organisation cellulaire, et le traitement des tissus neuronaux. Par ailleurs, les gènes responsables de la production de mucine dans les voies nasales, ainsi que ceux qui influencent les organes sensoriels, se sont également avérés être régulés à la baisse. Pris ensemble, ces résultats pourraient expliquer la perte d'odorat et de goût qui a été largement rapportée chez les patients COVID-19.
Les infections nosocomiales restent un défi majeur en milieu clinique. Ces infections surviennent souvent en raison d'environnements hospitaliers et de travailleurs de la santé contaminés. En raison de leur exposition prolongée dans les hôpitaux, les travailleurs de la santé sont des porteurs courants de microbes pathogènes.
Les auteurs ont trouvé des niveaux plus élevés d'agents pathogènes comme Escerichia, Klebsiella, et Burkholderia dans le microbiome nasal des travailleurs de la santé. L'abondance de Acinetobacter s’est avéré plus élevé chez les patients COVID-19 et les travailleurs de la santé, ce qui peut être dû à un éventuel transfert entre les deux.
Les résultats de la présente étude indiquent un changement dans le microbiome nasal des personnes infectées par le SRAS-CoV-2, avec une augmentation des agents pathogènes comme Pseudomonas aeruginosa . Une limitation de cette étude était le fait que seul le moment a été évalué; donc, les futures études qui comparent la composition du microbiome nasal des patients COVID-19, personnes en bonne santé, et l'hôpital au fil du temps peut améliorer la compréhension de la façon dont le microbiome nasal change.
bioRxiv publie des rapports scientifiques préliminaires qui ne sont pas évalués par des pairs et, donc, ne doit pas être considéré comme concluant, guider la pratique clinique/le comportement lié à la santé, ou traités comme des informations établies.
 Microbiome intestinal et MII - le lien peut-être dans l'alimentation selon une étude
Microbiome intestinal et MII - le lien peut-être dans l'alimentation selon une étude
 Les régimes à base d'ADN et les aliments médicaux personnalisés sont-ils l'avenir de la perte de poids ?
Les régimes à base d'ADN et les aliments médicaux personnalisés sont-ils l'avenir de la perte de poids ?
 Le microbiome intestinal est également une réalité dans la vie fœtale
Le microbiome intestinal est également une réalité dans la vie fœtale
 Pancréatite
Pancréatite
 Peu de preuves de thrombocytopénie associée au vaccin ARNm COVID-19,
Peu de preuves de thrombocytopénie associée au vaccin ARNm COVID-19,
 La composition et la structure du microbiome nasopharyngé sont liées à la gravité de la maladie COVID-19
La composition et la structure du microbiome nasopharyngé sont liées à la gravité de la maladie COVID-19
 Modification du microbiome des voies respiratoires supérieures chez les enfants liée à la susceptibilité au SRAS-CoV-2
Les chercheurs ont testé le microbiome nasopharyngé denfants jusquà 21 ans et ont découvert que le microbiome change avec lâge. Bactéries spécifiques, dont labondance change aussi avec lâge, sont asso
Modification du microbiome des voies respiratoires supérieures chez les enfants liée à la susceptibilité au SRAS-CoV-2
Les chercheurs ont testé le microbiome nasopharyngé denfants jusquà 21 ans et ont découvert que le microbiome change avec lâge. Bactéries spécifiques, dont labondance change aussi avec lâge, sont asso
 La bave de poisson pourrait être une source potentielle d'antibiotiques selon une étude
Avec lavènement des bactéries multirésistantes et le manque de nouveaux antibiotiques, les chercheurs sont à la recherche de nouvelles molécules capables de combattre ces agents pathogènes. Une équipe
La bave de poisson pourrait être une source potentielle d'antibiotiques selon une étude
Avec lavènement des bactéries multirésistantes et le manque de nouveaux antibiotiques, les chercheurs sont à la recherche de nouvelles molécules capables de combattre ces agents pathogènes. Une équipe
 Une nouvelle étude pourrait aider à prévenir les infections mortelles chez les bébés
Les nourrissons prématurés nés avant 28-30 semaines de vie sont à haut risque de nombreuses complications, parmi lesquelles les chances de mourir dune infection débutant dans lintestin sont très grand
Une nouvelle étude pourrait aider à prévenir les infections mortelles chez les bébés
Les nourrissons prématurés nés avant 28-30 semaines de vie sont à haut risque de nombreuses complications, parmi lesquelles les chances de mourir dune infection débutant dans lintestin sont très grand