Unsere ersten Ergebnisse übertrafen unsere optimistischsten Erwartungen. Als Test haben wir eine Rechenaufgabe ausgewählt, die wir zuvor mit etwa 900 Stunden Zeit bei Verwendung von Serverklasse-CPUs gemessen haben. oder ungefähr 13, 000 CPU-Kernstunden. Wir haben festgestellt, dass es auf einer einzigen NVIDIA Tesla V100 GPU in nur 8 Stunden fertig sein könnte. oder etwa 30 Minuten bei Verwendung von 16 GPUs, was die Analyselaufzeiten um mehrere Größenordnungen verkürzen könnte. Eine NVIDIA RTX 2080TI der Workstation-Klasse würde es in etwa 12 Stunden fertigstellen."
Igor Sfiligoi, SDSCs führender wissenschaftlicher Softwareentwickler für High-Throughput-Computing
"Die neue ausführbare Datei wird auch für die Erkundungsarbeit von enormem Wert sein, da der mittelgroße EMP-Datensatz, der früher 13 Stunden auf einer Serverklasse-CPU benötigte, jetzt in etwas mehr als einer Stunde auf einem Laptop mit einer mobilen NVIDIA GTX 1050-GPU ausgeführt werden kann, “ fügte Sfiligoi hinzu.
Sfiligoi hat mit Rob Knight zusammengearbeitet, Gründungsdirektor des Zentrums für Mikrobiom-Innovation, und Professor für Kinderheilkunde, Bioingenieurwesen und Informatik &Ingenieurwissenschaften an der Universität, und Daniel McDonald, wissenschaftlicher Leiter des American Gut Project. Mikrobiome sind das kombinierte genetische Material der Mikroorganismen in einer bestimmten Umgebung, einschließlich des menschlichen Körpers.
„Diese Arbeit begann ursprünglich nicht als Teil der COVID-19-Reaktion, " sagte Sfiligoi. "Wir haben die Diskussion über eine solche Beschleunigung lange vorher begonnen, aber UniFrac ist ein wesentlicher Bestandteil der COVID-19-Forschungspipeline."
UniFrac vergleicht Mikrobiome miteinander unter Verwendung eines Evolutionsbaums, der die DNA-Sequenzen miteinander in Beziehung setzt. "UniFrac spielte eine Schlüsselrolle im Human Microbiome Project, ermöglicht es uns zu verstehen, wie Mikroben in unserem Körper miteinander verbunden sind, und im Earth Microbiome Project, ermöglicht es uns zu verstehen, wie Mikroben auf unserem Planeten miteinander verwandt sind, “ sagte Knight. „Wir verwenden es, um zu verstehen, wie das Mikrobiom einer Person sie mehr oder weniger anfällig für COVID-19 machen könnte. und welche Mikroben in Umgebungen von Gesundheitseinrichtungen über Abwasser bis hin zu Gischt die Umwelt mehr oder weniger gastfreundlich für SARS-CoV-2 machen, das Coronavirus, das COVID-19 verursacht."
Knight bemerkte, dass Sfiligoi die neueste Version des Algorithmus beschleunigt hatte. veröffentlicht vor weniger als zwei Jahren in Naturmethoden , was selbst bereits eine dramatische Geschwindigkeitsverbesserung gegenüber früheren Implementierungen darstellte.
„Da die mikrobiellen Sequenzdaten exponentiell zunehmen, von Dutzenden von Sequenzen bis zu Milliarden, wir müssen alle Algorithmen neu implementieren, ", sagte er. "Dieser jüngste Schritt zeigt wirklich, wie durch die Optimierung der Forschungsinfrastruktur die Zeit bis zum Ergebnis drastisch verkürzt werden kann, während die Genauigkeit der Ergebnisse erhalten bleibt und völlig neue Frageskalen gestellt werden können."
Speziell, Sfiligoi verwendet OpenACC, eine benutzerorientierte, richtlinienbasiertes paralleles Programmiermodell, um die vorhandene Striped UniFrac-Implementierung auf GPUs zu portieren, da dies eine einzige Codebasis für CPU- und GPU-Code ermöglicht. Eine zusätzliche Beschleunigung wurde durch die sorgfältige Ausnutzung der Cache-Lokalität erreicht. Außerdem wurde der Einsatz von Gleitkomma-Mathematik mit geringerer Genauigkeit untersucht, um GPUs der Verbraucherklasse, die normalerweise in Desktop- und Laptop-Computern zu finden sind, effektiv auszunutzen.
UniFrac wurde ursprünglich unter Verwendung von Gleitkomma-Mathematik mit höherer Präzision entwickelt und implementiert. oft als fp64-Codepfad bezeichnet. Die Gleitkomma-Mathematik mit höherer Genauigkeit wurde verwendet, um die Zuverlässigkeit der Ergebnisse zu maximieren. Nach der Implementierung der Gleitkomma-Mathematik mit niedrigerer Genauigkeit, normalerweise als fp32-Codepfad bezeichnet, Forscher beobachteten fast identische Ergebnisse, aber mit deutlich kürzeren Rechenzeiten.
„Wir haben eine 3-fache Beschleunigung im fp32-Codepfad für Gaming-GPUs wie den 2080 Ti und den mobilen 1050 gesehen. und wir glauben, dass die Genauigkeit für die überwiegende Mehrheit der Studien ausreichend sein sollte, " erklärte Sfiligoi.
Außerdem, Die Codeänderungen, die eingeführt wurden, um die GPU-Berechnung zu beschleunigen, haben auch die Ausführung auf CPU-Ressourcen erheblich beschleunigt. Die oben erwähnte Rechenaufgabe kann jetzt in etwa 200 Stunden auf derselben Serverklasse-CPU abgeschlossen werden. eine 4-fache Beschleunigung, laut den Forschern.
"Berechnung auf GPU-fähigen persönlichen Geräten verfügbar machen, sogar Laptops, beseitigt für viele Wissenschaftler eine große Barriere innerhalb der Ressourceninfrastruktur, “ sagte Sfiligoi.
 Getreidefreies, milchfreies, nussfreies, kohlenhydratarmes Bananenbrot
Getreidefreies, milchfreies, nussfreies, kohlenhydratarmes Bananenbrot
 GSK-3-Inhibitoren sind vielversprechend bei der Behandlung von Coronavirus-Infektionen
GSK-3-Inhibitoren sind vielversprechend bei der Behandlung von Coronavirus-Infektionen
 FODMAP-arme Leckereien
FODMAP-arme Leckereien
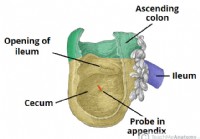 Der Blinddarm
Der Blinddarm
 Was ist operative Laparoskopie?
Was ist operative Laparoskopie?
 Veränderungen der Darmbakterien beeinflussen die Ergebnisse der Lupus-Behandlung in der Schwangerschaft
Veränderungen der Darmbakterien beeinflussen die Ergebnisse der Lupus-Behandlung in der Schwangerschaft
 Warum Sie natürliche Ballaststoffquellen in Ihre Ernährung aufnehmen sollten
Das Interesse an Low-Carb-Diäten wie der ketogenen Diät oder Atkins ist in den letzten Jahren gewachsen. Jedoch, wie schlechte Fette und gute Fette, Kohlenhydrate können je nach Zuckergehalt und Balla
Warum Sie natürliche Ballaststoffquellen in Ihre Ernährung aufnehmen sollten
Das Interesse an Low-Carb-Diäten wie der ketogenen Diät oder Atkins ist in den letzten Jahren gewachsen. Jedoch, wie schlechte Fette und gute Fette, Kohlenhydrate können je nach Zuckergehalt und Balla
 Präbiotika vs. Probiotika vs. Postbiotika – was ist der Unterschied?
Ihr Verdauungstrakt enthält Mikrobiota. Dies sind potenziell gute und schädliche Bakterien, Viren, Pilze und Protozoen. Um so gesund wie möglich zu sein, brauchen Sie ein Gleichgewicht zwischen den be
Präbiotika vs. Probiotika vs. Postbiotika – was ist der Unterschied?
Ihr Verdauungstrakt enthält Mikrobiota. Dies sind potenziell gute und schädliche Bakterien, Viren, Pilze und Protozoen. Um so gesund wie möglich zu sein, brauchen Sie ein Gleichgewicht zwischen den be
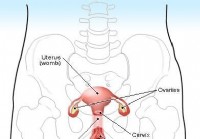 Gutartige Uteruswucherungen (Wucherungen der Gebärmutter)
Was sind Uteruswucherungen? Darstellung des weiblichen Fortpflanzungssystems Uteruswucherungen sind Gewebevergrößerungen der weiblichen Gebärmutter (Uterus). Uteruswucherungen können entweder durc
Gutartige Uteruswucherungen (Wucherungen der Gebärmutter)
Was sind Uteruswucherungen? Darstellung des weiblichen Fortpflanzungssystems Uteruswucherungen sind Gewebevergrößerungen der weiblichen Gebärmutter (Uterus). Uteruswucherungen können entweder durc