Nuestros resultados iniciales superaron nuestras expectativas más optimistas. Como prueba, seleccionamos un desafío computacional que previamente medimos que requería unas 900 horas de tiempo usando CPU de clase de servidor, o alrededor de 13, 000 horas de núcleo de CPU. Descubrimos que se podía terminar en solo 8 horas con una sola GPU NVIDIA Tesla V100, o unos 30 minutos si usa 16 GPU, lo que podría reducir los tiempos de ejecución del análisis en varios órdenes de magnitud. Una NVIDIA RTX 2080TI de clase de estación de trabajo lo terminaría en aproximadamente 12 horas ".
Igor Sfiligoi, Desarrollador de software científico líder de SDSC para computación de alto rendimiento
"El nuevo ejecutable también será de gran valor para el trabajo exploratorio, Dado que el conjunto de datos EMP de tamaño moderado que solía requerir 13 horas en una CPU de clase de servidor, ahora se puede ejecutar en poco más de una hora en una computadora portátil que contenga una GPU NVIDIA GTX 1050 móvil, "añadió Sfiligoi.
Sfiligoi ha estado colaborando con Rob Knight, director fundador del Center for Microbiome Innovation, y profesor de Pediatría, Bioingeniería e Informática e Ingeniería en la universidad, y Daniel McDonald, director científico del American Gut Project. Los microbiomas son el material genético combinado de los microorganismos en un entorno particular, incluido el cuerpo humano.
"Este trabajo no comenzó inicialmente como parte de la respuesta COVID-19, "dijo Sfiligoi." Comenzamos la discusión sobre tal aceleración mucho antes, pero UniFrac es una parte esencial de la línea de investigación de COVID-19 ".
UniFrac compara los microbiomas entre sí mediante un árbol evolutivo que relaciona las secuencias de ADN entre sí. "UniFrac jugó un papel clave en el Proyecto del Microbioma Humano, permitiéndonos comprender cómo se relacionan los microbios en nuestros cuerpos, y en el Earth Microbiome Project, permitiéndonos comprender cómo se relacionan los microbios en nuestro planeta, ", dijo Knight." Lo estamos utilizando para comprender cómo el microbioma de una persona podría hacerla más o menos susceptible al COVID-19, y qué microbios en entornos que van desde las instalaciones de atención médica hasta las aguas residuales y la pulverización del océano hacen que el entorno sea más o menos hospitalario para el SARS-CoV-2, el coronavirus que causa COVID-19 ".
Knight notó que Sfiligoi había acelerado la última versión del algoritmo, publicado hace menos de dos años en Métodos de la naturaleza , lo que en sí mismo ya representó una mejora dramática de la velocidad con respecto a implementaciones anteriores.
"A medida que los datos de la secuencia microbiana aumentan exponencialmente, de decenas de secuencias a miles de millones, tenemos que volver a implementar todos los algoritmos, ", dijo." Este último paso realmente muestra cómo la optimización de la infraestructura de investigación puede reducir drásticamente el tiempo de obtención de resultados, al tiempo que se preserva la precisión de los hallazgos y se permite formular escalas de preguntas completamente nuevas ".
Específicamente, Sfiligoi usó OpenACC, un usuario impulsado, modelo de programación paralela basado en directivas para trasladar la implementación de Striped UniFrac existente a las GPU porque esto permite una única base de código para el código de la CPU y la GPU. Se obtuvo una aceleración adicional explotando cuidadosamente la localidad de caché. También se exploró el uso de matemáticas de punto flotante de menor precisión para explotar de manera eficaz las GPU de nivel de consumidor que normalmente se encuentran en computadoras de escritorio y portátiles.
UniFrac se diseñó originalmente y siempre se implementó utilizando matemática de punto flotante de mayor precisión, a menudo llamado ruta de código fp64. Se utilizó la matemática de punto flotante de mayor precisión para maximizar la confiabilidad de los resultados. Después de implementar la matemática de punto flotante de menor precisión, generalmente llamado ruta de código fp32, los investigadores observaron resultados casi idénticos, pero con tiempos de cálculo significativamente más cortos.
"Vimos una aceleración 3 veces mayor en la ruta del código fp32 para GPU de juegos como la 2080 Ti y la 1050 móvil, y creemos que la precisión debería ser adecuada para la gran mayoría de estudios, "explicó Sfiligoi.
Es más, Los cambios de código introducidos para acelerar el cálculo de la GPU también aceleraron significativamente la ejecución de los recursos de la CPU. El desafío computacional mencionado anteriormente ahora se puede completar en aproximadamente 200 horas en la misma CPU de clase de servidor, una aceleración 4x, según los investigadores.
"Hacer que la computación esté disponible en dispositivos personales habilitados para GPU, incluso laptops, elimina una gran barrera dentro de la infraestructura de recursos para muchos científicos, "dijo Sfiligoi.
 ¿Por qué la salud intestinal aparece en las conversaciones con tanta frecuencia?
¿Por qué la salud intestinal aparece en las conversaciones con tanta frecuencia?
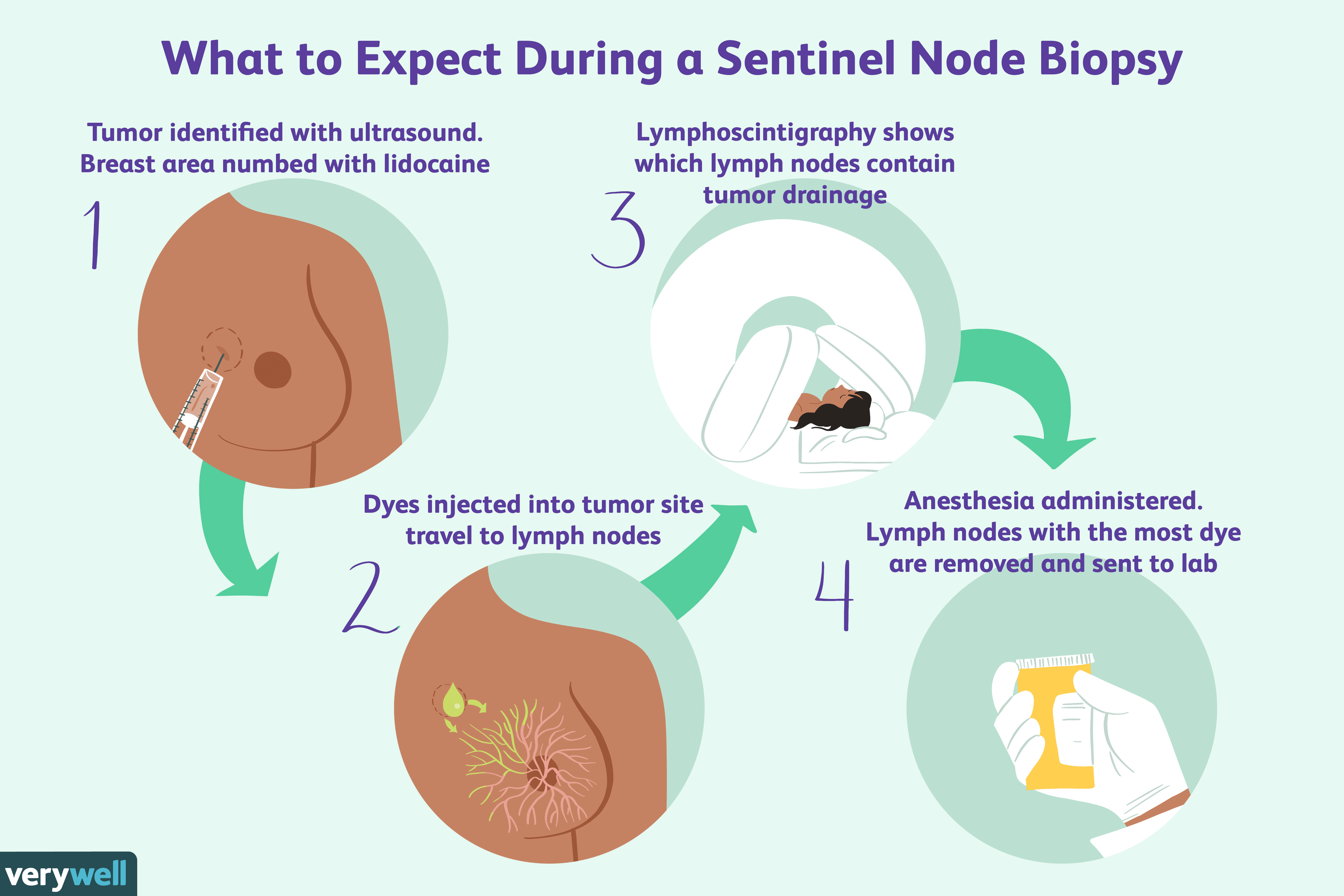 Una descripción general del cáncer de mama con ganglios linfáticos positivos
Una descripción general del cáncer de mama con ganglios linfáticos positivos
 7 remedios caseros para el estreñimiento
7 remedios caseros para el estreñimiento
 Agosto es el mes de concientización sobre la gastroparesia
Agosto es el mes de concientización sobre la gastroparesia
 ¿Qué alimentos desencadenan la colitis ulcerosa?
¿Qué alimentos desencadenan la colitis ulcerosa?
 Probióticos como terapia adyuvante para pacientes con COVID-19
Probióticos como terapia adyuvante para pacientes con COVID-19
 ¿Qué es una fisura anal?
¿Notas manchas de sangre roja brillante cuando te limpias? ¿Experimentas picazón anal? Si es así, estos podrían ser signos de una fisura anal. El revestimiento del ano es delicado y tiene la capacidad
¿Qué es una fisura anal?
¿Notas manchas de sangre roja brillante cuando te limpias? ¿Experimentas picazón anal? Si es así, estos podrían ser signos de una fisura anal. El revestimiento del ano es delicado y tiene la capacidad
 Enfermedad celíaca
La enfermedad celíaca es una afección médica gastrointestinal autoinmune que daña el revestimiento interno del intestino delgado. Para aquellos que tienen enfermedad celíaca, una sustancia llamada gli
Enfermedad celíaca
La enfermedad celíaca es una afección médica gastrointestinal autoinmune que daña el revestimiento interno del intestino delgado. Para aquellos que tienen enfermedad celíaca, una sustancia llamada gli
 Contrarrestar los efectos negativos del estrés
Se está produciendo una epidemia de inflamación de cortisol... y si no lo sabe, mire este video. El resultado final:el cortisol es extremadamente bueno para usted, pero es malo si sus niveles de cort
Contrarrestar los efectos negativos del estrés
Se está produciendo una epidemia de inflamación de cortisol... y si no lo sabe, mire este video. El resultado final:el cortisol es extremadamente bueno para usted, pero es malo si sus niveles de cort