El desarrollo de ciertos tipos de cáncer:cáncer gástrico y cáncer de cuello uterino, por ejemplo, tienen vínculos microbianos bien establecidos. Nuestras tecnologías para estudiar el microbioma humano han avanzado rápidamente en las últimas dos décadas y continúan avanzando a diario. Como resultado, ahora tenemos muchas técnicas nuevas para generar datos sobre la composición y actividades del microbioma humano, y muchos investigadores del cáncer están trabajando para utilizar esta información para comprender nuevos vínculos microbianos.
Sin embargo, dado que los métodos analíticos son tan nuevos, Actualmente se carece del software necesario para convertir estos datos en nuevos conocimientos. Con esta financiación, llenaremos ese vacío de software con el desarrollo de un nuevo software de código abierto para relacionar el microbioma humano con el cáncer. Esperamos que, en última instancia, eso nos permita comprender mejor el desarrollo del cáncer, para detectar el cáncer antes y para mejorar el tratamiento y la recuperación del cáncer ".
Greg Caporaso, Director, Centro de ciencia aplicada del microbioma
El proyecto financiado por el NCI permitirá a Caporaso y su equipo mejorar QIIME 2, la plataforma de software de bioinformática que lanzaron por primera vez a finales de 2016, para mejorar el acceso a los métodos y datos bioinformáticos del microbioma del cáncer. El equipo de Caporaso en NAU incluye estudiantes graduados y universitarios e ingenieros de software de tiempo completo.
Matthew Dillon y Evan Bolyen, dos ingenieros de software de investigación en el laboratorio de Caporaso y coautores del artículo QIIME 2, participará de forma centralizada en todos los aspectos de diseño y desarrollo de este proyecto. El equipo planea desarrollar QIIME 2 en una plataforma bioinformática multiómica de microbioma, Apoyar el análisis y la integración de genómica, metagenómico metabolómica y otros datos "ómicos", impulsado por las necesidades de la comunidad de investigadores del cáncer.
"Muchos proyectos importantes de microbiomas del cáncer han logrado avances al integrar diferentes tipos de datos, Sin embargo, quedan obstáculos técnicos considerables para hacer que la bioinformática multiómica del microbioma sea accesible para todos los investigadores cuyos proyectos se beneficiarían de estos métodos. "Dijo Caporaso.
Con experiencia en ingeniería de software, Proyecto anterior de Caporaso, QIIME 1, se inició hace 12 años en colaboración con su asesor de posdoctorado Rob Knight, ahora director del Centro de Innovación del Microbioma de la Universidad de California, San Diego (UCSD). QIIME 1 fue diseñado para facilitar sus propios estudios en microbiomas, como los que se encuentran en humanos o en el suelo, pero también para hacer que estos métodos sean accesibles a todos los investigadores de microbiomas.
Caporaso se incorporó a la facultad de NAU en 2011, donde continuó su trabajo en QIIME 1. A través de su trabajo con la Asociación para la Prevención del Cáncer de los Nativos Americanos, y posteriormente durante un año sabático en el NCI, Caporaso se dio cuenta de la importancia potencial del microbioma humano para el cáncer. Sus artículos principales sobre QIIME 1 y 2 ahora se han citado casi 25, 000 veces en la literatura de investigación primaria, convirtiéndolo en uno de los investigadores más citados en NAU, según Google Scholar, y Caporaso señala que casi el 20 por ciento de esas citas provienen de estudios sobre el cáncer. Esto lo llevó a comenzar a centrar sus esfuerzos en brindar un mejor apoyo a la comunidad de investigación del cáncer con QIIME, y finalmente a este premio de cinco años del NCI.
"Esto es emocionante para los investigadores del cáncer porque permitirá un nuevo tipo de estudio en la investigación del microbioma, ", dijo." QIIME se ha utilizado típicamente para generar una comprensión taxonómica del microbioma - qué microbios están presentes en este entorno, y cómo las comunidades de microbiomas se comparan entre sí en función de su composición taxonómica. Se están comenzando a aplicar nuevas tecnologías que nos ayuden a considerar otros factores, como las actividades biológicas en las que participan los microbios, y los productos metabólicos de esas actividades. Integrando estos datos, junto con datos sobre el anfitrión, como su genoma, es seguro que nos llevará a una nueva comprensión mecanicista del papel del microbioma en el cáncer ".
QIIME 2 apoyará el análisis de nuevos tipos de datos, como la metagenómica y la metabolómica, para responder preguntas sobre la actividad de los microbios. Esto incluirá información sobre los genes funcionales codificados en los genomas microbianos y los metabolitos presentes en el medio ambiente (moléculas pequeñas como cafeína o etanol y productos producidos por microbios) y cómo podrían afectar al huésped.
"Con el perfil de microbioma, nos estamos haciendo una idea de la biología; con perfil de metabolitos, estamos obteniendo una imagen de la química. Eso nos ayuda a entender lo más grande Una visión más holística de lo que está sucediendo en este entorno infinitamente complejo del microbioma intestinal donde hay billones de células que interactúan entre sí y con sus entornos. todos crean y consumen metabolitos, que impactan su comportamiento y el comportamiento de nuestras células. Podremos saber no solo quién está allí en términos de microorganismos, pero que estan haciendo dónde viven y cómo interactúan ".
Al igual que con QIIME 1, QIIME 2 es una plataforma de software de código abierto, gratis y disponible para que lo use cualquier persona. QIIME 2 fue diseñado para expandir los métodos automatizados de seguimiento e informes para mejorar la reproducibilidad de la investigación, y con esta financiación, el equipo creará nuevas herramientas para ayudar con el archivo de datos a largo plazo. Las actualizaciones de QIIME 2 se publican trimestralmente por el equipo de Caporaso, y ya han comenzado a trabajar para lograr algunos de los objetivos de esta subvención.
"Este es un proyecto increíblemente emocionante para la comunidad de investigación del microbioma del cáncer, "dijo Melissa Herbst-Kralovetz, profesora asociada en el Centro Oncológico de la Universidad de Arizona y directora del Programa de Investigación sobre la Salud de la Mujer en la Facultad de Medicina de la UA-Phoenix. "Mi laboratorio investiga el papel de la microbiota en el cáncer ginecológico, infecciones de transmisión sexual y salud de la mujer. En el presente, estamos aprovechando modelos humanos in vitro 3D para comprender mejor el papel de la microbiota en el desarrollo y la progresión del cáncer, que se basa en la integración de diversos tipos de datos de muestras clínicas y nuestros modelos 3D basados en laboratorio. La nueva funcionalidad que se está desarrollando para QIIME 2 nos ayudará a evaluar la precisión de estos modelos, y, en última instancia, traducir la información que obtenemos de estos modelos 3D a la clínica para combatir el cáncer ".
"Cuando podamos empezar a conectar la biología del huésped, la microbiología y la química, ahí es cuando realmente seremos capaces de descubrir algunos de los eslabones que faltan entre el microbioma y el desarrollo del cáncer o el tratamiento del cáncer. "Dijo Caporaso.
 Por qué Paleo FX reavivó mi compromiso con la salud
Por qué Paleo FX reavivó mi compromiso con la salud
 Histoplasmosis (enfermedad de las cavernas)
Histoplasmosis (enfermedad de las cavernas)
 tarta de chocolate crudo y mandarina
tarta de chocolate crudo y mandarina
 Lo que debe saber sobre el bentilo (diciclomina)
Lo que debe saber sobre el bentilo (diciclomina)
 ¿Cuál es la expectativa de vida de una persona con la enfermedad de Wilson?
¿Cuál es la expectativa de vida de una persona con la enfermedad de Wilson?
 Pastillas anticonceptivas retiradas del mercado debido al peligro de embarazo no deseado
Pastillas anticonceptivas retiradas del mercado debido al peligro de embarazo no deseado
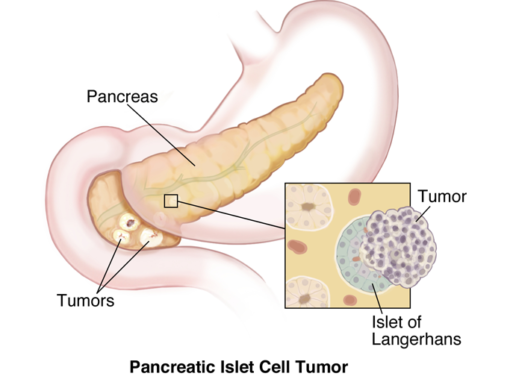 Tumor neuroendocrino pancreático (PNET)
Tumor neuroendocrino pancreático (PNET) se define como tumores de células de los islotes del páncreas que producen hormonas del tipo de las células de los islotes. PNET puede ser funcional o no funcio
Tumor neuroendocrino pancreático (PNET)
Tumor neuroendocrino pancreático (PNET) se define como tumores de células de los islotes del páncreas que producen hormonas del tipo de las células de los islotes. PNET puede ser funcional o no funcio
 El arma misteriosa que lleva dentro (Comprender su sistema inmunitario)
Imagina que es el año 2050... y a través de una serie de eventos, te han puesto a cargo de uno de los sistemas de defensa más sofisticados del universo... Una fuerza como algo que nunca antes habíamo
El arma misteriosa que lleva dentro (Comprender su sistema inmunitario)
Imagina que es el año 2050... y a través de una serie de eventos, te han puesto a cargo de uno de los sistemas de defensa más sofisticados del universo... Una fuerza como algo que nunca antes habíamo
 Una "colonoscopia virtual" no es suficiente para prevenir el cáncer de colon
En el Center for Colonoscopy Excellence (CCE), una división de Gastroenterología de San Francisco, entendemos que los pacientes pueden considerar una “colonoscopia virtual” en lugar de una normal. Una
Una "colonoscopia virtual" no es suficiente para prevenir el cáncer de colon
En el Center for Colonoscopy Excellence (CCE), una división de Gastroenterología de San Francisco, entendemos que los pacientes pueden considerar una “colonoscopia virtual” en lugar de una normal. Una