Le développement de certains types de cancer - cancer gastrique et cancer du col de l'utérus, par exemple - ont des liens microbiens bien établis. Nos technologies d'étude du microbiome humain ont rapidement progressé au cours des deux dernières décennies et continuent de progresser quotidiennement. Par conséquent, nous disposons désormais de nombreuses nouvelles techniques pour générer des données sur la composition et les activités du microbiome humain, et de nombreux chercheurs sur le cancer travaillent à utiliser ces informations pour comprendre de nouveaux liens microbiens.
Cependant, puisque les méthodes analytiques sont si nouvelles, le logiciel nécessaire pour transformer ces données en nouvelles connaissances fait actuellement défaut. Avec ce financement, nous comblerons cette lacune logicielle avec le développement de nouveaux logiciels open source pour relier le microbiome humain au cancer. Nous espérons que cela nous permettra à terme de mieux comprendre le développement du cancer, pour détecter le cancer plus tôt et pour améliorer le traitement et la guérison du cancer."
Greg Caporaso, Réalisateur, Centre de science appliquée du microbiome
Le projet financé par le NCI permettra à Caporaso et à son équipe d'améliorer QIIME 2, la plate-forme logicielle de bioinformatique qu'ils ont lancée pour la première fois fin 2016, améliorer l'accès aux méthodes et données bioinformatiques du microbiome du cancer. L'équipe de Caporaso à NAU comprend des étudiants diplômés et de premier cycle et des ingénieurs logiciels à temps plein.
Matthew Dillon et Evan Bolyen, deux ingénieurs logiciels de recherche dans le laboratoire de Caporaso et co-premiers auteurs de l'article QIIME 2, sera impliqué de manière centrale dans tous les aspects de conception et de développement de ce projet. L'équipe prévoit de développer QIIME 2 en une plate-forme bioinformatique multi-omique de microbiome, soutenir l'analyse et l'intégration de la génomique, métagénomique, métabolomique et autres données « omiques », motivés par les besoins de la communauté de recherche sur le cancer.
« De nombreux projets importants sur le microbiome du cancer ont progressé en intégrant différents types de données, pourtant, des obstacles techniques considérables demeurent pour rendre la bioinformatique multi-omique du microbiome accessible à tous les chercheurs dont les projets bénéficieraient de ces méthodes, " a déclaré Caporaso.
Avec une formation en génie logiciel, Le projet précédent de Caporaso, QIIME 1, a débuté il y a 12 ans en collaboration avec son conseiller postdoctoral Rob Knight, maintenant directeur du Center for Microbiome Innovation de l'Université de Californie, San Diego (UCSD). QIIME 1 a été conçu pour faciliter leurs propres études sur les microbiomes - tels que ceux trouvés chez l'homme ou dans le sol - mais aussi pour rendre ces méthodes accessibles à tous les chercheurs en microbiomes.
Caporaso a rejoint la faculté de NAU en 2011, où il a poursuivi son travail sur QIIME 1. Grâce à son travail avec le Partenariat pour la prévention du cancer des Amérindiens, puis lors d'un congé sabbatique au NCI, Caporaso a réalisé l'importance potentielle du microbiome humain pour le cancer. Ses articles principaux sur QIIME 1 et 2 ont maintenant été cités près de 25, 000 fois dans la littérature de recherche primaire, faisant de lui l'un des chercheurs les plus cités de la NAU, selon Google Scholar, et Caporaso note que près de 20 pour cent de ces citations proviennent d'études sur le cancer. Cela l'a amené à concentrer ses efforts sur un meilleur soutien à la communauté de la recherche sur le cancer avec QIIME, et finalement à ce prix de cinq ans du NCI.
"C'est passionnant pour les chercheurs sur le cancer car cela va permettre un nouveau type d'étude dans la recherche sur le microbiome, " Il a dit. " QIIME a généralement été utilisé pour générer une compréhension taxonomique du microbiome - quels microbes sont présents dans cet environnement, et comment les communautés de microbiomes se comparent les unes aux autres en fonction de leur composition taxonomique. De nouvelles technologies commencent à être appliquées pour nous aider à considérer d'autres facteurs, telles que les activités biologiques dans lesquelles les microbes sont engagés, et les produits métaboliques de ces activités. En intégrant ces données, ainsi que des données sur l'hôte telles que son génome, est sûr de nous conduire à de nouvelles compréhensions mécanistes du rôle du microbiome dans le cancer."
QIIME 2 supportera l'analyse de nouveaux types de données, comme la métagénomique et la métabolomique, pour répondre aux questions concernant l'activité des microbes. Cela inclura des informations sur les gènes fonctionnels codés dans les génomes microbiens et les métabolites présents dans l'environnement - de petites molécules comme la caféine ou l'éthanol et les produits fabriqués par des microbes - et leur impact potentiel sur l'hôte.
"Avec le profilage du microbiome, on se fait une idée de la biologie; avec profilage des métabolites, nous avons une image de la chimie. Cela nous aide à comprendre le plus grand, une vue plus holistique de ce qui se passe dans cet environnement infiniment complexe du microbiome intestinal où vous avez des milliards de cellules qui interagissent les unes avec les autres et avec leurs environnements, tous les métabolites créateurs et consommateurs, qui impactent leur comportement et le comportement de nos cellules. Nous pourrons non seulement savoir qui est là en termes de micro-organismes, mais ce qu'ils font, où ils vivent et comment ils interagissent."
Comme pour QIIME 1, QIIME 2 est une plateforme logicielle open source, gratuit et accessible à tous. QIIME 2 a été conçu pour étendre les méthodes automatisées de suivi et de reporting afin d'améliorer la reproductibilité de la recherche, et avec ce financement, l'équipe créera de nouveaux outils pour faciliter l'archivage des données à long terme. Les mises à jour de QIIME 2 sont publiées tous les trimestres par l'équipe de Caporaso, et ils ont déjà commencé à travailler vers certains des objectifs de cette subvention.
« C'est un projet incroyablement excitant pour la communauté de recherche sur le microbiome du cancer, " a déclaré Melissa Herbst-Kralovetz, professeur agrégé à l'Université de l'Arizona Cancer Center et directeur du programme de recherche sur la santé des femmes à l'UA College of Medicine-Phoenix. "Mon laboratoire étudie le rôle du microbiote dans le cancer gynécologique, les infections sexuellement transmissibles et la santé des femmes. Maintenant, nous utilisons des modèles humains 3D in vitro pour mieux comprendre le rôle du microbiote dans le développement et la progression du cancer, qui repose sur l'intégration de divers types de données provenant d'échantillons cliniques et de nos modèles 3D en laboratoire. La nouvelle fonctionnalité en cours de développement pour QIIME 2 nous aidera à évaluer la précision de ces modèles, et finalement traduire les informations que nous obtenons de ces modèles 3D à la clinique pour lutter contre le cancer. »
« Quand nous pourrons commencer à connecter la biologie de l'hôte, la microbiologie et la chimie, c'est à ce moment-là que nous pourrons vraiment comprendre certains des chaînons manquants entre le microbiome et le développement du cancer ou le traitement du cancer, " a déclaré Caporaso.
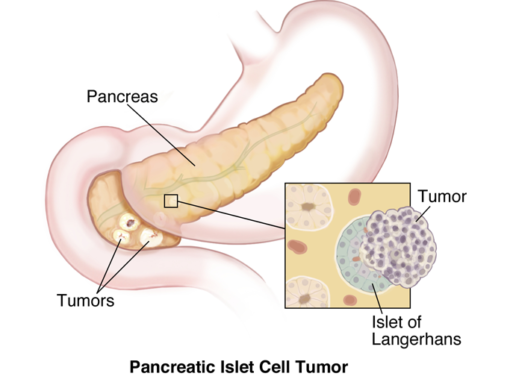 Tumeur neuroendocrine du pancréas (TNEP)
Tumeur neuroendocrine du pancréas tumeur (PNET) est définie comme des tumeurs des cellules insulaires du pancréas qui fabriquent des hormones de type cellules insulaires. PNET peut être fonctionnel ou
Tumeur neuroendocrine du pancréas (TNEP)
Tumeur neuroendocrine du pancréas tumeur (PNET) est définie comme des tumeurs des cellules insulaires du pancréas qui fabriquent des hormones de type cellules insulaires. PNET peut être fonctionnel ou
 15 déclencheurs d'inflammation à l'origine de votre poussée de diarrhée
Il était 6 heures du matin et les yeux de ma femme se remplissaient dépuisement. Elle était en travail depuis 12 heures et les choses ne progressaient pas. Nous étions restés debout presque toute la
15 déclencheurs d'inflammation à l'origine de votre poussée de diarrhée
Il était 6 heures du matin et les yeux de ma femme se remplissaient dépuisement. Elle était en travail depuis 12 heures et les choses ne progressaient pas. Nous étions restés debout presque toute la
 calculs biliaires
Que sont les calculs biliaires ? Les calculs biliaires sont des dépôts ressemblant à des pierres qui se forment dans la vésicule biliaire, un petit organe en forme de poire situé sous le foie. La vés
calculs biliaires
Que sont les calculs biliaires ? Les calculs biliaires sont des dépôts ressemblant à des pierres qui se forment dans la vésicule biliaire, un petit organe en forme de poire situé sous le foie. La vés