Anche prima della pandemia di COVID-19, sapevamo che i viaggi internazionali stavano contribuendo al rapido aumento globale e alla diffusione della resistenza antimicrobica. Ma la novità è che abbiamo trovato numerosi geni completamente nuovi associati alla resistenza antimicrobica che suggeriscono un problema preoccupante all'orizzonte ."
Alaric D'Souza, Studente di dottorato/dottorato, Co-primo autore dello studio, Università di Washington
Lo studio è stato pubblicato il 6 giugno in Medicina del genoma
La ricerca conferma che i viaggiatori internazionali spesso tornano a casa con una quantità inaspettata di nuovi ceppi batterici che si contendono una posizione tra le migliaia che normalmente risiedono nel microbioma intestinale.
Povertà, scarse condizioni igienico-sanitarie e il cambiamento delle pratiche agricole hanno trasformato molti a basso reddito, sviluppare le regioni in punti caldi per le malattie diffuse dai batteri, comprese le infezioni che sono sempre più resistenti a una serie di trattamenti farmacologici antibiotici.
L'elevata densità di popolazione rende facile la condivisione di questi batteri tra i residenti e i viaggiatori della comunità attraverso l'esposizione ad acqua potabile e cibo contaminati, o servizi igienici poco igienizzati, ristoranti, camere d'albergo e trasporti pubblici. A casa, i viaggiatori corrono il rischio di trasferire questi nuovi batteri alla famiglia, amici e altri residenti della comunità.
La ricerca, condotto con l'Università di Maastricht nei Paesi Bassi, ha coinvolto l'analisi delle comunità batteriche nei microbiomi intestinali di 190 adulti olandesi prima e dopo il viaggio in una delle quattro regioni internazionali in cui la prevalenza di geni di resistenza è elevata:Asia sudorientale, Asia del sud, Nord Africa e Africa Orientale.
I campioni fecali analizzati come parte dello studio sono stati selezionati casualmente da un più ampio, indagine multicentrica di circa 2, 000 viaggiatori olandesi, la maggior parte erano turisti, noto come studio sul trasporto di batteri multiresistenti dopo il viaggio (COMBAT).
"Abbiamo riscontrato significativi aumenti legati ai viaggi nell'acquisizione di geni di resistenza, abbondanza e diversità codificate da batteri endemici della regione visitata, "Questi risultati forniscono un forte supporto per i viaggi internazionali come vettore per la diffusione globale di geni di resistenza antimicrobica clinicamente importanti ed evidenziano la necessità di una più ampia sorveglianza dei batteri resistenti agli antimicrobici nei microbiomi intestinali dei viaggiatori di ritorno".
Il nuovo studio è stato progettato dagli autori co-senior John Penders, un microbiologo medico presso l'Università di Maastricht, e Gautam Dantas, dottorato di ricerca, professore di patologia e immunologia alla Washington University. Manish Boolchandani, dottorato di ricerca, un membro del Dantas Lab durante la ricerca e un laureato 2020 del programma di dottorato dell'università in Biologia computazionale e dei sistemi, è anche un primo autore sulla carta.
L'Organizzazione Mondiale della Sanità, i Centri statunitensi per il controllo e la prevenzione delle malattie, e altre agenzie hanno descritto la rapida diffusione della resistenza antimicrobica come una delle più gravi minacce per la salute pubblica che il mondo deve affrontare ora:una catastrofe medica incombente che potrebbe superare il caos creato dalla pandemia di COVID-19.
"Mentre studi precedenti hanno scansionato campioni di feci dei viaggiatori alla ricerca di noti batteri resistenti agli antimicrobici, abbiamo utilizzato una combinazione di sequenziamento shotgun dell'intero metagenoma e metagenomica funzionale per identificare geni noti e nuovi che codificano per la resistenza antimicrobica, "Danti ha detto.
Le tecniche genomiche più tradizionali cercano firme genetiche distintive dei singoli agenti patogeni. Ma tali test possono trovare solo agenti patogeni noti, mentre il sequenziamento metagenomico può identificare tutti gli organismi presenti in un dato campione:batteri buoni, batteri pericolosi e anche quelli completamente nuovi.
In tutto, i ricercatori hanno rilevato 121 geni di resistenza agli antimicrobici nei microbiomi intestinali dei 190 viaggiatori olandesi. Più del 40% di questi geni di resistenza (51 di essi) sono stati scoperti solo utilizzando la tecnica metagenomica più sensibile, suggerendo che i geni potenzialmente pericolosi vengono persi dagli approcci più convenzionali.
Altrettanto preoccupante, i risultati dello studio hanno confermato che 56 geni unici di resistenza antimicrobica erano diventati parte dei microbiomi intestinali dei viaggiatori durante i loro viaggi all'estero, compresi diversi cellulari, geni di resistenza ad alto rischio, come le -lattamasi a spettro esteso (ESBL) e il gene di resistenza alla colistina trasmesso da plasmidi, mcr-1.
La resistenza agli antibiotici beta-lattamici sta emergendo in tutto il mondo e conferisce un'ampia resistenza al trattamento con penicilline e altri importanti antibiotici.
I geni mcr-1 proteggono i batteri da un altro farmaco antimicrobico chiamato colistina, che è il trattamento di ultima istanza per le infezioni da batteri gram-negativi multiresistenti. Se la resistenza alla colistina si diffonde a batteri resistenti ad altri antibiotici, quei batteri potrebbero causare infezioni davvero non curabili, il CDC ha avvertito.
Poiché l'analisi metagenomica consente ai ricercatori di studiare tutti i batteri e i geni in una raccolta di campioni di microbioma intestinale come uno, grande comunità mista di organismi, fornisce anche l'opportunità di esplorare complesse interazioni ecologiche tra questi organismi.
Mentre i batteri possono sviluppare lentamente la resistenza da esposizioni ripetute agli antibiotici nel tempo, diverse comunità batteriche condividono anche geni di resistenza antimicrobica attraverso un processo più rapido noto come trasferimento orizzontale, di solito tramite lo scambio di elementi genetici mobili che consentono a frammenti di DNA di saltare da un batterio all'altro.
"Poiché i geni che codificano per la resistenza a diverse classi di antibiotici si trovano spesso sugli stessi elementi mobili, un singolo scambio orizzontale ha il potenziale per convertire batteri precedentemente sensibili agli antibiotici in un organismo multiresistente ai farmaci, " disse Dantas.
I ricercatori hanno anche utilizzato tecniche metagenomiche per mettere insieme importanti informazioni contestuali sulla posizione e la funzione dei geni di resistenza.
"C'era una significativa associazione di geni di resistenza con elementi genetici mobili, un modo primario in cui i geni di resistenza si diffondono tra i batteri, "D'Souza ha detto. "Anche se il nostro studio non è stato in grado di dimostrare che i geni di resistenza sono trasportati da batteri patogeni, è chiaro che questo è possibile. Inoltre, i viaggiatori internazionali hanno il potenziale per introdurre geni di resistenza nelle proprie comunità quando tornano a casa, e gli studi futuri che affrontino direttamente questa possibilità sono una priorità".
Dantas ha aggiunto:"L'identificazione di nuovi batteri e geni resistenti agli antimicrobici potrebbe svolgere un ruolo importante nel rallentare la diffusione globale della resistenza e guidare potenziali trattamenti per malattie correlate. Il nostro studio pone le basi per tali sforzi offrendo nuove informazioni sui meccanismi genetici che sono alla base del rapida acquisizione e condivisione di geni di resistenza antimicrobica attraverso i microbiomi intestinali delle persone durante i viaggi internazionali".
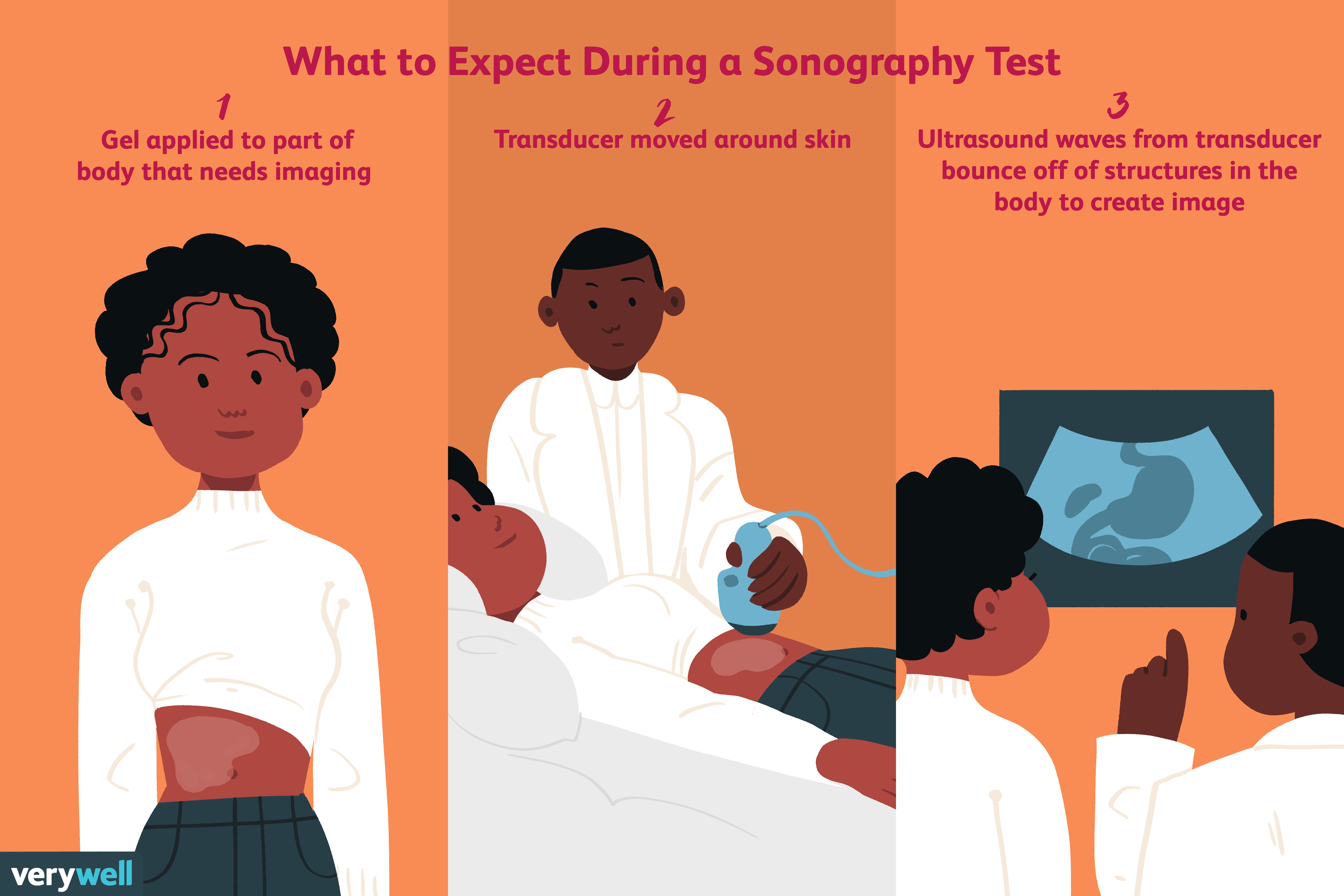 Che cos'è l'ecografia?
Lecografia è un test medico diagnostico che utilizza onde sonore ad alta frequenza, chiamate anche onde ultrasoniche, per rimbalzare sulle strutture del corpo e creare unimmagine. Opportunamente, il t
Che cos'è l'ecografia?
Lecografia è un test medico diagnostico che utilizza onde sonore ad alta frequenza, chiamate anche onde ultrasoniche, per rimbalzare sulle strutture del corpo e creare unimmagine. Opportunamente, il t
 Obiettivo fallito =tempo perso?
Una semplice definizione di progresso è:stai meglio oggi di ieri? Ma è difficile misurare un progresso costante, soprattutto se si tratta di un miglioramento dello 0,001% o dell1% al giorno. Ma è esa
Obiettivo fallito =tempo perso?
Una semplice definizione di progresso è:stai meglio oggi di ieri? Ma è difficile misurare un progresso costante, soprattutto se si tratta di un miglioramento dello 0,001% o dell1% al giorno. Ma è esa
 Come trovare i consigli nutrizionali giusti per un intestino sano? I dietisti registrati sfatano 5 miti comuni sulla salute dell'intestino
Come abbiamo spiegato in questo blog, il modo migliore per mantenere un intestino sano è prestare attenzione ai cibi che scegli di mangiare. Tuttavia, non tutti i consigli nutrizionali disponibili onl
Come trovare i consigli nutrizionali giusti per un intestino sano? I dietisti registrati sfatano 5 miti comuni sulla salute dell'intestino
Come abbiamo spiegato in questo blog, il modo migliore per mantenere un intestino sano è prestare attenzione ai cibi che scegli di mangiare. Tuttavia, non tutti i consigli nutrizionali disponibili onl