Nu, gå et skridt længere, forskere ved Harvard Medical School og Joslin Diabetes Center er gået ud over mikrobielle arter. Analyse af den genetiske sammensætning af bakterier i den menneskelige tarm, teamet har med succes sammenkædet grupper af bakterielle gener, eller "genetiske signaturer, "til flere sygdomme.
Arbejdet bringer forskere tættere på at udvikle test, der kan forudsige sygdomsrisiko eller identificere sygdomstilstedeværelse baseret på en stikprøve af den genetiske sammensætning af en persons mikrobiom.
Fundene, udgives 18. maj i Naturkommunikation , forbinder sæt af bakteriegener med tilstedeværelsen af koronararteriesygdom, skrumpelever, inflammatorisk tarmsygdom, tyktarmskræft, og type 2 diabetes. Analysen indikerer, at tre af disse tilstande-koronararteriesygdom, inflammatorisk tarmsygdom, og levercirrhose-deler mange af de samme bakteriegener. Med andre ord, mennesker, hvis tarme rummer disse bakterielle gener, synes mere tilbøjelige til at have en eller flere af disse tre tilstande.
Arbejdet repræsenterer et betydeligt fremskridt i den nuværende forståelse af forholdet mellem mikrober, der bor i den menneskelige tarm og specifikke sygdomme, sagde teamet. Hvis det bekræftes gennem yderligere forskning, resultaterne kunne informere om design af værktøjer, der kunne måle en persons risiko for en række forhold baseret på analyse af en enkelt fækal prøve, tilføjede de.
Dette åbner et vindue for udvikling af tests med kryds-sygdom, genbaserede indikatorer for patientsundhed. Vi har identificeret genetiske markører, som vi tror i sidste ende kan føre til test, eller bare en test, at identificere foreninger med en række medicinske tilstande. "
Braden Tierney, Undersøg første forfatter og kandidatstuderende, Biologisk og biomedicinsk videnskabsprogram, Harvard Medical School
Forskerne advarer om, at deres undersøgelse ikke var designet til at belyse præcist, hvordan og hvorfor disse mikrobielle gener kan være forbundet med forskellige sygdomme. Så langt, de sagde, det er stadig uklart, om disse bakterier er involveret i sygdomsudvikling eller blot er tilskuere i denne proces.
Målet med undersøgelsen var at afgøre, om grupper af gener pålideligt kunne indikere tilstedeværelsen af forskellige sygdomme. Disse nyligt identificerede mikrobielle genetiske signaturer, imidlertid, kunne studeres nærmere for at bestemme hvilken rolle, hvis nogen, organismerne spiller i sygdomsudvikling.
"Vores undersøgelse understreger værdien af datavidenskab til at drille komplekst samspil mellem mikrober og mennesker, "sagde undersøgelses seniorforfatter Chirag Patel, lektor i biomedicinsk informatik i Blavatnik Institute ved HMS.
Forskerne startede med at indsamle mikrobiomedata fra 13 grupper af patienter på i alt mere end 2, 500 prøver. Næste, de analyserede dataene for at fastslå sammenhænge mellem syv sygdomme og millioner af mikrobielle arter, mikrobielle metaboliske veje, og mikrobielle gener. Ved at afprøve en række modelleringsmetoder-beregning af i alt 67 millioner forskellige statistiske modeller-var de i stand til at observere, hvilke mikrobiomfunktioner der konsekvent opstod som de stærkeste sygdomsassocierede kandidater.
Af alle de forskellige mikrobielle egenskaber-arter, stier, og gener-mikrobielle gener havde den største forudsigelseskraft. Med andre ord, sagde forskerne, grupper af bakterielle gener, eller genetiske signaturer snarere end blot tilstedeværelsen af visse bakteriefamilier, var tættest forbundet med tilstedeværelsen af en given tilstand.
Nogle af de vigtigste observationer omfattede:
Klynger af bakterielle gener, eller genetiske signaturer frem for individuelle bakteriegener, forekommer impliceret i forskellige former for menneskelig sygdom.
Koronararteriesygdom, inflammatorisk tarmsygdom, og levercirrhose har lignende tarmmikrobiom genetiske signaturer.
Type 2 -diabetes, derimod, har en mikrobiomsignatur i modsætning til enhver anden fænotype testet.
Analysen fandt ikke en konsekvent sammenhæng mellem tilstedeværelsen af bakteriearten Solobacterium moorei og tyktarmskræft-en sammenslutning, der tidligere blev rapporteret i talrige undersøgelser. Imidlertid, forskerne identificerede bestemte gener fra en S. moorei -underart forbundet med tyktarmskræft. Dette fund indikerer, at analyse på gen-niveau kan give biomarkører for sygdom med større præcision og mere specificitet sammenlignet med nuværende fremgangsmåder.
Patel sagde, at dette resultat understreger forestillingen om, at det ikke kun er tilstedeværelsen af en given bakteriefamilie, der kan være en risiko, men snarere de stammer og gensignaturer af mikroberne, der betyder noget. Evnen til at identificere sammenkoblinger med en sådan præcision vil være afgørende for at designe tests, der kan måle risiko pålideligt, han tilføjede. Dermed, i dette specifikke eksempel, en test, der er beregnet til at måle tyktarmskræftrisiko ved blot at påvise tilstedeværelsen af S. moorei i tarmen, er muligvis ikke så pålidelig som en mere raffineret test, der måler bakteriegener til at detektere tilstedeværelsen af specifikke stammer af S. moorei, der er forbundet med tyktarmskræft.
To tilstande-ørebetændelse og godartede bløddelssvulster kaldet adenomer-viste svage associationer til tarmmikrobiomet, der tyder på, at mikroorganismer, der er bosat i den menneskelige tarm, sandsynligvis ikke vil spille en rolle i udviklingen af disse tilstande, det er heller ikke sandsynligt, at de er pålidelige indikatorer for, at disse betingelser er til stede.
I en tidligere undersøgelse, HMS-teamet brugte enorme mængder af offentligt tilgængelige DNA-sekventeringsdata fra humane orale og tarmmikrobiomer til at estimere størrelsen af universet af mikrobielle gener i menneskekroppen. Analysen afslørede, at der kan være flere gener i det kollektive menneskelige mikrobiom end stjerner i det observerbare univers.
I betragtning af det store antal mikrobielle gener, der findes i menneskekroppen, de nye fund repræsenterer et stort skridt fremad i forståelsen af kompleksiteten i samspillet mellem menneskelige sygdomme og det menneskelige mikrobiom, sagde forskerne.
"Det ultimative mål for beregningsvidenskab er at generere hypoteser fra et stort antal data, "sagde Tierney." Vores arbejde viser, at dette kan lade sig gøre og åbner så mange nye veje til forskning og undersøgelser, at vi kun er begrænset af tiden, mennesker, og de nødvendige ressourcer til at køre disse tests. "
 The Sad State of American Medicine:Why Patients and Practitioners Are Suffering (og hvordan vi kan løse det)
The Sad State of American Medicine:Why Patients and Practitioners Are Suffering (og hvordan vi kan løse det)
 Folk, der opfanger infektion fra dyrehandel Hvalpe afføring:CDC
Folk, der opfanger infektion fra dyrehandel Hvalpe afføring:CDC
 En oversigt over skørbug
En oversigt over skørbug
 Hele kropsvibrationer hjælper med at reducere betændelse,
Hele kropsvibrationer hjælper med at reducere betændelse,
 Opskrift på jordbærchokoladetrøfler
Opskrift på jordbærchokoladetrøfler
 De rigtige værktøjer maksimerer dine resultater
De rigtige værktøjer maksimerer dine resultater
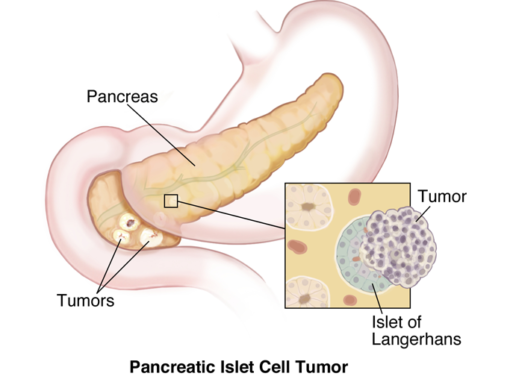 Pancreatic Neuroendocrine Tumor (PNET)
Pancreatic neuroendocrine tumor (PNET) er defineret som ø-celletumorer i bugspytkirtlen, der danner ø-celletypehormoner. PNET kan være funktionelt eller ikke-funktionelt. Funktionelt: (betyder, at de
Pancreatic Neuroendocrine Tumor (PNET)
Pancreatic neuroendocrine tumor (PNET) er defineret som ø-celletumorer i bugspytkirtlen, der danner ø-celletypehormoner. PNET kan være funktionelt eller ikke-funktionelt. Funktionelt: (betyder, at de
 GoodBelly Probiotic Supplement Review
GoodBelly Probiotic Supplement-kapsler er det nyeste produkt fra GoodBelly, virksomheden, der sælger probiotiske juicedrikke. Kapslerne indeholder samme stamme som deres juice:Lactobacillus plantarum
GoodBelly Probiotic Supplement Review
GoodBelly Probiotic Supplement-kapsler er det nyeste produkt fra GoodBelly, virksomheden, der sælger probiotiske juicedrikke. Kapslerne indeholder samme stamme som deres juice:Lactobacillus plantarum
 Top 5 fejl, folk begår på den specifikke kulhydratdiæt
Jeg ønskede at sammensætte en liste over de 5 mest almindelige fejl, som jeg ser i hele SCD-fællesskabet, når folk går over til SCD-livsstilen. Hvis du kan undgå disse 5 ting, er du godt på vej til su
Top 5 fejl, folk begår på den specifikke kulhydratdiæt
Jeg ønskede at sammensætte en liste over de 5 mest almindelige fejl, som jeg ser i hele SCD-fællesskabet, når folk går over til SCD-livsstilen. Hvis du kan undgå disse 5 ting, er du godt på vej til su