Genomische Profil Analyse der diffusen Typ Magenkrebs
Zusammenfassung
Hintergrund
Magenkrebs ist die dritte tödlichste weltweit unter allen Krebsarten. Obwohl Einfalls des intestinalen Typs Magenkrebs verringert hat, wird das Auftreten von diffuse-type noch zu erhöhen und ihr Fortschreiten ist notorisch aggressiv. Es ist nicht genügend Informationen über die Genomvariationen von diffusen Art Magenkrebs, weil seine Zellen in der Regel mit normalen Zellen vermischt werden, und diese geringe cellularity hat es schwierig gemacht, das Genom zu analysieren.
Ergebnisse
Wir ganze Genome und entsprechenden exomes analysieren Magenkrebs von diffusem-Typ, mit angepassten Tumor und normalen Proben von 14 diffusen Typ und fünf Darm-Typ Magenkrebs-Patienten. Somatischer Variationen der diffuse-type Magenkrebs zu finden sind, die denen der Darm-Typ verglichen und auf zuvor Varianten berichtet. Wir bestimmen die durchschnittliche exonische somatischen Mutationsrate der beiden Typen. Wir finden Kandidatentreiber Genen assoziiert und identifizieren neuartige sieben somatische Mutationen in CDH1
, die eine bekannte Magenkarzinom-assoziiertes Gen ist. Dreidimensionale Strukturanalyse des mutiertem E-Cadherin-Proteins deutet darauf hin, dass diese neuen somatische Mutationen signifikante funktionelle Störungen der kritischen Calciumbindungsstellen in dem EC1-2 Übergang verursachen könnte. Chromosomale Instabilität Analyse zeigt, daß die MDM2
Gen amplifiziert wird. Nach einer gründlichen Strukturanalyse, einem TSC2 neuen Fusionsgens
-RNF216
identifiziert wird, die gleichzeitig tumorunterdrückende Bahnen stören kann und tumorigenesis aktivieren.
Schlussfolgerungen
Wir berichten über die genomische Profil von diffusem-Typ Magen Krebserkrankungen einschließlich neuer somatischen Variationen, einem neuen Fusionsgens und Verstärkung und Löschung bestimmter Chromosomenregionen, die Onkogene und Tumorsuppressoren.
Hintergrund
Magenkrebs gilt als die drittwichtigste Ursache der globalen Krebssterblichkeit [1] enthalten. Histopathologisch können Magenkrebs (GC) in zwei Kategorien eingeteilt werden, basierend auf morphologischen Unterschiede: Darm-Typ GC (IGC) und diffus-Typ GC (DGC) [2, 3]. IGC ist in der Regel mit Helicobacter pylori-Infektion assoziiert
und ist besonders häufig in Japan und Korea [4-6]. DGC gleichmäßig geografisch verteilten und enthält aggressive klinische Formen, wie Linitis plastica, die eine schlechte Prognose haben, vor allem bei jungen Patienten [7, 8]. Genomische DNA Modifikationen führen zu GC kann als Folge von mehreren Umweltrisikofaktoren wie hoher Salzdiät und Tabakrauchen passieren [9]. Obwohl die Häufigkeit von IGC stetig über mehrere Jahrzehnte (44% Reduktion von 1978 bis 2005) zurückgegangen ist, stieg DGC schnell (um 62%) von 1978 bis 2000, bevor etwas in den Jahren 2001-2005 abnimmt [10]. Trotz der kumulativen Hinweise darauf, dass IGC und DGC über verschiedene krebserregende Wege entwickeln [11, 12], eine detaillierte Genomskalendaten für DGC fehlen wegen der begrenzten Verfügbarkeit von klinischen Proben und einem niedrigen Niveau an Reinheit der Krebszellpopulation.
An Bislang sind nur wenige Gene im Zusammenhang mit GC-Subtypen sind identifiziert worden. Die CDH1
Gen, das das E-Cadherin-Protein kodiert, sind die bekanntesten Gene im Zusammenhang mit erblichen DGC (HDGC) [13-16]. Genetische Screening für diese Mutationen wurde, um zu diagnostizieren früh einsetzende GC [17] vorgeschlagen. E-Cadherin-Dysfunktion, verursacht durch Mutationen, den Verlust der Heterozygotie und Promotor-Hypermethylierung, ist das gut etablierte Defekt in GC Initiierung und Entwicklung [18-20]. Eine genomweite Assoziationsstudie zeigte, dass Polymorphismen in der Prostata-Stammzell-Antigen-Gen (PSCA
) sind stark mit der Anfälligkeit für DGC assoziiert [21]. Die Microarray-basierte Verfahren ist jedoch beschränkt auf Einzel-Nukleotid-Variationen und kann nicht Kopie neutrale Strukturvariationen (SVs) erkennen. Zwei neuere Studien über GC exomes berichtet, und zeigten, dass Mutationen in dem Gen ARID1A
häufig in GC mit Mikrosatelliten-Instabilität festgestellt werden, und in Epstein-Barr-Virus (EBV) -positiven GCs [22, 23]. Keine Analyse der GC-Subtypen durchgeführt wurde, und die Mehrzahl der Proben in den analysierten Studien stammten von Patienten mit IGC.
Next-Generation-Sequencing (NGS) hat Forscher krankheitsassoziierter Variationen erlaubt, und half aufzudecken, die zugrunde liegenden Mechanismen zu erkennen, der Krankheitsentwicklung. Insbesondere ganze Genom-Sequenzierung (WGS) können die meisten genomischen Variationen, einschließlich SVs, wie intrachromosomalen und interchromosomaler Umlagerungen erkennen. Alternativ ganze Exoms Sequenzierung (WES), ein aufgenommenes Zielsequenzierungsverfahren kann für hoch Tiefe Sequenzierung einer großen Anzahl von Proben, die bei relativ niedrigen Kosten [24] verwendet werden, obwohl nur einzelne Nukleotid-Variationen (SNVs) und kleine Insertionen oder Deletionen (indels) können unter Verwendung dieses Verfahrens identifiziert werden. WGS und WES haben jeweils Vor- und Nachteile, und eine Reihe neuerer Studien, beide Methoden verwendet haben [25-27].
Präsentieren wir hier detaillierte Charakterisierung von DGC Genome von angepassten Tumor und normalen Proben durch ganze genomische Profile von WES gefolgt Erzeugung . Wir verwendeten Blutproben als normale Kontrolle, wie in früheren Studien [28-31]. Um DGC spezifische Variationen zu finden, wurden IGC Genome auch mit Variationen in der Genome von DGCS identifiziert analysiert und verglichen. Dreidimensionale Proteinstrukturanalyse wurde für neuartige somatische Mutationen des CDH1
Gen durchgeführt, und diese kritischen Bereiche identifiziert, die durch die Mutationen funktionell verändert wurden. Darüber hinaus fanden wir eine neue Fusionsgens, das in der Tumorentstehung beteiligt sein könnten.
Ergebnisse und Diskussion
Genome und Exoms Sequenzierung
Tumor und abgestimmt normalen (Blut) Proben von 14 Patienten mit DGC (die klinisch-pathologischen Merkmale dieser Patienten in Tabelle S1 Weitere Datei 1) dargestellt sind, die alle relativ jung (Durchschnittsalter 38 Jahre waren) koreanischen Frauen, wurden unter Verwendung eines Illumina HiSeq 2000 sequenziert, die Paired-End produziert, 90-Basis und 101-Base-DNA liest. Zusätzlich fünf Paare von Tumor- und normalen abgestimmte Proben von Patienten mit IGC (Durchschnittsalter 42 Jahre) wurden einer DNA-Sequenzierung unterzogen wird; Eine dieser Proben wurde später als ein Fall von Mikrosatelliten-Instabilität (MSI), und somit wurde aus der Mutationsanalyse ausgeschlossen identifiziert. Keine der Proben hatten keine Familienanamnese von Krebs, und die Subtypen wurden histologisch bestätigt. Nur Tumorzellen durch macrodissection nach Hämatoxylin-Färbung wurden gesammelt.
Für die gesamte Genomanalyse im Durchschnitt 92 Gigabasen (Gb) pro Probe wurden mit etwa 32-mal Sequenzierungstiefe erzeugt, 3,5 Terabasen (Tb) insgesamt erreicht und waren abgebildet mit dem Referenzgenom (NCBI bauen 37, hg19) mit einer Abbildungsrate von mehr als 94,5% (für die Sequenzierung Statistiken finden Sie unter Weitere Datei 1: Tabelle S2). Mit dem abschließenden 3,3 Tb der abgebildeten liest, wurde eine genomische Profil-Datenbank zur Erfassung von SNVs aufgebaut, die Kopienzahl Varianten (CNVs) und SVs. Da die zelluläre Reinheit einer Tumorprobe ein kritisches Merkmal in der Krebsgenomanalyse ist, wurde es bewertet ein hauseigenes Berechnungsmethode (siehe Materialien und Methoden, siehe Zusatzdatei 1: Tabelle S3 und Abbildung S1). Obwohl wir nur versucht, Tumorzellen zu sammeln, unsere Proben zeigten nach wie vor ein hohes Maß an Stroma-Beimischung. Um die Genauigkeit der Detektion von Mutationen in genen Regionen sogar in geringer Reinheit Proben, zusätzliche WES erhöhen wurde bei etwa 103 mal Sequenzierungstiefe im Durchschnitt durchgeführt, die insgesamt 17 Gb Sequenzdaten erzeugt. Das erfasste WES 93,1% der gene Region bei 10-fache oder größere Tiefe bedeckt, und diese Abdeckung ist ähnlich zu der zuvor berichteten Exoms Daten über GC [22, 23].
Der WGS und WES Daten kombinierend, entdeckten wir somatischen Veränderungen in den DGC Proben, und sie mit den IGC Veränderungen verglichen (die Daten sind in Abbildung 1 als Zirkusdiagramm zusammengefasst). Um unsere Daten zu überprüfen, wir kombiniert und analysiert sie mit zuvor Exoms Daten aus zwei verschiedenen Studien (24 IGC und 5 DGC Proben, ohne MSI und Mischproben) [22, 23] und von Array vergleichende Genomhybridisierung (CGH) Daten ( 16 IGC und 14 DGC Proben) [32]. Obwohl diese Studien hauptsächlich RK verwendet und nur eine kleine Anzahl von DGC Proben enthalten, könnten sie als Kontrolle unseren Daten ergänzen (durch eine erhöhte Anzahl von IGC Daten und Beseitigung von Gewebespezifität bereitstellt). In der kombinierten Datensatzes verglichen wir die Unterschiede in den Veränderungen zwischen den DGC und IGC Proben. Abbildung 1 Genome Verteilung von somatischen Mutationen und Vervielfältigung oder Deletionsereignisse in diffuse-type Magenkarzinome (DGCS). Alle somatischen Mutationen, einschließlich Vervielfältigung /Löschen Ereignisse, die in den 14 DGC Genome gefunden wurden, sind in der Zirkusstück verschmolzen. Von außen nach innen, präsentiert die Handlung die folgenden Eigenschaften: Chromosom Ideogramme, die Häufigkeit der kumulativen Verstärkung oder Deletionsereignisse (schwarz, Verstärkung, rot, Löschen), und die Anzahl der somatischen nicht auch Einzel-Nukleotid-Variationen (nsSNVs), indels und SNVs in Spleißstellen für jedes Gen. Schwarze Dreiecke zeigen stark mutierten Gene. Orange Dreiecke bezeichnen Onkogene und blaue Dreiecke zeigen die Tumorsuppressoren.
Identifizierung von diffusen typspezifischen SNVs und indels
In jeder Probe Paar identifizierten wir etwa 3,7 Millionen SNVs, die Polymorphismus single nucleotide mit verifiziert wurden (SNP ) Chips (durchschnittliche Konkordanzrate: 99,2%; siehe Zusatzdatei 1: Tabelle S4) und ca. 0,69 Mio. indels (Details siehe Zusatzdatei 1: Tabelle S5 und Tabelle S6). Wir ersten Mutationsfrequenz der beiden Arten von GC an der Einzel-Nukleotid-Ebene bewertet wird (siehe Zusätzliche Datei 1: Abbildung S2 a, b). Die somatische Mutation Spektrum wurde von C >dominiert; T (G > A) Übergänge in sowohl der DGC und IGC Proben, und es gab keine signifikanten Unterschiede in mutational Kontexte zwischen den beiden GC-Typen, in Übereinstimmung mit früheren Studien von GC [23, 30]. Wenn wir zwei zuvor berichtet Exoms Datensätze analysiert, fanden wir, dass das Spektrum des Verhältnisses Nukleotidaustausches unseren Daten ähnlich war (siehe Zusätzliche Datei 1: Abbildung S2c, d).
Obwohl das Mutationsspektrum von DGC ähnlich ist von derjenigen IGC, einzelne Mutationen in den betroffenen Gene waren unterschiedlich. Durch Subtraktion im normalen Blut Genome gefunden Mutationen identifizierten wir 922 nicht gleichbedeutend SNVs (nsSNVs) als somatische Mutationen in den 18 Tumorproben (siehe Zusatzdatei 1: Tabelle S7, siehe Weitere Datei 2). Die durchschnittliche Mutationsrate der 18 GCs (1,97 Mutationen /Mb) war vergleichbar mit der in anderen Studien über Kolon berichtet, Pankreas- und Leberkrebs [33-35]. Von 847 mutierten Gene durch die 922 nsSNVs betroffen waren 581 in 14 Fällen DGC, 288 in 4 IGC Fälle waren, und 22 (2,6%) wurden auf beide Typen gemeinsam. Die MSI-Probe, die aus der vergleichenden Analyse zeigte, dass etwa sechs Mal mehr SNVs und indels als taten die anderen Proben ausgeschlossen wurde; Dieses Ergebnis ist in Übereinstimmung mit einem früheren Bericht [22]. Wenn wir die beiden zuvor berichteten Exoms Datensätze kombiniert, identifizierten wir 967 und 2077 somatischen nsSNVs in 19 DGCS und 28 RK sind. Die somatischen Mutationsrate des RK (3,71 Mutationen /Mb in den 28 Proben) war höher als die der DGCS (2,29 Mutationen /Mb in den 19 Proben) (siehe Zusatzdatei 1: Tabelle S8). Bisher veröffentlichte Untersuchungen zeigen, dass Melanom und Lungenkrebs hohe Mutationsraten haben, aufgrund der Beteiligung von potenten mutagen [36]. Ebenso ist es möglich, dass IGC diese hohe Mutationsrate hat, weil seine tumorigenen Mechanismus mehr in Verbindung gebracht werden kann mit Umwelt- und /oder parasitären mutagene verglichen mit DGC.
Für individuelle Variationen wurden mutmaßliche Krebs-verursachende Gene durch Gen-Treiber Score-Berechnung vorhergesagt (siehe Zusätzliche Datei 1: Tabelle 1 und Tabelle S9). Das Gen
CDH1 wurde gefunden in DGC werden reichlich mutiert (P
= 1,29 x 10
-2), darunter sechs somatische Mutationen (drei Missense, ein Unsinn, ein Frameshift und eine Spleißstelle Mutationen) , die noch nicht berichtet worden, während nur eine Missense-Mutation in den IGC-Proben (Tabelle 2) gefunden wurde. Alle sieben CDH1
somatische Mutationen durch Sanger-Sequenzierung überprüft wurden (siehe Zusatzdatei 1: Tabelle S10 und Tabelle S11). In unseren DGC Proben, 35,7% (5/14) hatten somatische Mutationen
CDH1, und es wurde berichtet, dass die Frequenzen der CDH1
somatische Mutationen in sporadischen DGCS von 3% bis mehr als 50% variieren kann [19 , 37-40]. Es wurde in den Ländern mit einer hohen Inzidenz von sporadischen GC (wie Japan und Korea), die Häufigkeit von Keimbahnmutationen in familiärer GCs niedrig ist im Vergleich zu dem in Low-Inzidenz Länder [41, 42] bestätigt, dass. Daher spekulieren wir, dass die Gesamt GC Einfalls auch auf die Frequenz der CDH1
somatische Mutationen in Beziehung steht. Zusätzlich wurde eine Keimbahnmutation (T340A) in CDH1
in beiden Tumor und entsprechende Blut Genome von zwei Proben (; M-01, MSI-Typ D-14, DGC) gefunden. Obwohl T340A eine ursächliche Mutation in HDGC ist [43], wobei diese zwei Patienten hatten keine Familiengeschichte wie GC oder lobuläres Mammakarzinom. Zwei frühere Berichte Exoms Daten von GC Analyse nicht identifizieren CDH1
als hoch eingestuft Gen (nur eine Missense-Mutation in einer MSI-IGC Probe) [22, 23]. Diese Diskrepanz kann auf die kleine Anzahl von Proben von DGC in diesen Studien (2 von 22 und 3 von 15 Proben waren DGCS, respectively) fällig. In der vorliegenden Arbeit, PIK3CA
und TP53
bekannte Gene Krebs-assoziierten, waren die am häufigsten mutierten Gene in beiden DGC und IGC siehe Tabelle 1 und Tabelle S9 Weitere Datei 1. Mutationen in zwei bekannten PIK3CA
Hotspots (E545K und H1047L) wurden in vier DGC Proben gefunden. Zusätzlich wurde eine nsSNV Mutation (Q546K) neben der E545K-Mutation in einem DGC Probe gefunden. Insgesamt 5 von 14 DGC Proben (ca. 30%) beherbergte nsSNVs in PIK3CA
, die ein Onkogen mutiert ist, dessen Form eine erhöhte Kinase-Aktivität, Krebszellproliferation verursacht [44]. Wir verglichen dann die niedrige Frequenz (16-17%) der nsSNVs in PIK3CA
in Berichte von anderen [22, 23, 44] (die meist gebrauchte IGC Proben) und die Ergebnisse unserer kombinierten Analyse (31,5% für DGC , 14,3% für die IGC) (siehe Zusatzdatei 1: Tabelle S9). Es scheint, dass die relativ hohen Mutationsraten von PIK3CA
in DGC kann die Spezifität von Mutationen in diesem Gen zu dieser Art von Krebs zu reflektieren. Zusätzlich wurden drei Proben (zwei DGC und ein IGC) enthielt sowohl nsSNV und eine Kopie Verlust TP53
, einen homozygoten Verlust der Funktion in TP53
angibt, wie früher berichtet [45]. Ein SNP in dem PSCA
Gens (rs2976329) wurde berichtet, mit einem erhöhten Risiko von DGC in Japanisch und Koreanisch Populationen [21] in Verbindung gebracht werden. Diese SNP wurde auch in der Mehrzahl der Proben DGC in unserer Studie angereichert (9 von 14 Patienten), was darauf hinweist, dass unsere analysierten Proben typischen Patienten mit DGC in East Asia repräsentieren. Zusätzlich wurde eine Nonsense-Mutation (R1446 *) im ARID1A
Gen wurde in einer DGC Probe (D-08) gefunden. Obwohl Mutationen in ARID1A
häufig in MSI erkannt und in EBV-positiven GCs [22, 23], die D-08 Probe zeigte keine EBV-Infektion und ein MSI-Probe (M-01) hatte keine ARID1A haben
Gen-Mutationen entweder. Von Variationen in den Kandidaten Fahrer Gene, 88 nsSNVs, 4 kleine indels und 2 SNVs in einer Spleißstelle wurden unter Verwendung von herkömmlichen Sanger-Sequenzierung verifiziert. Sieben dieser Mutationen konnte wegen PCR Versagen getestet werden, und der verbleibenden 87 Mutationen wurden 96,6% als echte somatische Mutationen bestätigt wird (siehe Zusätzliche Datei 1: Tabelle S10 und Tabelle S11) .Tabelle 1 Top Kandidaten Fahrer Gene in 14 diffuse -Typ Magenkrebs
Gene
Proben, n
nsSNVs, n
SNVs in Spleißstelle, n
Indels, n
P
-Wertes
Treiber Gen-Score
PIK3CA
5
5
0
0
3,63 × 10 -12
9,83
CDH1
5
4 1
 Fundoplikatio (Antirefluxchirurgie)
Fundoplikatio (Antirefluxchirurgie)
 SIBO-freundliches Valentinstagsessen
SIBO-freundliches Valentinstagsessen
 Nahrungsergänzungsmittel können Leberschäden verursachen. Weitere Forschung und Regulierung sind erforderlich
Nahrungsergänzungsmittel können Leberschäden verursachen. Weitere Forschung und Regulierung sind erforderlich
 Wann man einen Stuhlweichmacher oder ein Abführmittel verwendet
Wann man einen Stuhlweichmacher oder ein Abführmittel verwendet
 Ursachen und Risikofaktoren für Speiseröhrenkrebs
Ursachen und Risikofaktoren für Speiseröhrenkrebs
 Übergewicht verdoppelt Ihre Chancen auf einen Schlaganfall
Übergewicht verdoppelt Ihre Chancen auf einen Schlaganfall
 Minderheitspatienten in der Notaufnahme erhalten mit geringerer Wahrscheinlichkeit Schmerzmittel gegen Bauchschmerzen
Neueste Nachrichten über chronische Schmerzen Astronauten mit dem Risiko einer „Weltraumanämie“ Könnte Gentherapie helfen, die Sichelzellanämie zu heilen? FDA genehmigt Virtual-Reality-System zur Lin
Minderheitspatienten in der Notaufnahme erhalten mit geringerer Wahrscheinlichkeit Schmerzmittel gegen Bauchschmerzen
Neueste Nachrichten über chronische Schmerzen Astronauten mit dem Risiko einer „Weltraumanämie“ Könnte Gentherapie helfen, die Sichelzellanämie zu heilen? FDA genehmigt Virtual-Reality-System zur Lin
 Wissenschaftler entwickeln einen Ansatz zur Impfung gegen Darmentzündungen
Entzündliche Darmerkrankung (IBD) ist ein Überbegriff, der viele Erkrankungen beschreibt, die eine chronische Entzündung des Verdauungstraktes beinhalten. einschließlich Colitis ulcerosa und Morbus Cr
Wissenschaftler entwickeln einen Ansatz zur Impfung gegen Darmentzündungen
Entzündliche Darmerkrankung (IBD) ist ein Überbegriff, der viele Erkrankungen beschreibt, die eine chronische Entzündung des Verdauungstraktes beinhalten. einschließlich Colitis ulcerosa und Morbus Cr
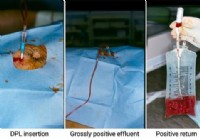 Wie wird eine Peritonealspülung durchgeführt?
Was ist Peritonealspülung? Die diagnostische Peritonealspülung wird bei instabilen Patienten angewendet, die ein schweres körperliches Trauma erlitten haben. Peritonealspülung ist ein bettseitig
Wie wird eine Peritonealspülung durchgeführt?
Was ist Peritonealspülung? Die diagnostische Peritonealspülung wird bei instabilen Patienten angewendet, die ein schweres körperliches Trauma erlitten haben. Peritonealspülung ist ein bettseitig