Genomisk profilanalyse av diffus-type mage kreft
Abstract
Bakgrunn
Magekreft er den tredje dødeligste blant alle krefttilfeller på verdensbasis. Selv om forekomsten av tarmtypen magekreft har avtatt, blir forekomsten av diffust-type fortsetter å stige og dens progresjon er notorisk aggressiv. Det er ikke tilstrekkelig informasjon om genom varianter av diffus type magekreft fordi cellene er vanligvis blandet med normale celler, og denne lave cellularity har gjort det vanskelig å analysere genomet.
Resultater
Vi analyserer hele genomer og tilsvarende exomes diffus-type magekreft, ved hjelp matchet tumor og normale prøver fra 14 diffus-type og fem intestinal-type magekreftpasienter. Somatiske variasjoner som finnes i den diffuse-type magekreft er sammenlignet med de til intestinal-type og til tidligere rapportert varianter. Vi bestemme den gjennomsnittlige exonic somatisk mutasjon hastighet av de to typene. Vi finne assosiert kandidat driver gener, og identifisere syv nye somatiske mutasjoner i CDH1
, som er et velkjent magekreft-assosierte genet. Tredimensjonal struktur analyse av det muterte E-cadherin-proteinet antyder at disse nye somatiske mutasjoner kan forårsake betydelige funksjonelle forstyrrelser av kritiske kalsium-bindingsseter i EC1-2 krysset. Kromosomal instabilitet Analysen viser at MDM2
genet er forsterket. Etter grundig strukturell analyse, en roman fusjonsgenet TSC2
-RNF216
er identifisert, noe som samtidig kan forstyrre tumor-undertrykkende stier og aktivere tumorigenesis.
Konklusjoner
Vi rapportere genomisk profilen til diffuse-type mage kreft, inkludert nye somatiske varianter, en roman fusjonsgenet, og forsterkning og sletting av visse kromosom regioner som inneholder onkogener og tumor dempere.
Bakgrunn
mage kreft rangert som den tredje viktigste årsaken til global kreftdødelighet [1]. Histopathologically, kan magekreft (GC) deles inn i to kategorier basert på morfologiske forskjeller: intestinal-type GC (IGC) og diffus-type GC (DGC) [2, 3]. IGC er vanligvis assosiert med Helicobacter pylori
infeksjon, og er spesielt vanlig i Japan og Korea [4-6]. DGC er jevnt geografisk fordelt, og omfatter aggressive kliniske former, så som linitis plastica, som har dårlig prognose, spesielt hos unge pasienter [7, 8]. Genomiske DNA modifikasjoner som fører til GC kan skje som et resultat av flere miljømessig risikofaktorer, slik som en høy-salt diett og tobakksrøyking [9]. Selv om forekomsten av IGC har sunket jevnt over flere tiår (44% reduksjon fra 1978 til 2005), økt DGC hurtig (ved 62%) fra 1978 til 2000, før minkende svakt i 2001-2005 [10]. Til tross for den akkumulerte bevis for at IGC og DGC utvikle seg via forskjellige baner karsinogene [11, 12], detaljert genomisk skala data for DGC mangler på grunn av begrenset tilgjengelighet av kliniske prøver og et lavt nivå av renhet av kreftcellepopulasjon.
Å Hittil har svært få gener assosiert med GC subtyper blitt identifisert. Den CDH1
-genet, som koder for det E-cadherin protein, er de best kjente gener assosiert med arvelig DGC (HDGC) [13-16]. Genetisk screening for disse mutasjoner er blitt foreslått for å diagnostisere tidlig innsettende GC [17]. E-cadherin dysfunksjon, forårsaket av mutasjoner, tap av heterozygositet og promoter hypermethylation, er den mest veletablert defekt i GC initiering og utvikling [18-20]. Et genom-wide forening studie viste at polymorfismer i prostata stamcelle-genet (PSCA
) er sterkt forbundet med følsomhet for DGC [21]. Den microarray-baserte metoden er imidlertid begrenset til single nucleotide variasjoner, og kan ikke gjenkjenne kopi nøytral strukturelle variasjoner (SVS). To nylige studier har rapportert på GC exomes, og viste at mutasjoner i ARID1A
genet blir ofte detektert i GC med mikro ustabilitet, og i Epstein-Barr virus (EBV) -positivt GCer [22, 23]. Ingen analyse av GC subtyper ble utført, og de fleste av de analyserte prøvene i studiene var fra pasienter med IGC.
Neste generasjons sekvensering (NGS) har tillatt forskere å oppdage sykdomsassosierte varianter, og hjalp avdekke de underliggende mekanismene av sykdomsutvikling. Spesielt kan hele genomet sekvensering (WGS) påvise de fleste genomiske variasjoner, deriblant SVs, slik som intrachromosomal og interchromosomal rearrangementer. Alternativt kan hele exome sekvensering (WES), et innfanget-target-sekvenseringsmetode, kan anvendes for høy-dybde-sekvensering av et stort antall prøver på en forholdsvis lav kostnad [24], selv om bare én nukleotidvariasjoner (SNVs) og små innsett eller slettinger (indels) kan identifiseres ved hjelp av denne metoden. WGS og WES hver har fordeler og ulemper, og en rekke nyere studier har brukt begge metoder [25-27].
Her presenterer vi detaljert karakterisering av DGC genomer fra matchet tumor og normale prøver ved å generere hele genomisk profiler etterfulgt av WES . Vi brukte blodprøver som en normal kontroll, som i tidligere studier [28-31]. For å finne DGC spesifikke variasjoner, ble IGC genomer også analysert og sammenlignet med variasjoner identifisert i genomene til DGCs. Tredimensjonale proteinstruktur-analyse ble utført for nye somatiske mutasjoner av CDH1
-genet, og dette identifiserte kritiske områder som er funksjonelt forandret av mutasjonene. I tillegg fant vi en ny fusjonsgenet som kan være involvert i tumordannelse.
(Resultater og diskusjon
Whole genome og exome sekvense
Tumor og matchet normale (blod) prøver fra 14 pasienter med DGC de clinicopathological egenskaper av disse pasientene er vist i tabell S1 igjen fil 1), som alle var relativt unge (median alder 38 år) koreanske kvinner, ble sekvensert ved hjelp av en Illumina HiSeq 2000, som produserte sammen-end, 90-base og 101-basen DNA leser. I tillegg ble fem par svulst og matchet normale prøver fra pasienter med IGC (median alder 42 år) er utsatt for DNA-sekvensering; en av disse prøvene ble identifisert senere som et tilfelle av mikro ustabilitet (MSI) og dermed ble ekskludert fra mutasjon analyse. Ingen av prøvene hadde noen familiær historie av kreft, og subtypene ble histopathologically bekreftet. Bare kreftceller ble innhentet av macrodissection etter hematoxylin farging.
For hele genomanalyse, i gjennomsnitt 92 gigabases (Gb) per prøve ble produsert på omtrent 32 ganger sekvense dybde, og nådde 3,5 terabases (TB) totalt, og var kartlagt til referanse genomet (NCBI bygge 37, hg19) ved en kartlegging hastighet større enn 94,5% (for sekvense statistikk, se tilleggsfiler 1: Tabell S2). Bruke den endelige 3.3 Tb av kartlagt leser, en genomisk profildatabase ble konstruert for å detektere SNVs, kopiere antall varianter (CNVs), og SVs. Fordi den cellulære renheten av en tumorprøve er en kritisk funksjon i kreftgenomanalyse, ble det evaluert med en in-house beregningsmetoden (se materialer og metoder, se tilleggsfiler 1: Tabell S3 og figur S1). Selv om vi forsøkt å samle bare tumorceller, våre prøver viste fortsatt et høyt nivå av stromal blanding. For å øke nøyaktigheten av mutasjonsdeteksjon i fremkallende regionene selv i lav renhet prøvene, ble ytterligere WES utført ved ca 103 ganger sekvense dybde i gjennomsnitt, som produserte totalt 17 Gb sekvensdata. WES det innfangede omfattet 93,1% av den kallende regionen på 10 ganger eller større dybde, og denne dekningen er lik den for tidligere rapporterte exome data på GC [22, 23].
Kombinere det WGS og WES data, vi har oppdaget somatisk endringer i DGC prøvene, og sammenlignet dem med IGC endringer (dataene er oppsummert i figur 1 som et sirkus diagram). For å verifisere våre data, vi kombinert og analysert dem med tidligere rapportert exome data fra to ulike studier (24 IGC og 5 DGC prøver, ikke inkludert MSI og blandede prøver) [22, 23] og fra utvalg komparativ genom hybridisering (CGH) data ( 16 IGC og 14 DGC prøver) [32]. Selv om disse studiene brukes hovedsakelig IGCs, og med bare et lite antall DGC prøver, kan de være komplementære til de data som en kontroll (ved å tilveiebringe et økt antall av IGC data og fjerning av vev-spesifisitet). I det kombinerte datasettet, sammenlignet vi forskjellene i endringer mellom de DGC og IGC prøver. Figur 1 Hele genomet distribusjon av somatiske mutasjoner og duplisering eller sletting hendelser i diffuse-type mage kreft (DGCs). Alle de somatiske mutasjoner, inkludert kopiering /sletting hendelser, som ble funnet i de 14 DGC genomer, er slått sammen i sirkuset tomten. Fra utsiden til innsiden, presenterer tomten følgende egenskaper: kromosom ideograms, hyppighet av kumulative forsterkning eller sletting hendelser (svart, forsterkning, rødt, sletting), og antall somatiske ikke-synonyme single nucleotide variasjoner (nsSNVs), indels, og SNVs i spleiseseter for hvert gen. Svarte trekanter indikerer sterkt muterte gener. Oransje trekanter betegne onkogener og blå trekanter viser tumorundertrykkere.
Identifikasjon av diffus-typespesifikke SNVs og indels
I hver prøve par, vi identifisert ca 3,7 millioner SNVs, som ble bekreftet ved hjelp enkeltnukleotidpolymorfi (SNP ) chips (gjennomsnittlig samsvar rente: 99,2%; se tilleggsfiler 1: Tabell S4), og ca 0.69 million indels (for detaljer, se tilleggsfiler 1: Tabell S5 og Tabell S6). Vi vurderte første mutasjonsfrekvensen for begge typer av GC på enkelt-nukleotid-nivå (se Ytterligere fil 1: Figur S2 a, b). Den somatisk mutasjon spekteret ble dominert av C > T (G > A) overganger i både DGC og IGC prøver, og det var ingen signifikante forskjeller i mutasjons sammenhenger mellom de to GC typer, i overensstemmelse med tidligere studier av GC [23, 30]. Når vi analyserte to tidligere rapportert exome datasett, fant vi at spekteret av nukleotidsubstitusjon forholdet var lik våre data (se tilleggsfiler 1: Figur S2C, d).
Selv om mutasjonen spekteret av DGC er lik som IGC, individuelle mutasjoner i berørte gener var annerledes. Ved å trekke mutasjoner funnet i normale blod genomer, identifiserte vi 922 ikke-synonyme SNVs (nsSNVs) som somatiske mutasjoner i de 18 tumorprøver (se tilleggsfiler 1: Tabell S7, se tilleggsfiler 2). Den gjennomsnittlige mutasjonsraten av de 18 GCer (1,97 mutasjoner /Mb) var sammenlignbar med den som er rapportert i andre studier på tykktarm, bukspyttkjertel og lever kreft [33-35]. Av 847 muterte gener påvirkes av 922 nsSNVs, 581 var i 14 DGC tilfeller, 288 var i 4 IGC tilfeller, og 22 (2,6%) var felles for begge typer. MSI prøven, som ble ekskludert fra den komparative analysen, viste omtrent seks ganger mer SNVs og indels enn gjorde de andre prøvene; Dette resultatet er i samsvar med en tidligere rapport [22]. Når vi kombinert de to tidligere rapporterte exome datasett, identifiserte vi 967 og 2,077 somatiske nsSNVs i 19 DGCs og 28 IGCs hhv. Somatiske mutasjonshastigheten av IGCs (3,71 mutasjoner /Mb i de 28 prøver) var høyere enn den til DGCs (2,29 mutasjoner /Mb i de 19 prøver) (se Ytterligere fil 1: Tabell S8). Tidligere publisert forskning tyder på at melanom og lungekreft har høy mutasjon priser, på grunn av involvering av potente mutagens [36]. Likeledes er det mulig at IGC dette har høy mutasjonshastighet fordi det er tumorigen mekanisme kan være assosiert med mer miljø- og /eller parasittiske mutagener sammenlignet med DGC.
For individuelle variasjoner, ble putative kreft-forårsakende gener som forutsagt av sjåføren gen stillingen beregning (se tilleggsfiler 1: Tabell 1 og Tabell S9). Den CDH1
-genet ble funnet å være rikelig mutert i DGC (P
= 1,29 x 10
-2), inkludert seks somatiske mutasjoner (tre missense, en nonsense, en rammeskifte, og ett spleise sete-mutasjoner) som ikke har vært rapportert tidligere, mens bare en missense mutasjon ble funnet i IGC prøvene (tabell 2). Alle sju CDH1
somatiske mutasjoner ble verifisert ved Sanger-sekvensering (se tilleggsfiler 1: Tabell S10 og Tabell S11). I våre DGC prøver, 35,7% (5/14) hadde CDH1
somatiske mutasjoner, og det har blitt rapportert at frekvensene av CDH1
somatiske mutasjoner i sporadiske DGCs kan variere fra 3% til mer enn 50% [19 , 37-40]. Det ble bekreftet at i land med en høy forekomst av sporadisk GC (for eksempel Japan og Korea), er frekvensen av kimlinje-mutasjoner i familiær GCer lavt sammenlignet med det i lav forekomst land [41, 42]. Derfor spekulere vi at den samlede GC forekomst er også relatert til frekvensen for CDH1
somatiske mutasjoner. I tillegg ble en germline mutasjon (T340A) i CDH1
finnes i både tumor og tilsvarende blod genomer fra to prøver (D-14, DGC, M-01, MSI-type). Selv T340A er en utløsende mutasjon i HDGC [43], gjorde disse to pasientene ikke har noen familiær historie som GC eller lobular brystkreft. To tidligere rapporter analyse exome data fra GC ikke identifisere CDH1
som en høyt rangert gen (bare en missense mutasjon i et MSI IGC prøve) [22, 23]. Denne uoverensstemmelse kan skyldes det lille antallet prøver av DGC i disse studiene (2 ut av 22 og 3 av 15 prøver var DGCs, henholdsvis). I dette arbeidet, PIK3CA Hotell og TP53
, kjente kreftassosierte gener, var de hyppigst muterte gener i både DGC og IGC se tabell 1 og tabell S9 i tilleggsfiler 1. Mutasjoner i to kjente PIK3CA
hotspots (E545K og H1047L) ble funnet i fire DGC prøver. I tillegg ble en nsSNV mutasjon (Q546K) tilstøtende til E545K mutasjonen funnet i en DGC prøven. I alt 5 ut av 14 DGC prøver (ca. 30%) hadde nsSNVs i PIK3CA
, som er et onkogen som har mutert skjema oppviser øket kinase-aktivitet og forårsaker kreft-celleformering [44]. Vi deretter sammenlignet med lav frekvens (16-17%) av de nsSNVs i PIK3CA
i rapporter fra andre [22, 23, 44] (som stort sett brukte IGC prøver) og resultatene av vår samlede analyse (31,5% for DGC , 14,3% for IGC) (se tilleggsfiler 1: Tabell S9). Det ser ut til at de relativt høye mutasjonsrater PIK3CA
i DGC kan reflektere spesifisiteten av mutasjoner i dette genet til denne type kreft. I tillegg tre prøver (to DGC og en IGC) inneholdt både nsSNV og en kopi tap av TP53
, noe som indikerer en homozygot tap av funksjon i TP53
, som tidligere rapportert [45]. En SNP i PSCA
genet (rs2976329) har blitt rapportert å være assosiert med økt risiko for DGC i japansk og koreansk populasjoner [21]. Denne SNP ble også beriket i de fleste DGC prøver i vår studie, (9 av 14 pasienter), noe som indikerer at våre analyserte prøvene representerer typiske pasienter med DGC i Øst-Asia. I tillegg har en nonsense-mutasjon (R1446 *) i den ARID1A
-genet, ble funnet i en DGC prøve (D-08). Selv om mutasjoner i ARID1A
ofte oppdages i MSI og EBV-positive GCer [22, 23], D-08 prøven viste ingen EBV infeksjon, og en MSI prøve (M-01) ikke har noen ARID1A
genmutasjoner heller. Fra variasjoner i kandidat driver gener, 88 nsSNVs, 4 små indels og 2 SNVs i et spleisesetet ble bekreftet ved hjelp konvensjonell Sanger-sekvensering. Sju av disse mutasjonene ble ikke testet på grunn av PCR svikt, og av de resterende 87 mutasjonene, ble 96,6% bekreftet som ekte somatiske mutasjoner (se tilleggsfiler 1: Tabell S10 og Tabell S11) .table en toppkandidat driver gener i 14 diffus -type mage kreft
Gene
Samples, n
nsSNVs, n
SNVs i spleisesete, n
Indels, n
P
-verdi
Driver genet poengsum
PIK3CA
5
5
0
0
3,63 × 10 -12
9.83
CDH1
5
4
en
1 4.64 × 10-10
8,02
SNRPN
2
2
0
0
1.86 × 10-07
5,60
TP53
2
2
0
0
4,88 × 10-07
5,36
CMKLR1
2
2
0
0
5.33 × 10-07
5,36
CYP2A7
2
2
0
0
1.53 × 10-06
4,99
GUCY1B3
2
2
0
0
1.97 × 10-06
4,99
PAPOLB
2
2
0
0
2.15 × 10-06
4.99
MYH9
3
3
0
0
2.27 × 10-06
4,99
FAM71B
en
2
0
0
2.51 × 10-06
4,99
C10orf90
2
2
0
0
3,76 × 10 -06
4.86
AKAP8
2
2
0
0
4.59 × 10-06
4,81
ZC3H12B
2
2
0
0
5.87 × 10-06
4,74
SFTA3
en
1 0
0
6.86 × 10-06
4,70
SENP7
2
2
0
0
7.65 × 10-06
4,68
TMPRSS6
2
2
0
0
8.38 × 10-06
4.67
side2
en
1 0
0
9.94 × 10-06
4,62
For ytterligere driver genet lister, se tilleggsfiler. 1: Tabell S9
Tabell 2 CDH1 endringer i 18 mage kreft
Sample
Skriv
Endring
CDH1
region
D-01 T
CNV
Loss
Eksoner 1-16
D-02 T
SNV
N256S
Exon 6
CNV
Loss
Eksoner 1-16
D-03 T
SNV
Splice nettstedet
Donor stedet Intron 4
D-04 T
CNV
Loss
Eksoner 1-16
D-05 T
SNV
D257N
Exon 6
INS
S829fs
Exon 16
D-09 T
SNV
V252G
Exon 6
SV
Break punkt
Intron 2
D -10 T
CNV
Loss
Eksoner 1-16
D-11 T
CNV
Loss
Eksoner 1-16
D-12 T
SNV
Q23 *
Exon 2
D-13 T
CNV
Loss
Eksoner 1-16
SV
Break punkt
Introner 2 og 10
D-14 T
SV
Break punkt
Introner 2, 5 og 9
I-01 T
CNV
Loss
Eksoner 1-16
jeg -02 T
CNV
Loss
Eksoner 1-16
I-03 T
SNV
D221G
Exon 5
SV
Break punkt
introner 10 og 13
I-04 T
CNV
Loss
Eksoner 1-16
CNV, kopiere nummer variasjon; INS, liten innføring; SNV, single nucleotide variasjon; SV, strukturell variasjon.
Den somatiske varianter ble deretter kartlagt på Kyoto Encyclopedia of gener og genomer (KEGG) trasé database. Denne analysen viste at de muterte gener DGCs var signifikant assosiert med kalsiumsignalveien (P
= 7,00 × 10 -5, se tilleggsfiler 1: Tabell S12 og Tabell S13). Lavt kalsiuminntak kan bidra til utvikling GC [46]. Kalsium er viktig for funksjonen av E-cadherin, og et tap av E-cadherin-mediert adhesjon er involvert i overgangen fra et godartet lesjon til invasiv metastatisk kreft [47]. Videre er de somatiske mutasjoner ble sterkt assosiert med trasé knyttet til småcellet lungekreft (P
= 1,00 x 10 -6 i DGC og P
= 4,24 x 10 -2 i IGC). Spesielt gener involvert i fokale heft trasé som ITGA
, PIK3CA
, og PTEN
, ofte ble mutert.
SV og CNV analyse
SVs ble påvist basert på discordantly kartlagt lese par, og eventuelle SVs som var til stede i pasientenes germline genomer ble ekskludert. I gjennomsnitt, fant vi 552 somatiske SVs per DGC prøve par (211 store innskudd, 264 store strykninger, 27 inversjoner, 44 intrachromosomal trans og 6 interchromosomal trans). Vi fant 664 somatiske SVs i hvert IGC prøve par (285 store innskudd, 283 store slettinger 34 inversjoner, 38 intrachromosomal trans og 24 interchromosomal trans) (for detaljer for hver prøve, se om tilleggsfiler 1: Tabell S14 og figur S3). I tillegg fant vi 2,258 gener for å bli svekket, og 1736 av disse ble funnet bare i DGC prøver (for data for hver prøve, se om tilleggsfiler 1: Tabell S15, og se om tilleggsfiler 3). Tre tumorsuppressorgener FHIT
, WWOX
, og MIPOL1
, som ble rapportert i en tidligere GC studie [30], hadde nedsatt på grunn av SVs (FHIT
i 11 prøver, WWOX
i 5 prøver, og MIPOL1
i 3 prøver)
Fusion gener som genereres av en kromosom omorganisering ble også analysert, og 19 fusjonsgenet kandidater ble identifisert (se tilleggsfiler 1: Tabell S16), inkludert en ny fusjon gen, TSC2
-RNF216
, som finnes i en prøve (figur 2a, b). TSC2
koder for tuberin protein ble tidligere foreslått som en tumor suppressor gen involvert i mammalian target of rapamycin (mTOR) pathway [48, 49]. I tillegg er RNF216
, kodings E3 ubiquitin-protein-ligase, er involvert i cytokin-funksjon ved å forhindre vedvarende aktivering av nukleær faktor (NF) -κB [50]. Den Rap GTPase-aktiverende protein (RAP-GAP) domenet av TSC2 protein, som er knyttet til den iboende GTPase aktivitet av Ras-relaterte proteiner RAP1A og RAB5, ble brutt ved denne translokasjon (figur 2c). I tillegg ble sinkfinger domenene til RNF216 protein ikke uttrykkes i fusjonsgenet, på grunn av en rammeskift som forårsakes for tidlig terminering. Ved hjelp av revers transkripsjon polymerasekjedereaksjon (RT-PCR), etterfulgt av sekvensanalyse, ble ekspresjonen av dette fusjonsgenet i pasientens vev bekreftet. Etter å ha testet et ekstra sett med 15 GC pasient vev, identifiserte vi 2 pasienter uttrykker fusjonsgenet (figur 2d, e). Denne translokasjon kan føre til endret cellulær oppførsel både ved å forstyrre den normale funksjon av genet, og som forårsaker ekspresjon av fusjonsgenet produkt, som kan konkurrere mot det normale genet. Fusjonsgenet kan kompetitivt forstyrre tumorsupressorproteinene trasé og aktiverer NF-kB-mediert cytokin signalering. Figur 2 TSC2 - RNF216 fusjonsgenet brekkasje. (A) Exon strukturen i TSC2
-RNF216
fusjonsgenet. Tallene i boksene er ekson antall av hvert gen. Røde linjer angir fusjons poeng. (B) Protein domene struktur av TSC2-RNF216 fusjonsprotein. Rap-GAP domenet TSC2 ble brutt, og RNF216 hadde en rammeskifte mutasjon som forårsaker for tidlig avslutning av interchromosomal omorganisering. (C) Oppbygging av TSC2 Rap-GAP domene. Den røde regionen er de resterende Rap-GAP domene regionen, og den grå regionen er Rap-GAP domene som er slettet i TSC2
-RNF216
fusjonsgenet. (D) RNA sekvens av TSC2
-RNF216
fusjonsgenet. Posisjon 136 er vist som N. enten A eller G basen produserer et termineringskodon (TAA eller TAG). (E) Verifisering av TSC2
-RNF216
fusjonstranskript i RNA (cDNA) ved hjelp av PCR-amplifikasjon og elektroforese.
I DGCs, kromosomer 16, 17, 19, 20, 21, og 22 inneholdt en økt mengde av blokk-slettinger, mens kromosomer 3, 7, 8, og 13 viser særlig økt duplikasjoner (figur 1). Mange tumorsuppressorgener, slik som CDH1
, PLA2G2A, RUNX3
, SMAD2
, og TP53
, ligger i stor utstrekning slettet kromosomale regioner. Spesielt, somatisk mutasjon (nsSNV eller spleisesete mutasjon) og kopiantall tap av CDH1
var generelt gjensidig utelukkende: fire av fem DGC prøver med somatisk mutasjon hadde ikke genkopitallet tap, og åtte av ni DGC prøver med en CDH1
genkopitallet tap ikke har noen somatiske mutasjoner i CDH1
. Bare én prøve (1/18, 5,6%) hadde både endringer (mutasjoner og kopiere antall tap) samtidig, og denne observasjonen sammenfaller med tidligere studier rapporterer at samtidige endringer i CDH1
er sjeldne [19, 40, 51, 52] . Når vi betraktet SVs i CDH1
sammen, fant vi at andre tre prøver hadde en mutasjon /kopiantall tap samtidig med SV. I tillegg er kopiantall av onkogen MYC
ble økt i fem DGC prøver (se tilleggsfiler 4), og kopiere antall MET
ble økt i tre DGC prøver [53]. De onkogener MOS
og ZHX2
også viste en kopi nummer gevinst på fem og fire DGC prøver, henholdsvis. Mer enn halvparten av prøvene (10 av 18) viste et kopiantall reduksjon av ARID1A
, som er en driver gen for ovarial klar celle karsinom, og en kromatin remo i GC [22, 54, 55]. Det er kjent at flertallet av GCer med ARID1A
mutasjoner viser lavere proteinuttrykk sammenlignet med GCer uten ARID1A
mutasjon [22]. Hvis doseringen effekt er viktig i disse kreftvev, kopierer antall reduksjon av ARID1A
kan være en mulig cancer-assosiert faktor, En stor region av kromosom 12 ble amplifisert i tre DGC genomer.; av disse tre genomer, ble prøver D-01 T og D-02 t viste utpreget høy forsterkning (figur 3a). Duplisering mønstrene var litt annerledes: D-01 T hadde en tandem duplisering av tre MBP, mens D-02 T hadde en omvendt duplisering av en MBP (figur 3b, c). En del av denne dupliseres regionen koder murine dobbel minutt (MDM2
) genet. Det ble rapportert at i en liten datasettet, ble MDM2
-genet ofte amplifisert [56], og at dette genet er assosiert med flere typer kreft [57]. MDM2
overekspresjon forårsaket av genamplifikasjon ble eksperimentelt bekreftet ved bruk av kvantitativ RT-PCR med tumoren og tilstøtende normalt vev sammenkoblet prøver som brukes for NGS analyser, og normale cellelinjer ble inkludert for sammenligning (figur 3d). MDM2
overekspresjon positivt korrelert med kopi nummer analyse av data. Selv om tidligere rapportert array-CGH data [32] hadde relativt lav oppløsning for CNV deteksjon, brukte vi disse dataene for å søke etter en skjevhet i endringer av genkopitallet i hvert histopatologiske type. En kopi antall gevinst på gener som koder kalsiumantagonister proteiner (CACNG6
, CACNG7
og CACNG8
, P
= 4.24 × 10 -2) var betydelig mer vanlig i DGC prøver (se Tilleggs fil 1: Tabell S17). All integrert endring informasjonen vises i Andre filer (se tilleggsfiler 1: Tabell S18 og tabell S19, se tilleggsfiler 5). Figur 3 duplisering regionen av MDM2-genet på kromosom 12 i prøver D-01 T og D-02 T. (a) Mapping dybde plott av de to kromosomer. (B) Tynne svarte toppene ble avlest ved kartlegging dybde på 2000-basen bredde. Y
-aksen viser relativ dybde. Hver enhet representerer omtrent 30 ganger sekvense dybde. (c) Gene stillinger og navn rundt forsterkede regioner. De svarte feltene viser genet steder. (D) MDM2
transkripsjonsnivåer i tumor og normalt vev tilstøtende sammenkoblede prøver og normale cellelinjer. Kvantitativ RT-PCR ble anvendt for å måle MDM2
mRNA-nivåer i prøver D-01 og D-02 (inneholdende amplifiserte MDM2
regioner), D-04, D-05, D-10, I-03, og I-04 (uten forsterkede MDM2
regioner), og tre normale cellelinjer (HDF, HMEC, og Hs 738.St/Int~~number=plural). Feilfelt ble beregnet ut fra to adskilte eksperimenter av tredoble reaksjoner.
3D strukturell analyse av mutert CDH1
For å forstå hvordan de oppdaget mutasjoner påvirke proteinstruktur /funksjon og aktivering av nedstrøms biologiske mekanismer som påvirker kreftutvikling, analyserte vi tredimensjonale ( 3D) strukturer av mutant E-cadherin protein som finnes i en IGC og fem DGC prøver (se Ytterligere fil 2). Den CDH1
genet koder for en kalsiumavhengig celle adhesjon glykoprotein og har fem ekstracellulære cadherin domener (EC1-EC5) (figur 4a). Det er kjent at samspillet mellom cadherin og kalsium er nødvendig for dimerisering, strukturell stivhet og beskyttelse mot proteolytisk degradering [58]. Mutasjoner i EC1-2 og EC2-3 veikryss er kjent for å forårsake feilaktig cadherin lokalisering og redusert celle adhesjon [59]. Strukturell analyse ble utført på fire nsSNVs (D221G, V252G, N256S, og D257N), med unntak av en tull SNV (Q23 *), en rammeskifte innsetting (S829fs), og et spleisesete (chr16: 68842472) mutasjon. Alle fire nsSNVs ble plassert i krysset mellom EC1 og EC2 (EC1-2 krysset) (figur 4b, c), og tre nsSNVs (D221G, N256S, og D257N) var i protein region som direkte samhandler med en kalsiumioner (figur 4d, e). Denne situasjonen kan føre til avvikende interaksjoner mellom cadherin domener. Det er rapportert at A298t, D231K og D231A mutasjoner, som har en lignende strukturell stilling ved EC1-2 knutepunkt til de somatiske mutasjoner som finnes i denne studien, viste et tap av celleadhesjon funksjon [60, 61]. En annen nsSNV mutasjon, V252G, ligger i β-sheet struktur av cadherin, og dens sidekjede er orientert mot det indre. Fordi β-barrel strukturer generelt inneholde alternerende polare og hydrofobe aminosyrer, med den hydrofobe rester orientert mot det indre av sylinderen for å danne en hydrofob kjerne, og de polare rester orientert mot utsiden av sylinderen på oppløsningsmiddel-eksponerte overflaten, dannelsen av den hydrofobe kjerne kan bli hindret av den V252G mutasjon (figur 4e). En tidligere exome studie rapportert to CDH1
mutasjoner, P127fs (rammeskifte mutasjon i et DGC) og V694I (i en MSI IGC) [22]. Dimerisering av to cadherin molekyler i enten en cis
eller trans
konfigurasjon skjedde i krysset mellom EC1-2 og EC1-2 [62], mens mutasjoner på EC3-4 og EC4-5 veikryss ikke signifikant innvirkning celleadhesjon [59]. Alle forfattere lese og godkjent den endelige manuskriptet.
 2015:The Year of Taking Action
Det er nyttår! Og jeg spyr hver gang jeg åpner innboksen min. Hvorfor? På grunn av alle e-postene jeg får om hvor enkelt det er å angivelig få en ny meg. Etter 7 år med arbeid for å få bedre helse, ri
2015:The Year of Taking Action
Det er nyttår! Og jeg spyr hver gang jeg åpner innboksen min. Hvorfor? På grunn av alle e-postene jeg får om hvor enkelt det er å angivelig få en ny meg. Etter 7 år med arbeid for å få bedre helse, ri
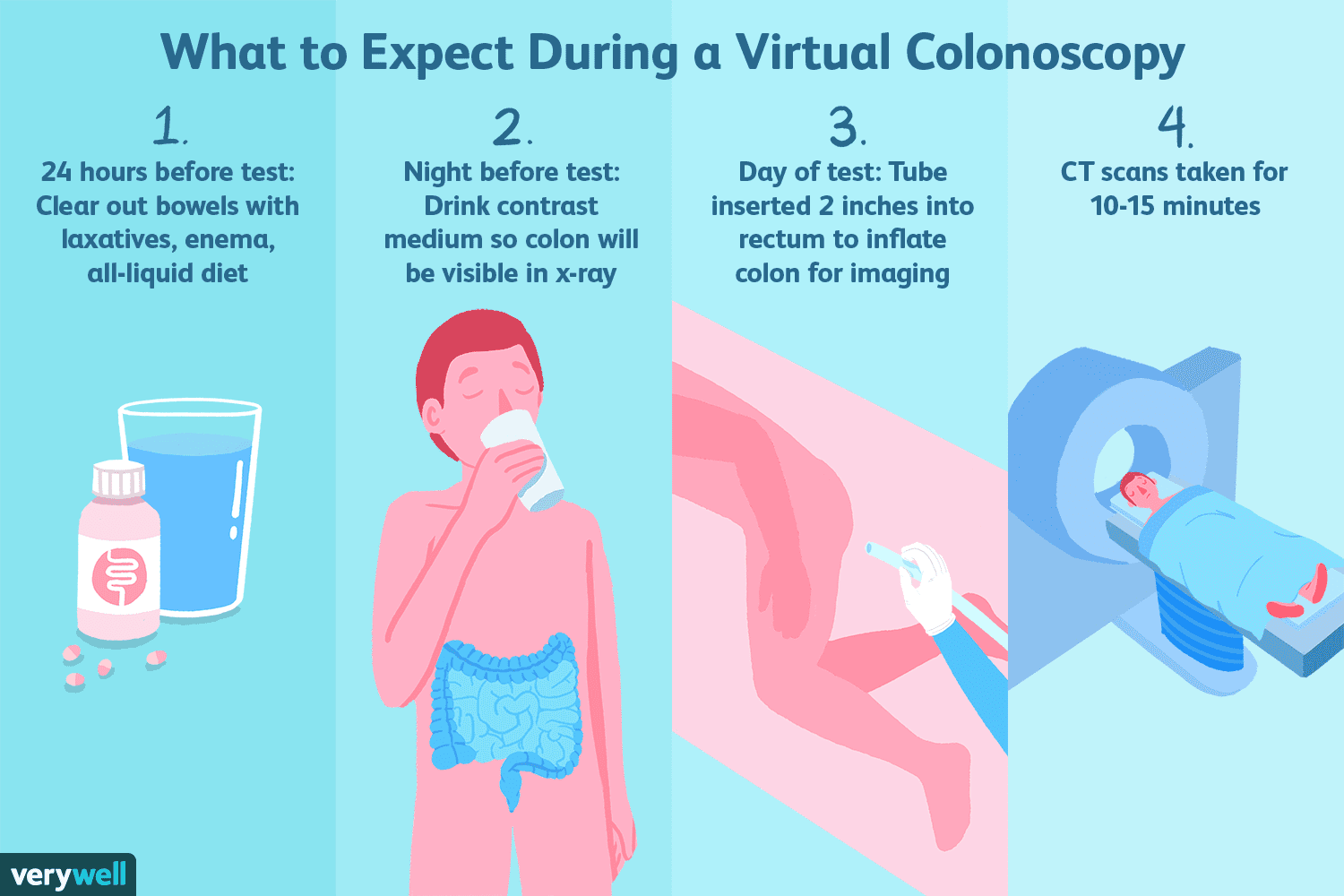 Hva er en virtuell koloskopi?
En virtuell koloskopi, også kalt CT-kolonografi, er en spesiell type datatomografi som tilbys som et alternativ til en konvensjonell koloskopi. Som en screeningtest for tykktarmskreft for personer med
Hva er en virtuell koloskopi?
En virtuell koloskopi, også kalt CT-kolonografi, er en spesiell type datatomografi som tilbys som et alternativ til en konvensjonell koloskopi. Som en screeningtest for tykktarmskreft for personer med
 Hva gjør du hvis magen din gjør veldig vondt?
Hvis magesmerter skyldes gass, oppblåsthet eller sure oppstøt, kan du bruke hjemmemedisiner og reseptfrie produkter. medisiner kan hjelpe Behandling av magesmerter avhenger av årsak, alvorlighetsgrad
Hva gjør du hvis magen din gjør veldig vondt?
Hvis magesmerter skyldes gass, oppblåsthet eller sure oppstøt, kan du bruke hjemmemedisiner og reseptfrie produkter. medisiner kan hjelpe Behandling av magesmerter avhenger av årsak, alvorlighetsgrad