För studien, publicerad 26 maj i Cell , internationella utredare samlade nästan 5, 000 prover under en treårsperiod i 60 städer i 32 länder och sex kontinenter. Utredarna analyserade proverna med en genomisk sekvenseringsteknik som kallas hagelgevärssekvensering för att upptäcka förekomsten av olika mikrober, inklusive bakterier, archaea (encelliga organismer som skiljer sig från bakterier), och virus som använder DNA som sitt genetiska material. (Andra typer av virus som använder RNA som sitt genetiska material, såsom SARS-CoV-2, viruset som orsakar COVID-19, skulle inte ha upptäckts med DNA-analysmetoderna som används i denna pre-pandemiska studie.)
Detta forskningsområde har viktiga konsekvenser för att upptäcka utbrott av både kända och okända infektioner och för att studera förekomsten av antibiotikaresistenta mikrober i olika stadsmiljöer.
"Varje gång du sätter dig i tunnelbanan, du pendlar troligen med en helt ny art, säger seniorförfattaren Dr Christopher Mason, meddirektör för WorldQuant Initiative for Quantitative Prediction och professor i fysiologi och biofysik vid Weill Cornell Medicine. Dr Mason är också medgrundare och betald konsult för Biotia och Onegevity Health, och en betald högtalare för WorldQuant LLC.
Den aktuella studien ledde till upptäckten av 10, 928 virus och 748 bakterier som inte finns i några referensdatabaser.
Dr Mason grundade MetaSUB (kort för Metagenomics and Metadesign of Subways and Urban Biomes) 2015, tillsammans med Dr Evan Afshin, som då var en grundstuderande vid Macaulay Honours College vid Queens College och nu är klinisk stipendiat i fysiologi och biofysik vid Weill Cornell Medicine och en betald konsult för Onegevity Health.
Den nyligen släppta studien leddes av Dr. Murare, David Danko, en doktorand från Weill Cornell Graduate School i Dr. Mason's lab under studien, och Daniela Bezdan, som var forskningsassistent i beräkningsbiomedicin vid Weill Cornell Medicine vid den tiden.
Genom att samla in prover av mikrober och analysera deras gener-gemensamt kända som mikrobiomet-hoppas forskarna att lära sig mer om bakterierna, virus och andra mikroorganismer som lever bland människor. Till exempel, forskningen kan hjälpa till att identifiera uppkomsten av antibiotikaresistenta stammar.
Att förutsäga antibiotikaresistens från enbart genetiska sekvenser är utmanande, men forskarna kunde kartlägga några gener som är kända för att vara kopplade till resistens, kvantifiera deras överflöd och bekräfta de genetiska markörernas förmåga att ge resistens. De fann att vissa städer hade fler resistensgener än andra, och att det kan finnas stadsspecifika signaturer för några av dessa gener.
Antimikrobiell resistens är fortfarande en stor global hälsoutmaning. "Medan ytterligare forskning behövs, denna dataset visar värdet och potentialen för kartläggning och övervakning av mikrobiomer, och de insikter det kan ge läkare, forskare och folkhälsoansvariga, "Dr Afshin sa.
Dessutom, Att lära sig om de små molekylerna och proteinerna från mikrober kan också leda till upptäckten av nya antibiotika liksom andra molekyler som har potential att utvecklas som läkemedel. Många antibiotika och läkemedel som för närvarande används har härletts från mikrobiella källor.
Upptäckter gjorda om nya mikrobiella arter kan också leda till nya laboratorieverktyg och metoder, till exempel nya sätt att använda det molekylära redigeringsverktyget som kallas CRISPR. I den här studien, forskarna hittade 838, 532 nya CRISPR-arrays-utdrag av viralt DNA som finns i bakterier-och 4,3 miljoner nya peptider (små proteiner).
På grund av dessa provtagningsinsatser Dr Mason sa att han kan förutsäga med cirka 90 procents noggrannhet var en person bor, bara genom att sekvensera DNA på deras skor. Många faktorer visade sig påverka stadens mikrobiom, inklusive den totala befolkningen och befolkningstätheten, elevation, närhet till havet och klimatet. Fynden om dessa distinkta signaturer kan möjliggöra framtida rättsmedicinska studier.
Ett mikrobiom innehåller molekylära ekon från platsen där det samlades. Ett kustprov kan innehålla saltälskande mikrober medan ett prov från en tätbefolkad stad kan visa slående biologisk mångfald, "Dr Danko sa.
Dr David Danko, Doktorand, Weill Cornell Graduate School.
Drs. Mason och Afshin började samla och analysera mikrobiella prover i tunnelbanesystemet New York City 2013. Efter att de publicerade sina första fynd, kallad PathoMap, de kontaktades av forskare från hela världen som ville göra liknande studier för sina egna städer.
Det internationella intresset inspirerade Dr. Mason's lab att skapa MetaSUB och rekryterade Daniela Bezdan som forskningsdirektör. "Vi behövde internationellt accepterade protokoll, logistik- och samarbetsavtal med forskare, försäljare, regeringskontor och filantropiska stiftelser för potentiellt 100 städer i 20 länder, "Sa Bezdan.
Idag fortsätter MetaSUB att växa och har expanderat till att samla RNA- och DNA -prover från luft, vatten och avlopp, förutom hårda ytor. Detta har lett till ett bidrag på 5 miljoner dollar för sekvensering av avloppsvatten och virusspårning i tre stater (Florida, New York och Wisconsin), och som är en del av Center for Disease Control and Prevention's new National Wastewater Surveillance System (NWSS).
Gruppen övervakar också projekt som Global City Sampling Day (gCSD), hålls varje år den 21 juni, och har gjort omfattande studier inklusive en omfattande mikrobiell analys av Rio de Janeiro tidigare, under och efter sommar -OS 2016. Många av de prover som analyserades i den aktuella studien samlades in på Global City Sampling Day 2016 och 2017.
Provtagningsinsatsen i New York genomfördes med stöd från Weill Cornell Medicine Clinical and Translational Science Center (CTSC), i samarbete med senior CTSC programchef Jeff Zhu. Dr Mason och hans kollegor förbereder för närvarande årets evenemang.
"När vi började 2015, konsortiet bestod av 16 städer; sex år senare har vi mer än 100 städer. Det är fantastiskt att ha den här gruppen nyfikna, självstartande och entusiastiska medforskare, "sa Dr Mason, som också är professor i beräkningsgenomik i beräkningsbiomedicin i HRH Prince Alwaleed Bin Talal Bin Abdulaziz Al-Saud Institute for Computational Biomedicine vid Weill Cornell Medicine.
"Även om prover samlas in över hela världen, mycket av analysen görs här i New York City vid Weill Cornell Medicine, "sade Dr Mason. Analysen och sammansättningen av sekvenser utnyttjade också Bridges and Bridges-2, Extreme Science and Engineering Discovery Environment (XSEDE) superdatorer vid Pittsburgh Supercomputing Center.
MetaSUB -forskare i Schweiz (Drs. Andre Kahles och Gunnar Rätsch) använde dessa sammansättningar och rådata för att bygga en sökbar, global DNA -sekvensportal (MetaGraph) som indexerade alla kända genetiska sekvenser (inklusive MetaSUB -data). Portalen kartlägger alla kända eller nyupptäckta genetiska element till deras plats på jorden och kan hjälpa till att upptäcka nya mikrobiella interaktioner och förmodade funktioner.
DNA -isolering från prover utfördes till stor del med stöd från Zymo Research och Promega, och sekvenserade i samarbete med Dr Shawn Levy vid HudsonAlpha Institute for Biotechnology, Dr Klas Udekwu från Stockholms universitet och New York Genome Center. Framtida och pågående studier kommer att titta på RNA och DNA med långa avläsningar och rumsliga avbildningsmetoder, samt spåra metaboliterna från de globala platserna, och fortsätta att uppdatera den genetiska kartan på planetarisk skala.
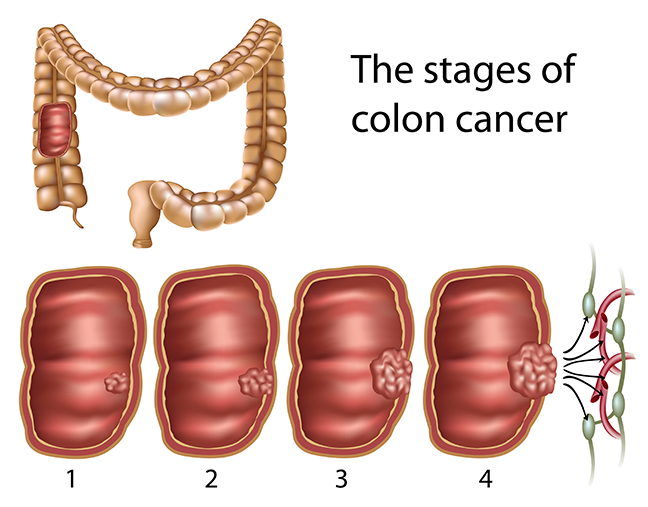 Cologuard istället för koloskopi?
Cologuard® är screeningtestet för tjocktarmscancer kan låta bra för en patient som har skjutit upp en koloskopi på grund av en antal skäl. Många är oroliga över kostnaden, komplexiteten, hörde att för
Cologuard istället för koloskopi?
Cologuard® är screeningtestet för tjocktarmscancer kan låta bra för en patient som har skjutit upp en koloskopi på grund av en antal skäl. Många är oroliga över kostnaden, komplexiteten, hörde att för
 Vita blodkroppar och deras roll i hjärnan
I en banbrytande studie, en grupp internationella forskare har funnit att hjärnan har specifika interna immunceller som hjälper till med normal hjärnutveckling och spelar en roll vid vissa neurologisk
Vita blodkroppar och deras roll i hjärnan
I en banbrytande studie, en grupp internationella forskare har funnit att hjärnan har specifika interna immunceller som hjälper till med normal hjärnutveckling och spelar en roll vid vissa neurologisk
 Vad är en Ileoanal Anastomosis (J-Pouch)-kirurgi?
Vad är en Ileoanal anastomosoperation (J-påse)? En ileal påse-anal anastomos (J-påse) är ett kirurgiskt ingrepp för att återställa kontinuiteten i magen och tarmen (gastrointestinal) efter kirurgisk
Vad är en Ileoanal Anastomosis (J-Pouch)-kirurgi?
Vad är en Ileoanal anastomosoperation (J-påse)? En ileal påse-anal anastomos (J-påse) är ett kirurgiskt ingrepp för att återställa kontinuiteten i magen och tarmen (gastrointestinal) efter kirurgisk