 Fækal transplantation fra visse donorer bedre end andre
Fækal transplantation fra visse donorer bedre end andre
 Tarmmikrobiom forbundet med adfærdsproblemer hos børn
Tarmmikrobiom forbundet med adfærdsproblemer hos børn
 Tarmmikrobiom ændrer sig ved tilberedning af vegetabilske fødevarer,
Tarmmikrobiom ændrer sig ved tilberedning af vegetabilske fødevarer,
 Transplantation af vaginal væske kan hjælpe med at behandle tilbagevendende bakteriel vaginose
Transplantation af vaginal væske kan hjælpe med at behandle tilbagevendende bakteriel vaginose
 COVID-19-restriktioner har ført til 86 procent fald i norovirusinfektioner i USA,
COVID-19-restriktioner har ført til 86 procent fald i norovirusinfektioner i USA,
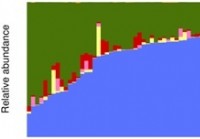 Undersøgelse beskriver den oprindelige baseline sunde tarmmikrobiomdatabase og overflodsprofil
Undersøgelse beskriver den oprindelige baseline sunde tarmmikrobiomdatabase og overflodsprofil
 Forskere bruger fagterapi til succesfuld behandling af alkoholisk leversygdom
For første gang, forskere har med succes brugt bakteriofag (fag) terapi til at eliminere alkoholrelateret leversygdom i en dyremodel. Billedkredit:Christoph Burgstedt / Shutterstock.com
Forskere bruger fagterapi til succesfuld behandling af alkoholisk leversygdom
For første gang, forskere har med succes brugt bakteriofag (fag) terapi til at eliminere alkoholrelateret leversygdom i en dyremodel. Billedkredit:Christoph Burgstedt / Shutterstock.com
 Xylitol og grapefrugtfrøekstrakt viser løfte om forebyggelse af SARS-CoV-2 infektion,
undersøgelse finder Coronavirussygdommen (COVID-19), forårsaget af det alvorlige akutte respiratoriske syndrom coronavirus 2 (SARS-CoV-2), har skabt kaos på verdensplan. Det har spredt sig siden dets
Xylitol og grapefrugtfrøekstrakt viser løfte om forebyggelse af SARS-CoV-2 infektion,
undersøgelse finder Coronavirussygdommen (COVID-19), forårsaget af det alvorlige akutte respiratoriske syndrom coronavirus 2 (SARS-CoV-2), har skabt kaos på verdensplan. Det har spredt sig siden dets
 Kvinder født med kejsersnit har større risiko for fedme og diabetes
En ny undersøgelse offentliggjort i open-access journal JAMA Network Open i april 2020 tyder på, at kvinder født ved kejsersnit har større risiko for at udvikle fedme samt type 2 diabetes mellitus i
Kvinder født med kejsersnit har større risiko for fedme og diabetes
En ny undersøgelse offentliggjort i open-access journal JAMA Network Open i april 2020 tyder på, at kvinder født ved kejsersnit har større risiko for at udvikle fedme samt type 2 diabetes mellitus i