Usando etiquetas moleculares especiales, el equipo pudo reducir la tasa de error del cinco al 15 por ciento del dispositivo MinION de Oxford Nanopore Technologies a menos del 0.005 por ciento, incluso al secuenciar muchos tramos largos de ADN a la vez.
"El MinION ha revolucionado el campo de la genómica al liberar la secuenciación del ADN de los confines de los grandes laboratorios, "dice Ryan Ziels, profesor asistente de ingeniería civil en la Universidad de Columbia Británica y coautor principal del estudio, que se publicó esta semana en Métodos de la naturaleza . "Pero hasta ahora, los investigadores no han podido confiar en el dispositivo en muchos entornos debido a su tasa de error bastante alta ".
Las secuencias del genoma pueden revelar mucho sobre un organismo, incluyendo su identidad, su ascendencia, y sus fortalezas y vulnerabilidades. Los científicos utilizan esta información para comprender mejor los microbios que viven en un entorno particular, así como desarrollar herramientas de diagnóstico y tratamientos.
Pero sin secuenciadores de ADN portátiles precisos, Los detalles genéticos cruciales pueden perderse cuando la investigación se lleva a cabo en el campo o en laboratorios más pequeños.
Entonces, Ziels y sus colaboradores en la Universidad de Aalborg crearon un sistema de código de barras único que puede hacer que las plataformas de secuenciación de ADN de lectura larga como MinION sean 1000 veces más precisas.
Después de etiquetar las moléculas objetivo con estos códigos de barras, los investigadores proceden como de costumbre:amplificando, o haciendo varias copias de, las moléculas marcadas utilizando la técnica de PCR estándar y secuenciando el ADN resultante.
Luego, los investigadores pueden usar los códigos de barras para identificar y agrupar fácilmente los fragmentos de ADN relevantes en los datos de secuenciación. en última instancia, producir secuencias casi perfectas a partir de fragmentos que son hasta 10 veces más largos de lo que pueden procesar las tecnologías convencionales. Los tramos más largos de ADN permiten la detección de variaciones genéticas incluso leves y el ensamblaje de genomas en alta resolución.
"Lo hermoso de este método es que es aplicable a cualquier gen de interés que pueda amplificarse, "dice Ziels, cuyo equipo ha creado el código y el protocolo para procesar los datos de secuenciación disponibles a través de repositorios de código abierto.
Esto significa que puede ser muy útil en cualquier campo donde la combinación de información genómica de alta precisión y largo alcance es valiosa. como la investigación del cáncer, investigación de plantas, genética humana y ciencia del microbioma ".
Ryan Ziels, Coautor principal del estudio y profesor asistente de ingeniería civil, Universidad de Columbia Britanica
Actualmente, Ziels colabora con Metro Vancouver para desarrollar una versión ampliada del método que permite la detección casi en tiempo real de microorganismos en el agua y las aguas residuales.
Con una imagen precisa de los microorganismos presentes en sus sistemas de agua, dice Ziels, las comunidades pueden mejorar sus estrategias de salud pública y tecnologías de tratamiento, y controlar mejor la propagación de microorganismos dañinos como el SARS-CoV-2.
 Antibióticos como tratamiento para el SII
Antibióticos como tratamiento para el SII
 Haga un recorrido:Centro para la Excelencia en Colonoscopia
Haga un recorrido:Centro para la Excelencia en Colonoscopia
 El misterio de la negligencia de $6500 millones de PPI
El misterio de la negligencia de $6500 millones de PPI
 Reducir estos tipos de alimentos puede ayudarlo a reducir en gran medida la incidencia del reflujo ácido
Reducir estos tipos de alimentos puede ayudarlo a reducir en gran medida la incidencia del reflujo ácido
 ¿Son seguras las desintoxicaciones y limpiezas?
¿Son seguras las desintoxicaciones y limpiezas?
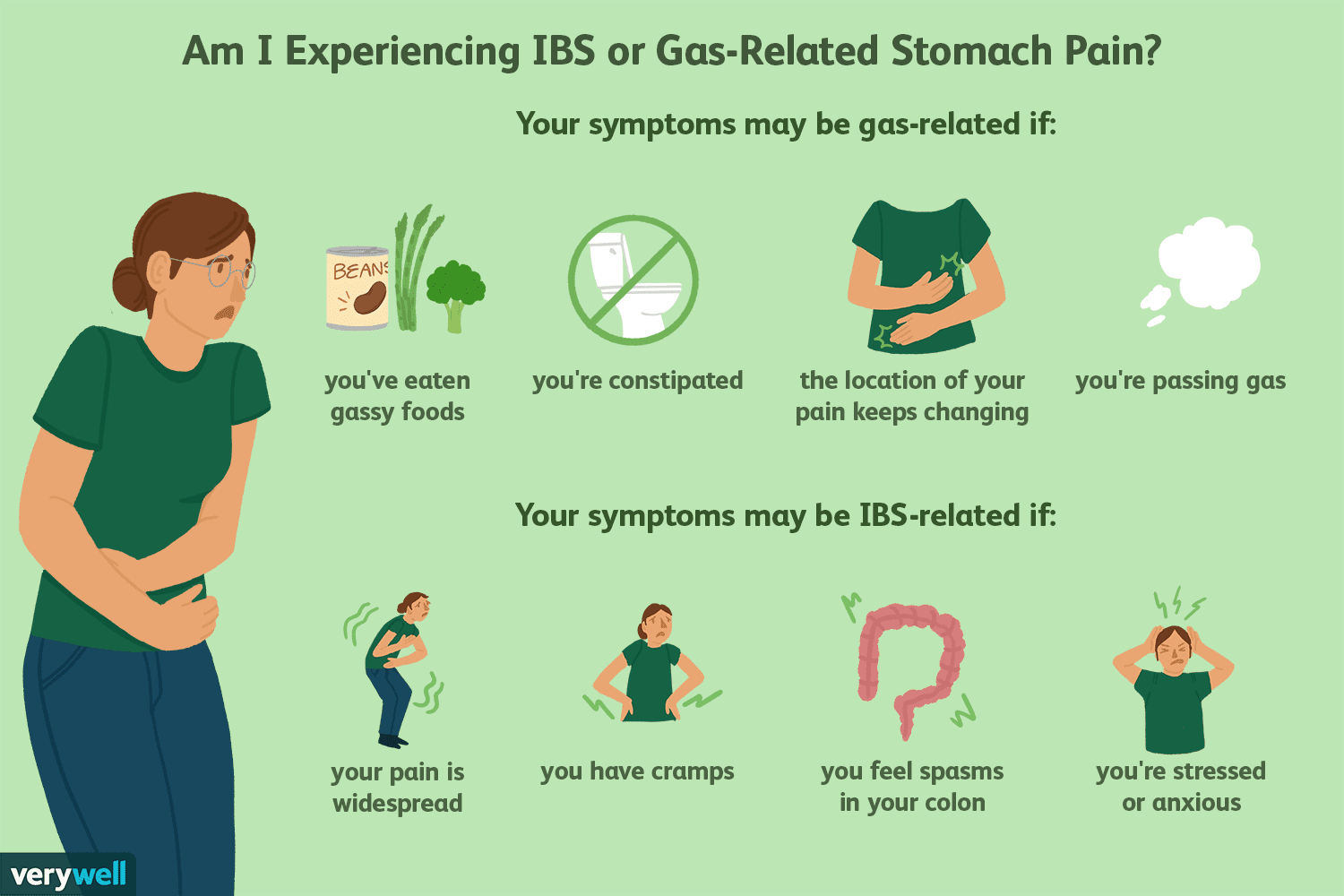 Dolor por gas atrapado y otras fuentes de dolor abdominal del SII
Dolor por gas atrapado y otras fuentes de dolor abdominal del SII
 El ejercicio de alta intensidad puede ser malo para los intestinos
Últimas noticias sobre digestión Cuando falla el trasplante de riñón, lo mejor es volver a intentarlo No permita que la acidez estomacal arruine su fiesta navideña La demanda de trasplantes de hígado
El ejercicio de alta intensidad puede ser malo para los intestinos
Últimas noticias sobre digestión Cuando falla el trasplante de riñón, lo mejor es volver a intentarlo No permita que la acidez estomacal arruine su fiesta navideña La demanda de trasplantes de hígado
 Cultivos de heces:cuándo se solicitan y por qué
Algunos problemas digestivos, como la diarrea, pueden desaparecer por sí solos, pero cuando no lo hacen, es necesario visitar al proveedor de atención médica. Un proveedor de atención médica querrá re
Cultivos de heces:cuándo se solicitan y por qué
Algunos problemas digestivos, como la diarrea, pueden desaparecer por sí solos, pero cuando no lo hacen, es necesario visitar al proveedor de atención médica. Un proveedor de atención médica querrá re
 ¿Cómo puedo recuperar la salud de mi hígado?
Tipos de enfermedad hepática Su médico puede sugerir cambios en el estilo de vida para mejorar la función hepática, como pérdida de peso, dieta saludable, reducción del colesterol, reducción del al
¿Cómo puedo recuperar la salud de mi hígado?
Tipos de enfermedad hepática Su médico puede sugerir cambios en el estilo de vida para mejorar la función hepática, como pérdida de peso, dieta saludable, reducción del colesterol, reducción del al