Utilizzando speciali tag molecolari, il team è stato in grado di ridurre il tasso di errore dal 5 al 15% del dispositivo MinION di Oxford Nanopore Technologies a meno dello 0,005%, anche durante il sequenziamento di molti lunghi tratti di DNA alla volta.
"Il MinION ha rivoluzionato il campo della genomica liberando il sequenziamento del DNA dai confini dei grandi laboratori, "dice Ryan Ziels, un assistente professore di ingegneria civile presso l'Università della British Columbia e co-autore dello studio, che è stato pubblicato questa settimana in Metodi della natura . "Ma fino ad ora, i ricercatori non sono stati in grado di fare affidamento sul dispositivo in molte impostazioni a causa del suo tasso di errore predefinito piuttosto elevato".
Le sequenze del genoma possono rivelare molto su un organismo, compresa la sua identità, i suoi antenati, e i suoi punti di forza e vulnerabilità. Gli scienziati usano queste informazioni per comprendere meglio i microbi che vivono in un particolare ambiente, nonché per sviluppare strumenti diagnostici e trattamenti.
Ma senza sequenziatori di DNA portatili accurati, dettagli genetici cruciali potrebbero essere persi quando la ricerca viene condotta sul campo o in laboratori più piccoli.
Così Ziels e i suoi collaboratori dell'Università di Aalborg hanno creato un sistema di codici a barre unico che può rendere le piattaforme di sequenziamento del DNA a lettura lunga come il MinION oltre 1000 volte più accurate.
Dopo aver etichettato le molecole bersaglio con questi codici a barre, i ricercatori procedono come farebbero di solito:amplificando, o fare più copie di, le molecole marcate utilizzando la tecnica PCR standard e sequenziando il DNA risultante.
I ricercatori possono quindi utilizzare i codici a barre per identificare e raggruppare facilmente i frammenti di DNA rilevanti nei dati di sequenziamento, producendo infine sequenze quasi perfette da frammenti che sono fino a 10 volte più lunghe di quanto le tecnologie convenzionali possano elaborare. Tratti di DNA più lunghi consentono la rilevazione di variazioni genetiche anche minime e l'assemblaggio di genomi in alta risoluzione.
"Una cosa bella di questo metodo è che è applicabile a qualsiasi gene di interesse che può essere amplificato, "dice Ziel, il cui team ha reso disponibile il codice e il protocollo per l'elaborazione dei dati di sequenziamento tramite repository open source.
Ciò significa che può essere molto utile in qualsiasi campo in cui la combinazione di informazioni genomiche ad alta precisione e a lungo raggio è preziosa, come la ricerca sul cancro, ricerca sulle piante, genetica umana e scienza del microbioma".
Ryan Ziel, Co-autore dello studio e assistente professore di Ingegneria Civile, Università della Columbia Britannica
Ziels sta attualmente collaborando con Metro Vancouver per sviluppare una versione ampliata del metodo che consente il rilevamento quasi in tempo reale di microrganismi nell'acqua e nelle acque reflue.
Con un quadro accurato dei microrganismi presenti nei loro sistemi idrici, dice Ziel, le comunità potrebbero essere in grado di migliorare le proprie strategie di salute pubblica e le tecnologie di trattamento e controllare meglio la diffusione di microrganismi dannosi come SARS-CoV-2.
 Malattia della cistifellea
Malattia della cistifellea
 Mese di sensibilizzazione IBS | La storia personale di Andrea
Mese di sensibilizzazione IBS | La storia personale di Andrea
 I problemi gastrointestinali possono essere uno svantaggio per la chirurgia dimagrante
I problemi gastrointestinali possono essere uno svantaggio per la chirurgia dimagrante
 Cisti pancreatiche
Cisti pancreatiche
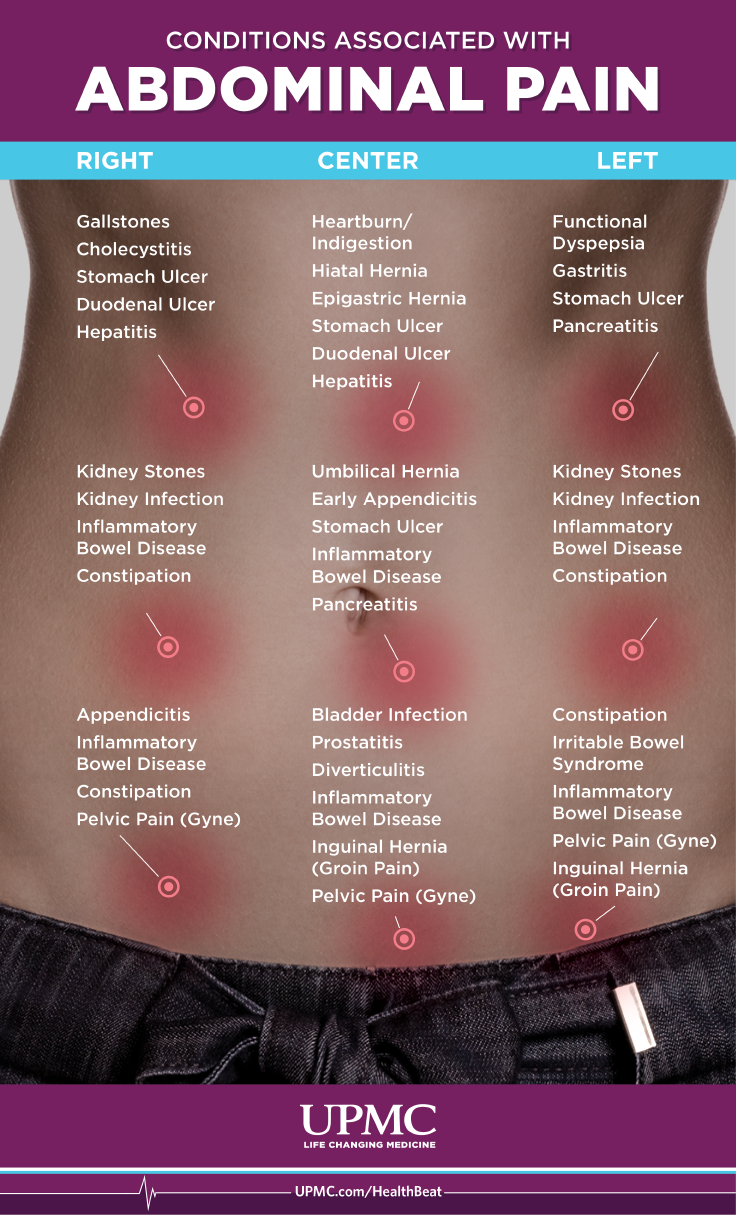 Dolore di stomaco:quando preoccuparsi del mal di stomaco
Dolore di stomaco:quando preoccuparsi del mal di stomaco
 Cosa possono dirci le feci antiche sull'evoluzione del microbioma intestinale umano?
Cosa possono dirci le feci antiche sull'evoluzione del microbioma intestinale umano?
 Cosa sapere prima di iniziare una resina acida biliare
Le resine degli acidi biliari, note anche come sequestranti degli acidi biliari, sono una classe di farmaci per abbassare il colesterolo che abbassano principalmente il colesterolo LDL. I farmaci in q
Cosa sapere prima di iniziare una resina acida biliare
Le resine degli acidi biliari, note anche come sequestranti degli acidi biliari, sono una classe di farmaci per abbassare il colesterolo che abbassano principalmente il colesterolo LDL. I farmaci in q
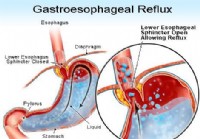 Oltre al cibo:cosa scatena il bruciore di stomaco e la GERD?
Cosa provoca il bruciore di stomaco? Nella malattia da reflusso gastroesofageo (GERD), il LES non si chiude correttamente consentendo al contenuto dello stomaco di fuoriuscire o refluire nel tubo d
Oltre al cibo:cosa scatena il bruciore di stomaco e la GERD?
Cosa provoca il bruciore di stomaco? Nella malattia da reflusso gastroesofageo (GERD), il LES non si chiude correttamente consentendo al contenuto dello stomaco di fuoriuscire o refluire nel tubo d
 La metà dei farmaci in uso danneggia i batteri intestinali,
dice nuovo studio Una presentazione sorprendente alla UEG Week 2019, a Barcellona, rivela che il microbioma intestinale è ad alto rischio di danno ogni volta che usiamo un farmaco da uno o più dei 18
La metà dei farmaci in uso danneggia i batteri intestinali,
dice nuovo studio Una presentazione sorprendente alla UEG Week 2019, a Barcellona, rivela che il microbioma intestinale è ad alto rischio di danno ogni volta che usiamo un farmaco da uno o più dei 18