Ora, un gruppo di scienziati ha ideato un nuovo metodo per un'analisi rapida e completa di ogni tipo di batterio nell'intestino, nonché un elenco di specie trovate nell'intestino umano sano per tipo e numero (GutFeelingKB), e un nuovo modello di report chiamato Fecal Biome Population Report (FecalBiome) che renderà più facile capire esattamente cosa sta succedendo nell'intestino.
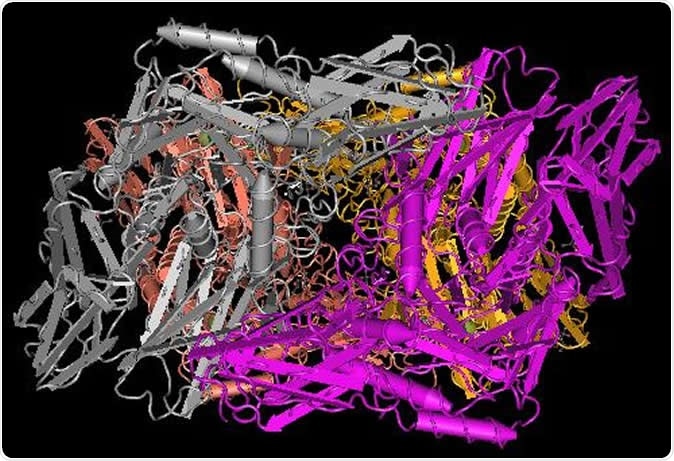 Struttura cristallina della presunta beta-galattosidasi da Bacteroides fragilis. Credito d'immagine:Istituti Nazionali di Sanità
Struttura cristallina della presunta beta-galattosidasi da Bacteroides fragilis. Credito d'immagine:Istituti Nazionali di Sanità microrganismi, o microbi, sono in giro da molto tempo, e modellare sia l'ambiente esterno che quello interno degli esseri umani. La parola “microbioma” è stata coniata da Joshua Lederberg nel 2001, come un modo per focalizzare l'attenzione degli scienziati sulle molteplici interazioni tra i microbi che abitano e nel nostro corpo, e la loro interazione con la nostra fisiologia umana. Il termine è stato ora definito come "una comunità multispecie di microrganismi in qualsiasi ambiente:ospite, habitat, o ecosistema.” Lungi dall'essere semplici invasori intenzionati alla nostra distruzione, il microbioma umano comprende in sé un intero mondo vivente, portando un set completo e altamente diversificato di geni che interagiscono e cambiano, e hanno anche un impatto sulla salute umana. Questo mix genetico microbico è chiamato metagenoma. Il progetto Human Microbiome (HMP) è decollato nel 2008 e ha contribuito a catalizzare una maggiore caratterizzazione e comprensione di come funzionano queste comunità.
La ricerca sul microbioma intestinale richiede una raccolta e un'analisi di dati accurati ad alto rendimento, nonché strutture per integrare i dati elaborati in modo organizzato per l'archiviazione, condivisione e accesso tra gruppi di ricerca. La maggior parte degli studi precedenti si concentrava su specifici geni o gruppi di organismi, tralasciando ampi segmenti dei genomi microbici. Anche, diversi standard di riferimento hanno portato a una varietà di affermazioni sulla composizione intestinale.
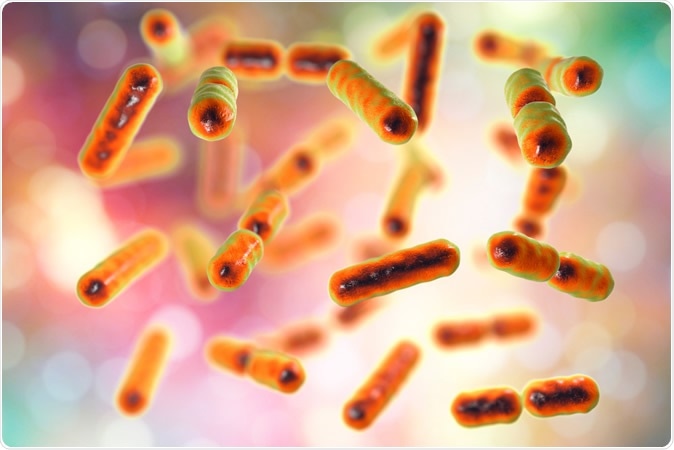 Batteri Bacteroides fragilis, uno dei principali componenti del normale microbioma dell'intestino umano, Illustrazione 3D Credito:Kateryna Kon / Shutterstock
Batteri Bacteroides fragilis, uno dei principali componenti del normale microbioma dell'intestino umano, Illustrazione 3D Credito:Kateryna Kon / Shutterstock Infatti, la maggior parte degli studi sul metagenoma utilizza solo un piccolo set di riferimento di sequenze di acidi nucleici da microbi o geni microbici già selezionati. Ciò è dovuto alla difficoltà di accoppiare i dati sperimentali con il database completo di nucleotidi disponibile con l'NCBI (NCBI-nt). Però, nuovi algoritmi possono ora utilizzare quest'ultimo per consentire un'analisi più accurata dei dati sperimentali per produrre un profilo di abbondanza microbica.
Il presente lavoro si basa su questa base per formare una base di conoscenza di Gut Feeling - GutFeelingKB - con campioni da un insieme sano di partecipanti. Questi campioni di microbiota intestinale sono stati sequenziati per ottenere un'immagine di come appare un metagenoma intestinale sano. Il numero del campione è stato compilato utilizzando altre 50 sequenze selezionate casualmente dall'HMP.
I ricercatori hanno anche raccolto sequenze contigue assemblate, o conti, che non corrispondono ad alcuna sequenza NCBI-nt ma possono essere rilevati in campioni fecali sani. I contig sono quindi materia oscura, non riconoscibile da alcuna sequenza nota di acidi nucleici, ma che può essere costruito in sequenze di 10, 000 nucleotidi o più. Questa lunghezza è stata scelta per ridurre il numero di contig di materia oscura estranea (artefatto) pur includendo la materia oscura microbica. Il GutFeelingKB è quindi una base di conoscenza completa sul microbioma intestinale umano sano.
Questo è stato quindi utilizzato come riferimento per costruire un modello di segnalazione standard in cui è possibile riportare i singoli microbiomi, per consentire il confronto diretto dei risultati tra studi e campioni.
Gli scienziati hanno anche creato un nuovo flusso di lavoro utilizzando diversi programmi per computer e un database di sequenze microbiche intestinali filtrate chiamato Filtered-nt, contenente quasi 35, 000, 000 sequenze che consentono l'interpretazione biologicamente rilevante di sequenze di campioni, offrendo allo stesso tempo la garanzia che l'intero spazio di sequenza noto è stato incluso.
Così, GutFeelingKB rappresenta una raccolta accuratamente curata di sequenze nucleotidiche con metadati di 157 organismi in 60 generi.
Il microbioma umano sano contiene quindi membri di 8 phyla, 18 famiglie, 60 generi e 109 specie, principalmente dai phyla Firmicutes (40%) e Bacteroidetes (20%). Un altro 20% proviene da Actinobacteria. Tra Firmicuti, oltre la metà sono Clostridi, seguito da vicino da Bacteroides, Bifidobatteri, Enterobatteri e Lattobacilli.
Tutti i campioni sono risultati positivi per 84 dei 109 organismi, che forse rappresenta l'elenco delle specie principali.
Però, è importante notare che in tutto il mondo, organismi specifici che non sono sul GutFeelingKB sono stati mappati, come Fusobatteri, alcune specie Actinobacter e Bacteroides. La funzione di questa piattaforma sarà quella di fungere da trampolino di lancio per confrontare i risultati dell'analisi del campione di individui sani, e fornire maggiori informazioni sulle variazioni osservate sotto l'influenza di fattori dietetici, malattie e farmaci.
Bacteroides è il genere più abbondante in molti paesi, in salute. Questi sono generalmente utili all'interno dell'intestino, ma se fuoriescono, colgono la possibilità di causare infezioni che sono spesso resistenti ai farmaci e possono portare a un tasso di mortalità del 20%. Però, nell'intestino sono protettivi contro altri agenti patogeni e aiutano a scomporre i carboidrati nella dieta.
Allo stesso modo, I bifidobatteri sono tra i primi colonizzatori dell'intestino, si trovano spesso nei probiotici, e producono l'importante acetato di acidi grassi a catena corta (SCFA) che rafforza la barriera epiteliale intestinale contro le infezioni. Un ceppo di Bifidobacterium longum è stato trovato in un individuo in un gruppo di persone particolarmente longeve in Cina.
Il numero di bifidobatteri aumenta con una maggiore assunzione di proteine, e soprattutto con proteine vegetali. Anche la fibra alimentare solubile ne favorisce la crescita. Akkermansia è legata ai grassi saturi e all'acido linoleico, ma negativamente associata agli acidi grassi polinsaturi (PUFA). Bacteroides ovatus cresce di numero con l'aumento dell'assunzione di cibo, obesità e circonferenza vita. Questi esempi ci aiutano a capire come questi numeri possono essere utilizzati per intraprendere azioni sanitarie per correggere gli squilibri del microbioma in futuro.
Il modello di segnalazione pubblicato nel presente studio ha lo scopo di sostituire i formati non standardizzati utilizzati da diversi gruppi commerciali e di ricerca, che ne ostacola l'interpretazione e il confronto. Converte i dati della ricerca in un rapporto clinico, contribuendo a renderlo immediatamente fruibile.
FecalBiome ha tre domini, Campione, Paziente e risultato, simile a un rapporto del pannello metabolico. Consente inoltre ai collaboratori di condividere rapidamente molte informazioni, quando la ricerca avviene in un'ampia gamma di località. La soglia per la segnalazione può essere impostata individualmente secondo lo scopo dello studio. Riporta l'abbondanza, l'abbondanza media e le informazioni sui microbi presenti.
Il presente rapporto consente quindi di collegare la ricerca sui batteri intestinali a informazioni comprensibili sulla salute, rendendolo rilevante per la pratica medica e per il paziente. Consente inoltre un facile confronto tra gli studi. Aiuterà a rivedere più rapidamente i prodotti sostitutivi dell'intestino, e aiutare la medicina basata sull'evidenza a progredire.
Lo studio è stato pubblicato l'11 settembre 2019, in PLOS UNO .
 Epatica e crisi alcolica
Epatica e crisi alcolica
 Il legame tra disfunzione del pavimento pelvico e sintomi dell'IBS
Il legame tra disfunzione del pavimento pelvico e sintomi dell'IBS
 Austin Gastroenterology festeggia 20 anni al servizio del Texas centrale
Austin Gastroenterology festeggia 20 anni al servizio del Texas centrale
 Endoscopia della baia:aiuto nel rilevamento, prevenzione e diagnosi
Endoscopia della baia:aiuto nel rilevamento, prevenzione e diagnosi
 Quali sono i sintomi della sindrome da dumping? Precoce vs. Tardi
Quali sono i sintomi della sindrome da dumping? Precoce vs. Tardi
 Quale medicina aiuta con l'intossicazione alimentare?
Quale medicina aiuta con l'intossicazione alimentare?
 Metanogeni:la causa principale del gonfiore di cui non hai sentito parlare (e una nuova soluzione per SIBO)
Gonfiore... quellenorme sensazione di pressione come se qualcuno ti avesse appena fatto esplodere laddome con una pompa ad aria... e ora i tuoi pantaloni si sono ridotti di due taglie. O forse è quel
Metanogeni:la causa principale del gonfiore di cui non hai sentito parlare (e una nuova soluzione per SIBO)
Gonfiore... quellenorme sensazione di pressione come se qualcuno ti avesse appena fatto esplodere laddome con una pompa ad aria... e ora i tuoi pantaloni si sono ridotti di due taglie. O forse è quel
 E. coli superbatterio che si diffonde a causa della scarsa igiene del bagno,
non attraverso il cibo Un nuovo studio pubblicato su Le malattie infettive della lancetta il 22 ottobre, 2019, dice che un superbatterio comune che ne provoca più di 5, 000 casi di intossicazione al
E. coli superbatterio che si diffonde a causa della scarsa igiene del bagno,
non attraverso il cibo Un nuovo studio pubblicato su Le malattie infettive della lancetta il 22 ottobre, 2019, dice che un superbatterio comune che ne provoca più di 5, 000 casi di intossicazione al
 I grandi fagi appena scoperti offuscano il confine tra vita e non vita
Un nuovo studio pubblicato sulla rivista Natura mostra che ci sono letteralmente centinaia di virus abbastanza grandi da consumare batteri, e con proprietà tipiche di un organismo vivente piuttosto
I grandi fagi appena scoperti offuscano il confine tra vita e non vita
Un nuovo studio pubblicato sulla rivista Natura mostra che ci sono letteralmente centinaia di virus abbastanza grandi da consumare batteri, e con proprietà tipiche di un organismo vivente piuttosto