La ricerca di un team internazionale di scienziati, compresi i brasiliani, ha trovato un legame tra il cancro del colon-retto e i cambiamenti nei modelli del microbiota intestinale che non dipendono dalle abitudini alimentari delle popolazioni studiate. La scoperta apre la strada allo sviluppo di test non invasivi in grado di predire l'insorgenza della malattia.
Lo studio è pubblicato questo lunedì, 1 aprile, sulla rivista Nature Medicine. Il suo primo autore è Andrew Maltez Thomas, che ha un dottorato di ricerca in bioinformatica presso l'Università di São Paulo (USP), Brasile. Thomas è stato sostenuto dalla São Paulo Research Foundation - FAPESP tramite una borsa di studio per uno stage di ricerca presso l'Università di Trento in Italia.
In una delle più vaste e variegate indagini mai condotte sul tema, i ricercatori hanno combinato la metagenomica, bioinformatica e apprendimento automatico (con l'uso dell'intelligenza artificiale) per correlare l'insorgenza del cancro del colon-retto con i dati sul microbiota intestinale per 969 persone in Canada, Cina, Francia, Germania, Giappone e Stati Uniti.
I risultati hanno identificato gruppi di microrganismi associati al cancro del colon-retto in tutte le popolazioni studiate, e firme nel metabolismo microbico (schemi di metaboliti prodotti da microrganismi) che possono essere utilizzati per prevedere l'insorgenza della malattia. La ricerca ha avuto altri due risultati importanti. Uno è la scoperta nell'intestino di pazienti affetti da cancro del colon-retto di una specifica specie di batterio che si trova comunemente nella bocca e nelle vie aeree. L'altro è una correlazione tra il cancro del colon-retto e la presenza del gene per un enzima microbico che degrada la colina, un nutriente essenziale del gruppo della vitamina B.
Lo studio ha trovato livelli più elevati della specie batterica Fusobacterium nucleatum nei pazienti con cancro del colon-retto rispetto agli individui sani. Questo batterio abita normalmente le regioni della bocca, e si pensava che l'acidità di altre parti del tratto gastrointestinale gli fosse fatale.
"Un numero maggiore di batteri orali tende a viaggiare verso l'intestino nei pazienti con cancro del colon-retto. Questa migrazione può causare infiammazione nell'intestino, dando origine al tumore, " Thomas ha detto. "Tuttavia, non conosciamo il motivo effettivo della migrazione, solo che c'è un legame tra la presenza di questi batteri nell'intestino e il cancro del colon-retto e che il legame merita di essere studiato ulteriormente".
L'altra scoperta una presenza significativa del gene per l'enzima microbico colina trimetilammina-liasi (cutC) in campioni fecali di pazienti con cancro del colon-retto, rafforza la possibilità di un legame cancerogeno tra il microbiota intestinale e una dieta grassa, indicato da ricerche precedenti. "Quando l'enzima scinde la colina, che è abbondante nelle diete contenenti grandi quantità di carne rossa e altri cibi grassi, rilascia acetaldeide, un noto cancerogeno, " disse Tommaso.
Nello studio, i ricercatori hanno utilizzato i dati sulla composizione e sull'abbondanza di tutti i batteri trovati in 969 campioni fecali. Sviluppare un metodo di analisi semplice che possa essere ampiamente utilizzato da cliniche e ospedali, hanno selezionato i batteri statisticamente significativi.
"I nostri risultati su 16 specie di batteri erano paragonabili a quelli delle analisi che utilizzano tutte le specie. Questo è un passo importante nello sviluppo di un semplice strumento diagnostico che elimina la necessità di sequenziare l'intero microbiota ma ha la precisione necessaria, " disse Tommaso.
L'associazione non è un nesso causale
La ricerca sui legami tra microbiota intestinale e salute umana è cresciuta negli ultimi dieci anni, ma il nuovo studio innova concependo i batteri come indicatori dello sviluppo della malattia.
"I marcatori vengono in genere ricercati direttamente in associazione con le cellule tumorali. Usiamo un concetto diverso. La nostra analisi si basa sui cambiamenti in un insieme relativamente piccolo di batteri in uno spettro di centinaia di batteri che vivono nell'intestino e possono indicare la presenza di una malattia, " disse Emmanuel Dias-Neto, ricercatore presso l'International Research Center (CIPE) dell'AC Camargo Cancer Center e coautore dell'articolo.
Il sequenziamento del DNA ottenuto dal microbiota intestinale ha permesso ai ricercatori di identificare i batteri presenti in ogni campione fecale, misurare la quantità di ciascun batterio, e per identificare varianti nei loro genomi che possono essere collegate a esiti diversi, come un aumento del rischio di cancro del colon-retto.
Va sottolineato, però, che lo studio non ha dimostrato che le alterazioni del microbiota intestinale causino il cancro del colon-retto.
"Abbiamo rilevato un'associazione, ma ciò non implica necessariamente un nesso causale. La domanda è se batteri specifici possono causare il cancro o se il cancro crea un ambiente diverso nel dotto colorettale e quindi favorisce alcuni batteri rispetto ad altri. Non abbiamo ancora una risposta, che sarebbe fondamentale per i risultati della ricerca descritta nell'articolo per aiutare a sviluppare terapie per curare il cancro del colon-retto, " ha detto João Carlos Setubal, Professore Ordinario presso il Dipartimento di Biochimica dell'Università di San Paolo, coordinatore dell'Interunit Graduate Program in Bioinformatica della stessa università, e anche co-autore dell'articolo. Setubal e Dias-Neto hanno supervisionato la ricerca di dottorato di Thomas.
Analisi computazionale
Secondo i ricercatori, questo potrebbe essere il più grande studio mai realizzato sul cancro del colon-retto basato su dati provenienti da campioni fecali e da popolazioni così diverse. Il gruppo ha analizzato i dati di cinque studi pubblici e altri due studi di ricercatori dell'Università di Trento.
Con i dati di questi sette studi sono stati in grado di identificare enzimi e batteri, e per capire come il microbiota intestinale può prevedere lo sviluppo del cancro del colon-retto. Hanno usato i dati di altri due studi con 200 campioni per convalidare i loro risultati.
"Sequenziamento del DNA dei campioni, che richiedeva la distinzione tra DNA microbiota e DNA umano, era un mezzo per identificare e quantificare le specie di microrganismi e i loro geni presenti nei campioni, " Thomas ha detto. "Abbiamo estratto il DNA dai campioni fecali e lo abbiamo sequenziato. Abbiamo quindi utilizzato metodi computazionali per analizzare i dati. Di conseguenza siamo stati in grado di identificare e quantificare l'abbondanza di specie e geni".
Poiché i dati provengono da diversi studi, i ricercatori hanno utilizzato sofisticati metodi statistici per analizzarli come un insieme.
"Abbiamo utilizzato metodi statistici meta-analitici e tecniche di apprendimento automatico per scoprire quanto fossero predittivi i risultati, " disse Tommaso.
I risultati sono stati convalidati da Nicola Segata, biologo computazionale dell'Università di Trento e supervisore all'estero del progetto, e rafforzato da un altro studio condotto presso il Laboratorio europeo di biologia molecolare (EMBL) in Germania sui legami tra microbioma intestinale e cancro. Nello stesso numero di Nature Medicine è pubblicato un articolo sullo studio EMBL.
"Durante la preparazione degli articoli, abbiamo scambiato dati e informazioni con l'altro gruppo in una partnership che si è rivelata molto importante per rafforzare i nostri risultati. Sebbene abbiamo utilizzato tecniche di apprendimento automatico e diversi metodi statistici, siamo arrivati alla stessa conclusione che il microbioma intestinale può prevedere la presenza di cancro del colon-retto in diverse popolazioni e studi, " disse Tommaso.
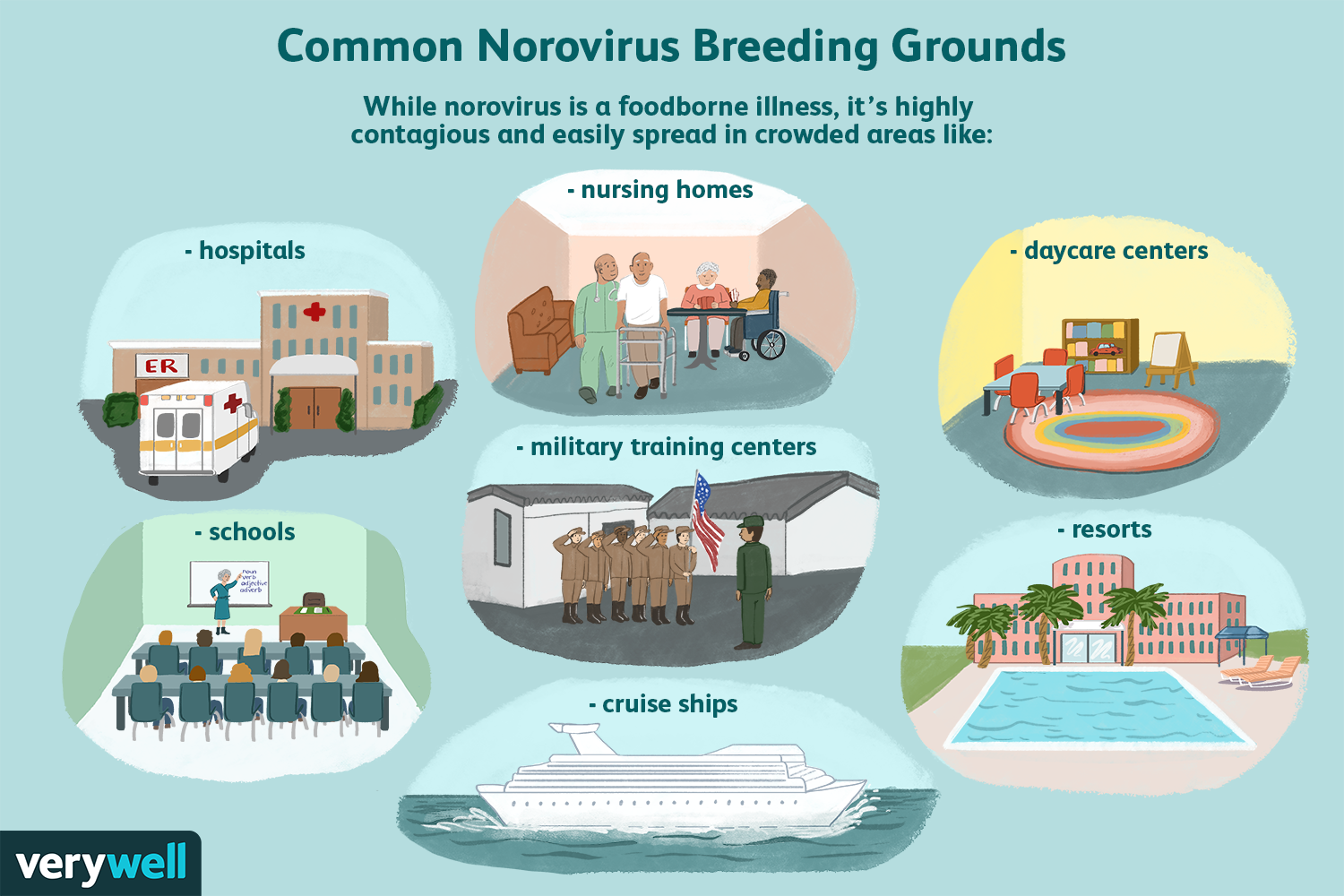 Che cos'è il norovirus?
Il norovirus è la prima causa di gastroenterite (linfluenza dello stomaco) negli Stati Uniti. È un virus altamente contagioso che provoca infiammazioni allo stomaco e allintestino. Questo porta a sint
Che cos'è il norovirus?
Il norovirus è la prima causa di gastroenterite (linfluenza dello stomaco) negli Stati Uniti. È un virus altamente contagioso che provoca infiammazioni allo stomaco e allintestino. Questo porta a sint
 L'infezione da SARS-CoV-2 prolunga la diffusione virale e la perdita di linfociti nei pazienti con cancro
I pazienti con cancro hanno un momento più difficile con le infezioni durante il trattamento, il che li pone ad alto rischio di gravi malattie e morte da COVID-19. Però, Il modo in cui la sindrome res
L'infezione da SARS-CoV-2 prolunga la diffusione virale e la perdita di linfociti nei pazienti con cancro
I pazienti con cancro hanno un momento più difficile con le infezioni durante il trattamento, il che li pone ad alto rischio di gravi malattie e morte da COVID-19. Però, Il modo in cui la sindrome res
 Quando dovresti andare dal dottore per le emorroidi?
Cosa sono le emorroidi? Se hai le emorroidi, dovresti consultare il medico se si verifica unemorragia dal retto, che potrebbe essere un segno di una condizione medica più grave, o dolore grave o ri
Quando dovresti andare dal dottore per le emorroidi?
Cosa sono le emorroidi? Se hai le emorroidi, dovresti consultare il medico se si verifica unemorragia dal retto, che potrebbe essere un segno di una condizione medica più grave, o dolore grave o ri