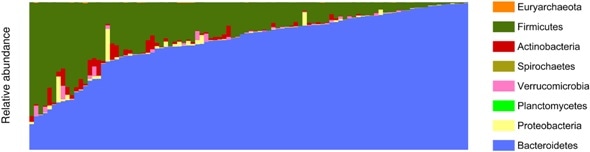
 Staplad stapeldiagram över fylogenetisk sammansättning av alla mikrobiometaxor i denna studie kollapsade på phyla -nivå i fekala prover. Gröna staplar representerar Firmicutes och de blå representerar Bacteroidetes, de två vanligaste bakteriefamiljerna. För estetiska ändamål är proverna (n =98, botten) sorterades efter deras sammansättning av Bacteroidetes och Firmicutes för att demonstrera hur baslinjens tarmmikrobiomresultat från denna studie kunde användas i samband med resultat från tidigare studier. Upphovsman:King et al, 2019
Staplad stapeldiagram över fylogenetisk sammansättning av alla mikrobiometaxor i denna studie kollapsade på phyla -nivå i fekala prover. Gröna staplar representerar Firmicutes och de blå representerar Bacteroidetes, de två vanligaste bakteriefamiljerna. För estetiska ändamål är proverna (n =98, botten) sorterades efter deras sammansättning av Bacteroidetes och Firmicutes för att demonstrera hur baslinjens tarmmikrobiomresultat från denna studie kunde användas i samband med resultat från tidigare studier. Upphovsman:King et al, 2019 Även om forskningsintresset för människans tarmmikrobioms betydelse för den allmänna hälsan fortsätter att växa, det finns för närvarande ingen omfattande tarmmikrobiomreferenslista tillgänglig för forskare och patienter. I den här studien, King och kollegor börjar katalogisera den organismala sammansättningen av friska mänskliga tarmmikrobiomer, och utvecklat en prototyprapporteringsmall för kliniker för att vidarebefordra resultat till patienter.
För att sammanställa deras databas, författarna genetiskt sekvenserade 48 avföringsprover från sexton friska deltagare som rekryterades från George Washington University campus i Washington, D.C., förutom att använda 50 fekala metagenomiska prover som laddats ner från Human Microbiome Project från individer som screenades som ”friska”.
Efter att ha analyserat alla provs genomiska och metagenomiska sekvenser med ett nytt mjukvarubaserat arbetsflöde, King och kollegor sammanställde en första databas med bekräftade mikrober och deras relativa överflöd i alla prover, GutFeelingKB, med hjälp av NCBI:s omfattande och allmänt tillgängliga genetiska information för att tillhandahålla metadata om de beskrivna organismerna.
GutFeelingKB beskriver 157 organismer (155 bakterier och två arkaeala organismer) över 60 olika släkten. Det största antalet bakterier som representerades var Firmicutes (40 procent av alla organismer på listan), som i sin tur bestod av 20 procent Clostridia, 19 procent Bakterioidia, 17 procent Bifidobacteriales, 14 procent Enterobacterales, och 14 procent Lactobacillales -bakterier - bakterieklasser som också finns i yoghurt och andra probiotiska livsmedel. Författarna noterar också att 84 organismer var gemensamma för alla prover, vilket kan indikera att dessa kan vara kärnarter för människans tarm.
Denna studie rekryterade bara sexton deltagare, ett litet urval. Men medan ytterligare studier kan fortsätta att identifiera ytterligare organismer som finns i friska tarmar från hela världen, GutFeelingKB är ett viktigt första steg. Databasen kan fungera som en utgångspunkt för jämförande analys av prover och utveckling av framtida patientmikrobiombehandlingar.
Författarna lägger till:
Vårt mål är att kartlägga det friska tarmmikrobiomet så att vi och andra forskare kan använda våra data för att utveckla sjukdomsspecifika förutsägelsemodeller. "
 Leder hypertoni alltid till allvarlig COVID-19?
Leder hypertoni alltid till allvarlig COVID-19?
 Cryptosporidios förvärrades av vanligt förekommande probiotika
Cryptosporidios förvärrades av vanligt förekommande probiotika
 Rotavirusvaccination är inte kopplad till risk för typ 1 -diabetes mellitus
Rotavirusvaccination är inte kopplad till risk för typ 1 -diabetes mellitus
 Forskning visar hur tarmmikrober påverkar maginfluensa
Forskning visar hur tarmmikrober påverkar maginfluensa
 Apoptos är en viktig förmedlare av patogenes vid djurinfektion med coronavirus
Apoptos är en viktig förmedlare av patogenes vid djurinfektion med coronavirus
 Hydrogen Breath är inte från Bikini Atoll
Hydrogen Breath är inte från Bikini Atoll
 DNA -metabarkodning kan förbättra analysen av människans kost
En ny studie har visat att DNA -metabarkodning fungerar som ett kraftfullt nytt verktyg för att övervaka människors intag av växter, som kan hjälpa forskare att förstå mer om vad vi äter och hur det p
DNA -metabarkodning kan förbättra analysen av människans kost
En ny studie har visat att DNA -metabarkodning fungerar som ett kraftfullt nytt verktyg för att övervaka människors intag av växter, som kan hjälpa forskare att förstå mer om vad vi äter och hur det p
 Det är säkert att mata råfoder till husdjur,
hittar en ny studie En stor multinationell studie visar att husdjursägare inte tror att utfodring av rå mat ökar risken för infektion hos hushållsmedlemmar. Råmat inkluderar allt okokt kött och andra
Det är säkert att mata råfoder till husdjur,
hittar en ny studie En stor multinationell studie visar att husdjursägare inte tror att utfodring av rå mat ökar risken för infektion hos hushållsmedlemmar. Råmat inkluderar allt okokt kött och andra
 Män som äter yoghurt två gånger i veckan är mindre benägna att utveckla tarmcancer
Ny forskning har avslöjat att män som äter två eller flera portioner yoghurt per vecka kan minska risken för att utveckla cancer före tillväxt som kan leda till tarmcancer. Om resultaten kan replikera
Män som äter yoghurt två gånger i veckan är mindre benägna att utveckla tarmcancer
Ny forskning har avslöjat att män som äter två eller flera portioner yoghurt per vecka kan minska risken för att utveckla cancer före tillväxt som kan leda till tarmcancer. Om resultaten kan replikera