sequenciamento profundo do carcinoma gástrico revela mutações somáticas relevantes para a medicina personalizada da arte abstracta
Fundo
Globalmente, o câncer gástrico é a segunda causa mais comum de morte relacionada ao câncer, com a maioria dos encargos de saúde suportados pelos economicamente menos países -desenvolvida.
Métodos
Aqui, nós relatamos a caracterização genética de 50 amostras de adenocarcinoma gástrico, usando matrizes Affymetrix SNP e arrays de expressão de mRNA Illumina, bem como Illumina seqüenciamento das regiões codificantes dos 384 genes pertencentes a várias vias conhecidas ser alterados em outros tipos de câncer.
resultados
alterações genéticas foram observadas no WNT, Hedgehog, ciclo celular, danos no DNA e epitelial-a-mesenquimal de transição de vias.
Conclusões
os dados sugerem alvo terapias aprovadas ou em desenvolvimento clínico do carcinoma gástrico seria de benefício para ~ 22% dos pacientes estudados. Além disso, as novas mutações detectadas aqui, são susceptíveis de influenciar a resposta clínica e sugerem novos alvos para a descoberta de medicamentos.
Fundo
Apesar do recente declínio das taxas de mortalidade por câncer gástrico na América do Norte e na maior parte do Norte e na Europa Ocidental , câncer de estômago continua sendo uma das principais causas de morte em todo o mundo e é comum no Japão, Coréia, Chile, Costa Rica, Federação Russa e outros países da antiga União Soviética [1]. Apesar da melhoria das modalidades de tratamento e rastreio, o prognóstico de pacientes com adenocarcinoma gástrico continua pobre [2]. Para compreender a patogénese e a desenvolver novas estratégias terapêuticas, é essencial para dissecar os mecanismos moleculares que regulam a progressão do cancro gástrico. Em particular, os mecanismos oncogénicas que pode ser alvo de medicina personalizada.
O termo "dependência oncogene" para descrever células de cancro altamente dependente de uma dada via oncogene ou oncogénico foi introduzido por Weinstein [3, 4]. O conceito reforça o desenvolvimento de terapias específicas que tentam inactivar um oncogene, crítica para a sobrevivência de células cancerosas ao mesmo tempo poupando as células normais que não são viciadas semelhante.
Vários oncogenes activados em alta frequência em outros cancros têm também sido mostrado para ser mutado no câncer gástrico. Segue-se que a terapêutica comercializados segmentação destes oncogenes iria tratar eficazmente uma proporção de carcinomas gástricos, quer como agentes únicos ou em combinação. Em janeiro de 2010, o trastuzumab foi aprovado em combinação com quimioterapia para o tratamento de primeira linha do cancro gástrico avançado e metastático -positivo ERBB2
. Trastuzumab é o primeiro agente direccionado para ser aprovado para o tratamento do carcinoma gástrico e um aumento de 12,8% na taxa de resposta foi observada com a adição de trastuzumab à quimioterapia no adenocarcinoma gástrico positiva ERBB2
[5, 6]. Estima-se que 2-27% dos cancros gástricos abrigar ERBB2
amplificações e pode ser tratada com inibidores de ERBB2 [7, 8]. Da mesma forma, a sobre-expressão de um outro receptor tirosina quinase (RTK) EGFR
, foi observado no câncer gástrico e vários ensaios de inibidores de EGFR
neste tipo de câncer estão em andamento (revisto em [9, 10]). Além disso, alguns tipos de câncer gástrico abrigar amplificação de DNA ou a sobre-expressão do RTK MET
[11, 12] e sua parálogo MST1R
[13] e pode ser tratado com MET
ou MST1R
inibidores [14-20 ]. Finalmente, FGFR2
sobre-expressão e amplificação foi observado em uma pequena proporção de cancros gástricos (cirrosos) [21], e inibidores têm mostrado alguma eficácia clínica em [22].
Jusante das RTKs, KRAS
amplificação de tipo selvagem e de mutação também foi encontrada em cerca de 9-15% dos cancros gástricos [23, 24] e podem ser eficazmente tratados com inibidores de MEK [25, 26]. A activação da via PI3K /Akt /mTOR também tem sido visto em 4-16% de cancro gástrico [27-30] e assim podem ser sensíveis a inibidores de PI3K [31-34]. Do mesmo modo, do ciclo celular cinase AURKA
tem sido mostrado para ser activados em cancro gástrico [35, 36] e inibidores AURKA em desenvolvimento clínico [37] pode ter um benefício clínico.
Relatórios da frequência de diferentes tipos de activação oncogénica e os seus co-ocorrência são limitadas. Em contraste com gastrointestinonal tumores do estroma (GIST), que são caracterizados por uma elevada frequência de KIT e PDGFRA
activação [38] e, portanto, eficazmente tratada na maioria por imitanib sunitinib e [39, 40], adenocarcinoma gástrico aparece ser uma doença heterogénea molecularmente sem perturbação oncogénica de alta frequência, até agora descoberto. Isto é ilustrado por um estudo recente de mutação somática de genes codificantes de cinase através de 14 linhas celulares de cancro gástrico e três tecidos de cancro gástrico que variações de um único nucleótido descobertos mais de 300 quinase e romance variantes estruturais relacionada com quinase. No entanto, a mutação não é muito frequentemente recorrente ou quinase mutante foi descoberta [41].
Com o objetivo de elucidar o potencial para o tratamento de carcinoma gástrico com terapias direcionadas tanto no mercado, no desenvolvimento ou a serem descobertos, temos caracterizado clínica amostras de carcinoma gástrico para detectar ativação de oncogenes.
Fizemos uma abordagem global através da análise das amostras em Affymetrix matrizes SNP e arrays de expressão de mRNA Illumina. Estas tecnologias são bem validado para a detecção do genótipo, cópia de DNA variação do número e perfil de expressão de mRNA. Eles são passíveis de amostras clínicas heterogéneas. As amostras também foram interrogados pela segunda geração (Illumina) sequenciamento. Relativamente novas tecnologias de sequenciamento de segunda geração oferecem maior rendimento e capacidade de sequenciamento de profundidade. Este último é especialmente importante para a caracterização de amostras cancerosas que tendem a incluir uma mistura de tipos de células incluindo as células normais que se infiltram, vasculatura e de células de tumor de diferentes genótipos. Neste estudo foram utilizados enriquecimento alvo e tecnologia de sequenciação Ilumina para sequenciar as regiões de codificação de 384 genes. Decidimos para favorecer profundidade de cobertura sobre uma cobertura mais ampla, a fim de capturar mutações presentes em subpopulações dentro dos tumores. Estudos recentes têm mostrado cancros tendem a abrigar muitas mutações em um menor número de vias de sinalização [42, 43], portanto, concentrou-se em genes nestas vias. Também incluímos genes que codificam proteínas mostrados anteriormente para afetar resposta a terapias específicas e mais propensos a ser alvo com sucesso por pequena intervenção molécula, como o nosso objectivo é encontrar formas mais eficazes e inovadoras para o tratamento de carcinoma gástrico.
Métodos
As amostras de tecido
amostras de DNA e RNA foram obtidos de hospitais na Rússia e Vietnam acordo com a IRB aprovou protocolos e com o IRB aprovado formulários de consentimento para análise molecular e genética. Os próprios centros médicos também têm comitês de ética internos com avaliação do protocolo e ICFs. As amostras foram adquiridos por meio de soluções de tecido Ltd http: //www. Tissue-soluções com /.. Para características da amostra ver adicional tabela de arquivo 1 S1
Arrays
genótipos e copiar perfis números foram gerados para cada amostras utilizando 1 � de DNA de execução em matrizes Affymetrix SNP V6 usando protocolos Affymetrix. dados de variação do número de cópia foi analisada dentro do http software ArrayStudio: //www. Omicsoft com.. Os dados foram normalizados utilizando algoritmo de Affymetrix e segmentado usando CBS. Um perfil de transcrição foi gerado para cada amostra usando 1 mg de prazo total de RNA em Illumnia HG-12 matrizes de expressão RNA seguintes protocolos Illumina. Os dados foram analisados dentro do http software Illumina GenomeStudio:.. //Www illumina com /software /genomestudio_ software ILMN.. Como um procedimento de pré-processamento de dados, um conjunto de sonda só foi mantido, se tiver um "presente" (ou seja, dois desvios padrão acima do fundo) Chamada em pelo menos uma das amostras. Os valores dos sinais dos conjuntos de sondas restantes foram transformados em escala logarítmica 2-baseado e normalização quantil foi realizada. cópia de DNA e os níveis de expressão de RNA foram integrados ao nível do gene dentro do http software ArrayStudio: //www. Omicsoft com.. análise de enriquecimento via foi realizada dentro do GeneGo suíte análise MetaCore http: //www. GeneGo com /.. Todos os dados de matriz deste estudo está disponível no GEO http: //www NCBI NLM nih gov /geo /sob o número de série GSE29999
sequenciamento de DNA profunda alvejado
5..... ug de DNA foi PCR enriquecido para os exons de codificação de qualquer transcrição conhecido de 384 genes de interesse (arquivo adicional de 2 tabela S2) usando a plataforma http Raindance:... //www gias raindancetechnol com /
As bibliotecas alvo resultantes foram sequenciados utilizando Illumnia GAII em um comprimento ler de 54 nt. Sequência lê foram mapeados para o genoma de referência (hg18) utilizando o programa BWA [44]. Bases fora das regiões-alvo foram ignoradas quando resumindo estatísticas de cobertura e chamadas variantes. SAMtools foi usado para analisar os alinhamentos e fazer chamadas genótipo [45], e qualquer chamada que se desvia da base de referência foi considerado como uma variante potencial. O pacote SAMtools gera estimativas de qualidade de consenso e de qualidade variante para caracterizar as chamadas de genótipos. Precisão de chamadas genótipo foi estimado pela concordância ao genótipo chamadas da Affymetrix 6,0 SNP microarrays. matrizes de concordância de amostras com base em dados de SNP e seqüência foram gerados para verificar se há rotulagem inadequada amostra (adicional de arquivo 3 figura S1). Concordância e quantidade de chamadas de genótipos foram tabulados para limiares de qualidade do consenso, a qualidade variante, e profundidade. O conjunto final de chamadas variantes foram identificados utilizando qualidade de consenso maior do que ou igual a 50 e de qualidade superior a variante 0. Para identificar exclusivamente alterações somáticas, apenas as mutações presentes na amostra de cancro e não foi detectado em qualquer das amostras normais foram retidos. Como um filtro adicional para as variantes da linha germinativa, presentes em todas as variantes e dbSNP 1000 conjuntos de dados de polimorfismo genoma foram removidos.
Q-PCR
Q-PCR foi realizada por meio do protocolo padrão utilizando Fluidigm 48 * 48 matriz dinâmica. Em primeiro lugar, a execução de validação foi realizado usando RNA de controle agrupados de três espécimes. Quatro quantidades de ARN de entrada foram testados (125 ng, 250 ng, 375 ng e 500 ng). Os pontos de dados em triplicado foram obtidos para a diluição, subsequentemente, 10 pontos por cada condição de série por ensaio. Os melhores resultados globais foram de 250 ou 500 ng, o que originou valores de rendimento ~ 85%. Portanto quantidade de 250 ng de entrada para as amostras experimentais. Os dados foram produzidas em triplicado e média combinada. Os valores Ct foram convertidos em abundância usando a fórmula abundância padrão = 10 (40-CT /3.5). Os dados de teste foi normalizada para empregadas usando o método de análise de covariância por meio de que as duas donas (GAPDH e beta-actina) foram utilizados para calcular uma nota robusta e a pontuação foi utilizado como covariável para ajustar os outros genes. A análise dos dados foi realizado no software Arraystudio.
seqüenciamento Sanger
primers Genomic DNA PCR foram ordenados de IDT (Integrated DNA Technologies Inc, Coralville, Iowa). As reacções de PCR foram realizadas utilizando polimerase Platnium Invitrogen (Invitrogen, Carlsbad, CA). 50 ng de ADN genómico foi amplificado durante 35 ciclos a 94 ° C durante 30 segundos, 58 ° C durante 30 segundos e 68 ° C durante 45 segundos. Os produtos de PCR foram purificados utilizando Agencourt AmPure (Agencourt Bioscience Corporation, Beverly, MA). A sequenciação directa de produtos de PCR purificados com os iniciadores de sequenciação foram realizadas com o AB-BigDye Terminator v3.1 kit de ciclo de sequenciação (Applied Biosystems, Foster City, CA) e as reacções de sequenciação foram purificados utilizando Agencourt CleanSeq (Agencourt Bioscience Corporation, Beverly, MA). As reacções de sequenciação foram analisadas utilizando um Analisador Genético 3730XL (Applied Biosystems, Foster City, CA). Resultados Todos os dados dos resultados de sequências foram reunidos e analisados utilizando Codon Código alinhador (CodonCode Corporation, Dedham, MA).
Padrões de DNA e amplificação de RNA em toda amostras são consistentes com estudos anteriores
consistente com a maioria dos outros cancros humanos, Número de cópia alterações ocorreram entre os genomas das amostras de cancro gástrico em comparação com 50 amostras normais correspondentes (Figura 1). Grandes regiões de amplificação frequente foram encontrados em regiões cromossômicas 8q, 13q, 20q e 20p. oncogenes conhecidos MYC
e CCNE1
estão localizados nas 8Q e 20p amplicões, respectivamente, e que possam contribuir para um crescimento vantagem conferida pela amplificação. Estas amplificações foram observados em estudos anteriores no câncer gástrico, juntamente com a amplificação de 20p para o qual ZNF217
e TNFRSF6B
têm sido sugeridos como genes motorista candidato [46]. Figura 1 Vista de aberrações CNV em todas as amostras de carcinoma gástrico 50, para cada autosome. O eixo y corresponde à soma do número de alterações positivas ou negativas para um determinado segmento com o rácio de log2 aqueles mudança. Áreas com um número aumentado ou diminuído cópia consistente ao longo de todas as amostras analisadas ou muito grandes alterações em algumas amostras irá mostrar grandes tamanhos positivos e negativos de mudança. Cada ponto ou segmento na figura é colorido por amostra. O código de cor é arbitrária com cada uma das amostras de cancro do 50 sendo atribuída uma cor. segmentos amplificados incluem 8q cromossomo, 20q, 20p, 3T, 7P, e 1q.
concordância entre cópia de DNA ganho número e expressão do RNA entre as amostras de câncer foi avaliada e os 200 genes contidos dentro de uma região de cópia de alta DNA frequente em amostras de câncer e que tinham altos níveis de mRNA (em comparação com o tecido normal combinado) estão tabulados na adicionais arquivo 4 S3 mesa. A maioria dos genes nesta lista são de regiões cromossômicas 20Q e 8q, sugerindo que estas amplificações têm o maior efeito sobre os níveis de mRNA, em minoria são genes para 20p, 3T, 7P, e 1T. A Figura 2 mostra os perfis de ARN medidos por Q-PCR de um gene exemplar de cada região mostrando a sobre-expressão em geral cancro gástrico, particularmente em determinadas amostras. Além MYC Comprar e CCNE1
, existem vários genes nessas regiões, que possam contribuir para uma vantagem de crescimento para a célula cancerosa. As vias biológicas mais significativamente enriquecidas para genes amplificados e sobre-expressos envolvidos na regulação da tradução (p = 0,000015) e reparação de danos no ADN (p = 0,003). As amostras com amplificações nestas regiões genômicas são anotados na Figura 3. Não há tendência perceptível para amplificações nessas regiões para co-ocorrer ou ser exclusiva. De acordo com um estudo anterior [47], o PERLD1
lócus foi amplificado (dentro do ERBB2
amplicon) em amostra de 08.280 e MMP9
foi overexpressed mas não discernibly amplificado. Também na Figura 3 amplificações de DNA focais com expressão do RNA concordantes de genes susceptíveis de afectar a resposta a terapias-alvo são indicadas, por exemplo dados subjacentes ver adicional de arquivo 5 figura S2. Figura 2 Expressão de exemplos de genes de cada região cromossómica amplificado através de amostras de estudo confirmou por Q-PCR. pontos vermelhos indicam amostras cancerosas e pontos brancos indicam amostras normais. O eixo dos y indica a abundância de ARNm.
Figura 3 perfil mutacional de amostras. As amostras de tecido são exibidas na parte superior e anotações relevantes para eles estão em colunas abaixo. Caixas vermelhas indicam a amplificação do ADN e a sobre-expressão de ARNm concordante, caixas de laranja denotar superexpressão do RNA com nenhuma evidência de amplificação de ADN, pontos vermelhos denotam perda de ADN. As caixas azuis denotam mutação somática nonsynonymous validado pelo sequenciamento Sanger e caixas roxas indicam mutações somáticas nonsynonymous, observada nos dados Illumina com nenhuma tentativa para confirmar por seqüenciamento Sanger. mudanças aminoácidos são observadas nas caixas e as mudanças que levam à perda ou ganho de um códon de parada estão em texto vermelho. Dados Sequencing
mostra elevada concordância com a genotipagem
Sequencing preparação biblioteca falhou por seis das amostras originais 50 cancerosas e quatorze das amostras emparelhadas originais normais. Portanto dois pares mais pareados foram adicionados à análise, resultando em um conjunto de dados de 44 amostras de câncer, 36 com pares normais pareados (adicional tabela de arquivo 1 S1). A região alvo incluídos 3,28 MB em todo 6.547 exons únicos em 384 genes (adicional arquivo de 2 tabela S2). cobertura mediana de em todas as amostras foi de 88,3% e caiu para 74% ao exigir cobertura mínima de 20. Toda a sequenciação foi realizada para um mínimo de 110x cobertura média de leitura em todas as regiões genômicas enriquecidas para cada amostra. A lê foram alinhados contra o genoma humano e variantes do genoma de referência foram chamados. Como um controlo, uma análise para comparar a genotipagem de chamadas a partir das matrizes Affymetrix V6 SNP e a sequenciação foi realizada Ilumina. As regiões-alvo para a sequenciação continha 1.005 loci cobertas pelas matrizes Affymetrix V6 SNP. Sem filtragem da variante sequenciamento exige métricas de qualidade, o acordo mediano entre os resultados de genotipagem e sequenciamento foi de 97,8%, com um intervalo de 65-99% (6a arquivo adicional, Figura S3a). A concordância chamada genótipo global bruto foi de 96,8%. métricas de qualidade foram escolhidos para maximizar o acordo entre a genotipagem e as chamadas de sequenciamento, minimizando falsos negativos. A métrica mais informativo foi a qualidade de consenso e um corte de ≥50 resultou na perda de cerca de 10% dos genótipos compartilhados mas um aumento global de 2% em concordância para 98,7% (6b arquivo adicional, Figura S3B). chamadas de genótipos variantes foram isolados para análise de concordância. Neste conjunto, um limiar de >qualidade variante; 0 aumento da precisão das chamadas genótipo variante a 98,9% (6c arquivo adicional, Figura S3C). Quando ambos os limiares de qualidade foram aplicados a concordância amostra média é de 99,5% (6d arquivo adicional, Figura S3D), que está dentro da região de erro matriz genotipagem. Seis amostras (08362T1, 08373T2, 336MHAXA, 08337T1, 89362T2, DV41BNOH) tiveram uma concordância de < 98% e dois deles (08393T2 e DV41BNOH) tiveram uma concordância de 82% e 88%, respectivamente. Portanto, com uma qualidade de consenso ≥ 50 e uma qualidade variante > 0, a taxa positiva falsa foi de 0,5% e 1,6% para os genótipos de referência e genótipos variantes, respectivamente (Figura 6e arquivo adicional S3E).
De todas as alterações de um só nucleótido que atinge os limiares acima, todas as variantes presente em nenhuma das amostras normais ou nas bases de dados de polimorfismo de dbSNP (v130) ou 1000 genomas eram consideradas variantes da linha germinativa e descartados. Variantes presentes apenas nos exões de amostras de cancro foram assumidos como sendo somática e retida. 18,549 variantes somáticas foram detectados no total em todas as 44 amostras (arquivo adicional 7 Tabela S4), 3357 foram previstos para ser exônico e nonsynonymous. Para dar prioridade para mutações com impacto funcional nos concentramos todas as futuras análises sobre mutações nonsynonymous e mutações destacadas que levam à perda ou ganho de códons de parada. Nós aplicamos o algoritmo SIFT [48] para prever as mudanças de aminoácidos que não são toleradas na evolução e por isso são mais susceptíveis de afectar a função da proteína, de 1509 mutações somáticas nonsynonymous ter uma pontuação SIFT de < 0,05. A taxa de mutações com pontuação SIFT < 0,05 por gene, corrigidos para comprimento CDS foi calculada (4). A Figura 4 mostra, os genes com maior concentração de mutações marcando baixo SIFT foram S1PR2
, LPAR2
, SSTR1
, TP53
, GPR78 Comprar e RET
, com S1PR2 sendo mais extremo. Há quinze mutações com pontuação SIFT < 0,05 em todo o 353aa CDS de S1PR2
, concentrados em nove amostras. S1PR2
também conhecido como
EDG5 codifica para um receptor acoplado a proteína G de S1P e activa RhoGEF, LARG
[49]. Pouco se sabe sobre o seu papel no câncer e somáticas mutações não foram observados nos 44 tecidos sequenciados para S1PR2
no banco de dados COSMIC [50]. Figura 4 Bar carta de taxa de mutações deletérias em toda gene sequenciado. Os genes sequenciados são mostrados no eixo-x. O número de mutações somáticas nonsynonymous deletérios observados em cada gene /número de aminoácidos em cada CDS em traçados. Dados Sequencing
é confirmada por seqüenciamento Sanger
Algumas mutações somáticas nonsynonymous foram selecionados para ser confirmado por seqüenciamento Sanger. Todas as mutações relatadas em azul na Figura 3 foram confirmados por sequenciamento Sanger e também foram confirmados para ser somática por sequenciação da sequência de tipo selvagem no tecido normal combinado (ver arquivo adicional 8 Figura S4 de vestígios exemplo de sequenciamento). Embora 74% foram confirmados, algumas mutações detectadas no seqüenciamento Illumnia não foram confirmados como mutações somáticas por seqüenciamento Sanger. Dezasseis dos 68 (24%) mutações que tentativas para confirmar se presentes na amostra normal e cancro, estes são mutações da linha germinativa mas não detectada em nenhuma das amostras normais por sequenciação Ilumina e também não representados na dbSNP ou dados 1000 genomas. Cinco dos dezesseis mutações germinativas eram de amostras de câncer sem tecido normal combinado incluído no conjunto de dados, os outros onze vieram de amostras de câncer com a sequência de tecido normal combinado incluído no conjunto de dados. Isto evidencia uma taxa de contaminação da linha germinativa não eliminados pelos controles normais pareados ou a comparação de bases de dados de polimorfismo conhecidos. Pode ser que a cobertura das substituições no tecido normal passa a ser menor do que na amostra de cancro e assim algumas mutações da linha germinativa permanece, apesar dos filtros somáticas. Dois dos 68 (3%) mutações que tentativas para confirmar não estavam presentes na amostra normal ou cancro por Sanger de sequenciação. Uma das causas pode ser falsos positivos nos dados Illumnia devido a artefacto; no entanto arquivo adicional 6 Figura S3 mostra a taxa de falsos positivos a ser baixa, pelo menos para aquelas variantes representadas nas matrizes Affymetrix V6. Outra possibilidade é que estes estão presentes em um subconjunto da amostra abaixo da sensibilidade do método de Sanger, mas detectado pelo sequenciamento Ilumina. Portanto, mutações do seqüenciamento Illumina também são relatados em roxo na Figura 3, alguma cautela é justificada ao interpretar estes resultados como eles podem ser polimorfismos germinativas ou presente apenas em um subconjunto da amostra de tumor.
Alterações no RAS /RAF /MEK /ERK
Três amostras de tumor tinha KRAS
alterações genéticas (Figura 3), sugerindo oportunidade terapêutica para o tratamento com inibidores de MEK. Uma destas alterações é uma mutação G12D. KRAS
G12D mutações têm sido mostrados para iniciar a carcinogénese e sobrevivência do tumor [51]. Amplificação e superexpressão de tipo selvagem KRAS
foi visto nas outras 2 amostras. KRAS
amplificação foi observada antes em 5% dos cancros gástricos primários. linhas celulares de cancro gástrico com KRAS tipo selvagem
apresentam amplificação KRAS constitutivos
activação e sensibilidade para KRAS
RNAi knockdown [24]. Uma nova mutação no KRAS
também foi observada; . (Em amostra 08393) a conseqüência funcional é desconhecida
O PIK3CA
mutação co-ocorrendo com KRAS
G12D, é conhecido por afetar a sensibilidade aos inibidores MEK [25]; Além disso, novas mutações observadas neste estudo também pode ter consequências para a mesma classe de terapêutica. Por exemplo: KSR2
funciona como um andaime molecular para promover sinalização ERK [52, 53]. Portanto, mutações em KSR2
tal como pode ser visto em sete amostras podem afectar a sensibilidade a inibidores de MEK. Um segundo exemplo é ULK1
, que controla positivamente autofagia a jusante de mTOR [54] e está mutado em catorze amostras. Autophagy é aumentada juntamente com a fosforilação de ERK, quando células cancerosas gástricas são tratadas com um inibidor de proteassoma [55], por conseguinte, as mutações no ULK1
pode afectar a sensibilidade aos tratamentos inibidores de proteossoma, tal como bortezomib como um agente único ou em combinação com inibidores de MEK.
Alterações na via PI3K /AKT
Houve interrupção de sequência substancial dos genes da via fosfatidilinositol-3-quinase (PI3K) no conjunto da amostra. Há uma série de inibidores de PI3K /AKT /mTOR em desenvolvimento clínico e pacientes com mutações de activação da via são candidatos para o tratamento [56]. PIK3CA
mutações de oncogenicity conhecidos foram encontrados em quatro amostras. Isto resulta numa frequência de mutação PIK3CA
ponto de acesso de 9%, ligeiramente mais elevado do que as estimativas anteriores de 6% (12/185) [27] e 4,3% (4/94) [57]. As mutações de ponto de acesso comum PIK3CA de oncogenicity conhecido (E545K e H1047R) [58] foram observados duas vezes cada um. Outra mutação em PIK3CA
K111E, que também foi observada antes nos quatro amostras em cósmica, foi observada uma vez e, potencialmente, novas mutações somáticas foram observadas em mais duas amostras. Foram observadas
Cinco AKT1 nonsynonymous
mutações. Embora AKT1
mutações são encontrados em cerca de 2% de todos os cancros, que ocorrem principalmente no aminoácido 15 e a importância funcional da mutação em outros locais é desconhecida. Outra mutação nonsynonymous em AKT2
foi observada em amostras 08407. Akt2
mutações são muito mais raros do que AKT1
mutações, embora um AKT2
mutação tem sido observado antes em carcinoma gástrico, com uma frequência de 2% [ ,,,0],59]. Finalmente mutação do PTEN
ou MTOR
podem afetar a resposta com a via inibidores. Vários PTEN
mutações são anotadas e mTOR
mutações são frequentes.
Alterações no receptor tirosina quinases
Os receptores tirosina-quinase (RTK) e droga tem como alvo EGFR
, ERBB2 Comprar e MET
foram cada um amplificados (log2 > 0,6) e sobre-expresso ao nível de ARN em uma amostra de cancro. Segue-se que os tumores podem ser sensíveis aos inibidores das RTKs amplificados. Além disso, mutações múltiplas nonsynonymous são observados nas suas regiões de codificação. mutações jusante seria esperado para influenciar a resposta. Por exemplo, no TEM
amostra amplificada uma mutação truncando no AKT3
pode afectar a sensibilidade a inibidores de MET.
FGFR2
é amplificado e sobre-expresso de RNA em duas amostras, também existem múltiplas mutações em FGFR1
-4. inibidores de RTK conjunto amplo, que visam FGFRs entre outras quinases, pode ser eficaz nestes pacientes [60, 61].
Alterações no celular Proteínas de Ciclo
O viral oncogene homólogo SRC
é mutado em quatro do tumor amostras, duas das mutações são previstos para ter um efeito deletério incluindo a introdução de um codão de paragem. Isso pode contra-indicar inibidores da SRC. TEM
amplificação é também um marcador de resistência conhecido para terapêutica anti-Src como dasatanib [62, 63]. A quinase do ciclo celular relacionados, AURKA
foi amplificado e sobre-expresso em uma amostra. inibidores AURKA estão em desenvolvimento para tumores sólidos [37] e pode ser indicada neste caso. CCNE1
foi amplificado em duas amostras (08390 e 08357). Altos níveis de CCNE1
foram mostrados para ser freqüentemente associada com câncer gástrico precoce e os níveis de metástase mas a expressão não se correlacionam com a sobrevivência [64, 65]. Os níveis elevados de CCNE1
têm sido sugeridas como um marcador de sensibilidade para as terapias dirigidas para genes pró-drogas activadas por enzimas [66]
Activação da via Wnt é comum nas amostras de carcinoma
As mutações foram observadas no APC
gene em 22 amostras. A APC é um supressor tumoral que se sabe activar CTNNB1 e sinalização via Wnt, entre outros efeitos [67]. A via Wnt foi previamente encontrado para ser frequentemente ativada no câncer gástrico [68]. Usamos uma assinatura da transcrição, gerado a partir de estudos anteriores [69, 70] e disponíveis no banco de dados do Broad Institute MSigDB para classificar as amostras de estudo por suas assinaturas wnt de transcrição. A Figura 5A mostra um mapa de calor dos níveis de transcrição dos genes de assinatura WNT nos conjuntos de dados. A activação desta via é mais alta em quase todas as amostras de cancro em comparação com as amostras normais. inibidores de Wnt são objecto de intensa investigação em produtos farmacêuticos e de pesquisa acadêmica [71-73]. Estes resultados sugerem que eles vão ter uma indicação no câncer gástrico, bem como muitos outros tipos de câncer. Figura 5 assinaturas de transcrição através de amostras. heatmap agrupado mostrando expressão de uma genes assinatura Wnt e genes assinatura hedgehog B, através de amostras no estudo. Todos os valores de expressão são Zscore normalizada. Zscore < -1 são azuis, Z-score > 1 são vermelhas com uma coloração graduada através branco em 0. nomes de exemplo estão no eixo x, que estão agrupadas por padrão de expressão e as amostras com alta pontuação de assinatura são para a direita. As amostras com mutações somáticas nonsynonymous APC (A) ou mutações Ptch1 (B) e indicado por um asterisco acima da mapas térmicos. WNT genes assinatura (cima para baixo): FSTL1, DACT1, CD99, LMNA, SERPINE1, TNFAIP3, GNAI2, ID2, MVP, ACTN4, CAPN1, LUZP1, MTA1, RPS19, PTPRE, AXIN2, NKD2, SFRS6, CCND1, SCAP, CPSF4 , SENP2, DKK1, PRKCSH, SLC1A5, HDGF, CBX3, SCML1, PCNA, RPS11, SNRPA1, TGM2, LY6E, IFITM1, NSMAF, TCF20, BCAP31, AXIN1, AGRN, PLEKHA1, SLC2A1, CTNNB1, eIF5A, IMPDH2, GSK3B, PFN1 , UBE, MAP3K11, ARHGDIA, HNRPUL1, FLOT2, GYPC, NCOA3, CENTB1, SYK, POLR2A, KRT5, DHX36, ELF1, SMG2, FGD6, MAPKAP1, LOC389435, RPL27A, SRP19, RPL39L, SFRS2IP, FUSIP1
; genes assinatura do ouriço (cima para baixo):. LRFN4, JAG2, RPL29, genes WNT5A, SNAI2, FST, MYCN, BMP4, CCND1, BMI1, CFLAR, PRDM1, GREM1, FOXF1, CCND2, CD44
ativação do hedgehog via também é comum nas amostras de carcinoma
PTCH1
é um supressor tumoral e actua como um receptor para os ligandos hedgehog e inibe a função de smoothened. Quando smoothened é libertado, ele sinaliza intracelularmente que conduz à activação dos factores de transcrição GLI [74]. Múltiplas mutações somáticas de PTCH1
são registrados no COSMIC, consistentes com sua função supressora de tumores. A mutação D362Y visto neste estudo em FICJG amostra, é no quarto domínio transmembranar de PTCH1 e foi anteriormente visto como uma mutação de linha germinal de perda de função em um doente com síndrome de Gorlin, predispondo a neoplasias (D513Y numerado devido a transcrição diferente ) [75].
 Doença Inflamatória Intestinal (DII) Causas, Sintomas, Tratamento
Doença Inflamatória Intestinal (DII) Causas, Sintomas, Tratamento
 Estudo com gêmeos mostra que os sintomas do COVID-19 têm contribuição genética
Estudo com gêmeos mostra que os sintomas do COVID-19 têm contribuição genética
 5 razões para investir em sua saúde intestinal agora
5 razões para investir em sua saúde intestinal agora
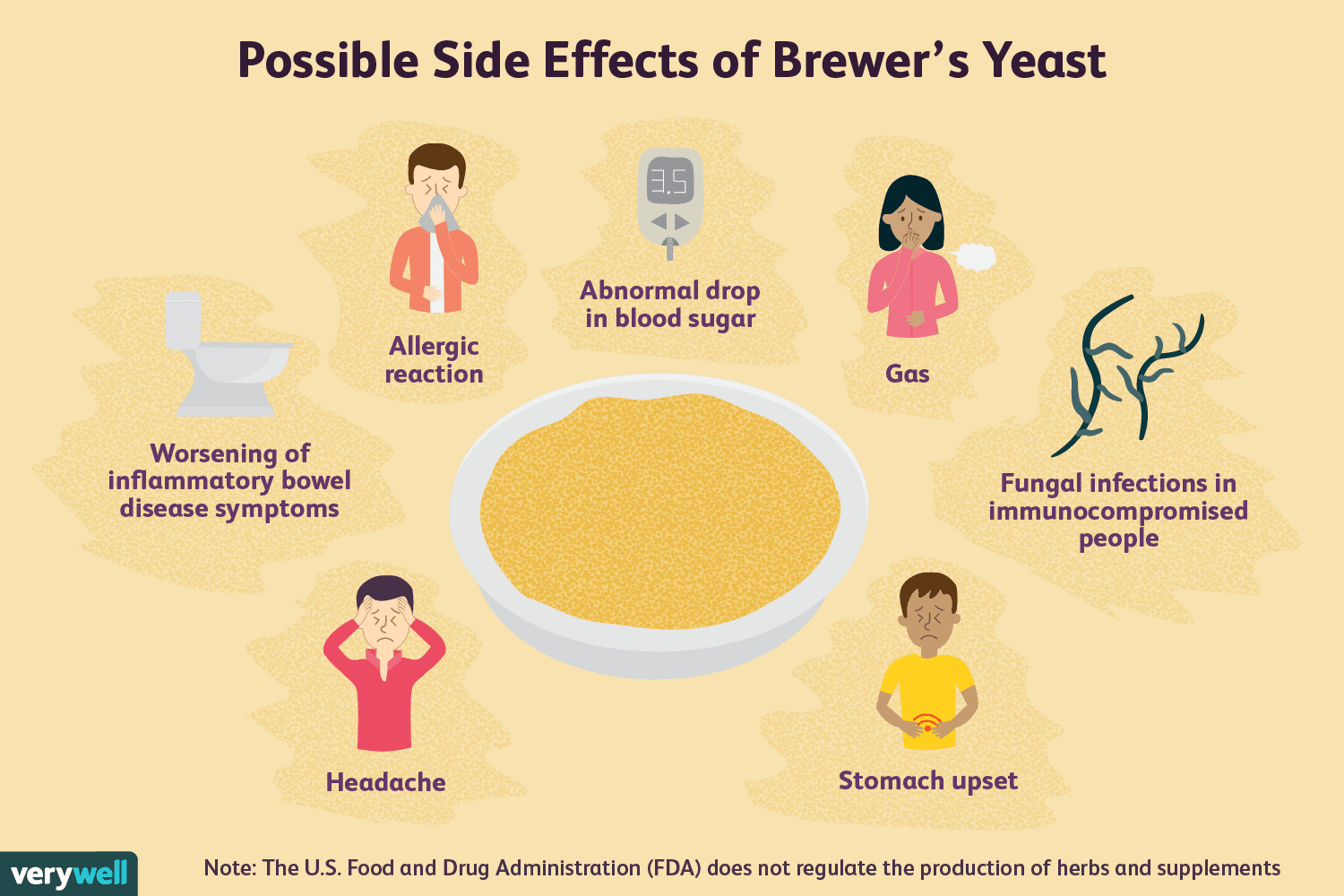 Os benefícios para a saúde da levedura de cerveja
Os benefícios para a saúde da levedura de cerveja
 Quais alimentos você deve evitar se tiver a síndrome do intestino permeável?
Quais alimentos você deve evitar se tiver a síndrome do intestino permeável?
 Dica de saúde:sinais de cálculos biliares
Dica de saúde:sinais de cálculos biliares
 PENTAX Medical levanta $ 125,
000 para o World Cancer Research Fund por meio de sua campanha MILES FOR HEALTH PENTAX Medical, um fornecedor líder de soluções de endo-imagens, comemora os 100 º aniversário da fundação da Asahi Op
PENTAX Medical levanta $ 125,
000 para o World Cancer Research Fund por meio de sua campanha MILES FOR HEALTH PENTAX Medical, um fornecedor líder de soluções de endo-imagens, comemora os 100 º aniversário da fundação da Asahi Op
 Por que fazer uma colonoscopia?
O câncer colorretal continua sendo o segundo maior assassino de câncer nos EUA e é prevalente em todos os grupos raciais e étnicos. Os números por trás do câncer colorretal são assustadores: O risco
Por que fazer uma colonoscopia?
O câncer colorretal continua sendo o segundo maior assassino de câncer nos EUA e é prevalente em todos os grupos raciais e étnicos. Os números por trás do câncer colorretal são assustadores: O risco
 O consumo excessivo de sal é ruim, mesmo para a microbiota intestinal
O sal está em todos os nossos pratos, no pão, no presunto, no queijo e em quase todos os alimentos processados. Costumamos exceder a quantidade recomendada de ingestão de sódio por dia, que segundo a
O consumo excessivo de sal é ruim, mesmo para a microbiota intestinal
O sal está em todos os nossos pratos, no pão, no presunto, no queijo e em quase todos os alimentos processados. Costumamos exceder a quantidade recomendada de ingestão de sódio por dia, que segundo a