Hintergrund
Um das Potenzial von Serum miRNAs als Biomarker für die Früherkennung von Magenkrebs (GC), einer populationsbasierten Studie untersuchen wurde in Linqu, mit hohem Risiko durchgeführt Bereich der GC in China.
Methodik /wesentlichen Ergebnisse
wurden alle Probanden aus zwei großen Kohortenstudien ausgewählt. Differential miRNAs wurden in Serumpools von GC und Kontrolle mittels TaqMan niedriger Dichte Array und validiert in einzelnen identifiziert aus 82 Paaren von GC und Kontrolle, und 46 Paare von Dysplasie und Kontrolle durch quantitative Echtzeit-Reverse Transkription-Polymerase-Kettenreaktion. Die zeitlichen Trends der identifizierten Serum miRNA-Expression wurden weiter in einer retrospektiven Studie von 58 GC-Patienten untersucht, die auf der Grundlage der langfristigen Follow-up-Bevölkerung mindestens eine Pre-GC Diagnose Serumprobe hatte. Die miRNA-Ergebnisse zeigten, dass 16 miRNAs deutlich in GC-Patienten hochreguliert wurden im Vergleich zu den Kontrollen. Eine weitere Validierung identifiziert eine Gruppe von drei Serum miRNAs (miR-221, miR-744 und miR-376c) als potentielle Biomarker für die GC-Erkennung und receiver operating characteristic (ROC) -Kurve basierte Risikobewertung Analyse ergab, dass dieses Panel GCs unterscheiden konnte von Kontrollen mit 82,4% Sensitivität und 58,8% Spezifität. MiR-221 und miR-376c, zeigten eine signifikant positive Korrelation mit schlechter Differenzierung von GC und miR-221 zeigte eine höhere Ebene in Dysplasien als die Kontrolle. Darüber hinaus ergab die retrospektive Studie eine steigende Tendenz dieser drei miRNA Ebenen während GC Entwicklung ( P Schlussfolgerungen /Bedeutung Diese Daten legen nahe, dass Serum miR-221, miR-376c und miR-744 haben ein großes Potenzial als neuartige nicht-invasive Biomarker für die Früherkennung von GC. Citation: Song My, Pan Kf, Su Hj, Zhang L, Ma Jl, Li Jy, et al. (2012) Identifizierung von Serum MicroRNAs als Novel nicht-invasive Biomarker für die Früherkennung von Magenkrebs. PLoS ONE 7 (3): e33608. doi: 10.1371 /journal.pone.0033608 Herausgeber: Joseph Califano, John Hopkins Medical School, Vereinigte Staaten von Amerika Empfangen: 15. November 2011; Akzeptiert: 13. Februar 2012 um; Veröffentlicht: 14. März 2012 Copyright: © 2012 Song et al. Dies ist eine Open-Access-Artikel unter den Bedingungen der Lizenz Creative Commons, die uneingeschränkte Nutzung erlaubt, die Verteilung und Vervielfältigung in jedem Medium, vorausgesetzt, der ursprüngliche Autor und Quelle genannt werden Finanzierung:. Diese Arbeit wurde durch Zuschüsse aus dem National Grund Research Program von China (973 Programm: 2010CB529303) unterstützt, A3 Foresight-Programm von Natural Science Foundation of China (30921140311) und der National Natural Science Foundation of China (81041012 und 81171989). Die Geldgeber hatten keine Rolle in Studiendesign, Datenerfassung und Analyse, Entscheidung oder Vorbereitung des Manuskripts zur Veröffentlichung Konkurrierende Interessen:.. Die Autoren haben erklärt, dass keine Interessenkonflikte bestehen Einführung Magenkrebs (GC) ist die zweithäufigste Ursache für Krebstod in der Welt, mit fast die Hälfte in China auftreten [1], [2]. Die Prognose der GC variiert bemerkenswert durch das Stadium der Krebs mit der 5-Jahres-Überlebensrate 90% in Stufe I erreicht aber weniger als 5% in Stadium IV [3]. Daher ist die Früherkennung von GC eine der wichtigsten Maßnahmen, die Sterblichkeit und die Verbesserung der Prognose von GC zu reduzieren. Obwohl gastroskopischen Screening für GC sehr zuverlässig ist, ist es invasive und teuer, vor allem für die Entwicklungsländer. Aus diesem Grund hat viel Mühe gemacht worden in leicht zugänglichen Proben weniger teuren vorläufigen Screening-Tests zu entwickeln. viele bisher untersuchten Analyten, wie beispielsweise Pepsin (PG) I /II-Verhältnis, das karzinoembryonale Antigen (CEA) und Kohlenhydrat-Antigen 19-9 (CA 19-9), waren jedoch nicht sensitiv und spezifisch genug für die GC-Screening [3], [ ,,,0],4]. Daher besteht ein dringender Bedarf an neuen nicht-invasiven Biomarker die Früherkennung von GC zu verbessern. MicroRNAs (miRNAs) sind eine reiche Klasse von kleinen nicht-kodierenden RNAs, die sich negativ auf die Genexpression durch Basenpaarung regulieren mit die 3'-untranslatierte Region des Ziel-mRNAs, was entweder mRNA-Spaltung oder translationale Repression [5], [6]. Zunehmende Beweise haben gezeigt, dass miRNAs in verschiedenen biologischen Prozessen beteiligt sind, einschließlich der Entwicklung, Zelldifferenzierung, Proliferation und Apoptose [7] und in menschlichen Karzinogenese als Onkogene oder Tumorsuppressoren teilnehmen [8]. Studien haben gezeigt, dass miRNA Expressionsprofil unter Tumorarten unterschiedlich und können Krebs und normalen Geweben [9], [10] unterscheiden. Vor kurzem haben zirkulierende miRNAs großes Potential als Biomarker für viele Krebsarten vorgeschlagen worden, einschließlich GC [11] - [16]. Allerdings hat sich der Wert von miRNAs bei der Früherkennung von GC zirkulierenden noch nicht berichtet worden. Seit 1989 haben wir eine Reihe von Studien in Linqu County durchgeführt, einem Gebiet mit hohem Risiko von GC in der Provinz Shandong, China , einer bevölkerungsbasierten Kohortenstudie von präkanzerösen Läsionen des Magens einschließlich [17], [18] und einer randomisierten Studie die Progression der Magen-Läsionen [19] zu hemmen. Diese Kohorte erlaubt uns, die dynamischen Veränderungen in zirkulierenden miRNA Ebenen während GC Entwicklung zu untersuchen, und bietet uns eine einmalige Gelegenheit, das Potenzial der zirkulierenden miRNAs bei der Früherkennung von GC zu erkunden. Wir berichten hier über die Ergebnisse der Differential berichten miRNAs im Serum von GC und Dysplasie (DYS) und einer retrospektiven Studie entwickelt, um die dynamische Veränderungen während der GC-Entwicklung zu bewerten. Materialien und Methoden Studiendesign und Bevölkerung Die Einzelheiten der Studienpopulation, die Verfahren der endoskopischen Untersuchung, die Kriterien von Magen-Histopathologie und Follow-up an anderer Stelle beschrieben [17] - [19]. Kurz gesagt, eine endoskopische Screening-Umfrage wurde im Jahr 1994 im Jahr 1989 unter 3399 Einwohner im Alter von 35-64 Jahren in Linqu County, einem Gebiet mit hohem Risiko von GC, und die Probanden ohne GC Diagnose wurden anschließend folgte mit der wiederholten endoskopische Untersuchung durchgeführt, ins Leben gerufen [17 ], [18]. 1995 wurde eine randomisierte, Placebo-kontrollierten Interventionsstudie wurde in dieser Population bis 2003, als wiederholte endoskopische Untersuchung eingeleitet wurde durchgeführt [19]. Alle Probanden unterzogen Blutentnahme sowohl in den Basis- und Endpunkt Umfragen jeder Studie, und einige mit fortgeschrittenem Magen-Läsionen, wie intestinale Metaplasie (IM) oder DYS, auch Blut zur Verfügung gestellt und erhielt endoskopische Untersuchung in den Jahren 1992 und 1999 in der aktuellen Studie, ein mehrstufiges, verschachtelt Fall-Kontroll-Studie von zwei großen Kohorten wurde entwickelt, um die Differential Serum miRNAs in GC und DYS zu identifizieren und zu validieren und eine retrospektive Studie wurde durchgeführt, das Potenzial der Kandidaten Serum zu untersuchen miRNAs bei der Früherkennung von GC (Abbildung 1). Diese Studie wurde in vier Phasen unterteilt schrittweise. In Phase I, Serumpools von 14 GC-Patienten und 14 alters- und geschlechts gematchten Kontrollen mit der Diagnose oberflächliche Gastritis (SG) oder milde chronische atrophische Gastritis (CAG) wurden verwendet, miRNA Profile von Array-Analyse zu erzeugen. Differential miRNAs identifiziert wurden, und die extrem upregulated miRNAs oder mäßig upregulated miRNAs mit funktionellen oder Biomarker-bezogene Berichte wurden für die weitere Analyse ausgewählt. Verwendung quantitative reverse Transkription-Polymerase-Kettenreaktion (qRT-PCR) auf den einzelnen Serumproben von 14 GCs und Kontrollen in der Phase II wurden identifiziert miRNAs validiert, die für das Array-Test in der Phase I. In Phase III, die signifikant verändert miRNAs verwendet wurden wurden anschließend in 68 und 46 GC DYS Patienten sowie deren alters- und geschlechts gematchten Kontrollen validiert. Receiver operating characteristic (ROC) -Kurve basierte Risikobewertung Analyse wurde dann unter den 68 GC-Steuerpaare ausgeführt, um die diskriminierende Wirkung von Serum miRNAs auf GC zu bewerten. Jedes Subjekt wurde entweder in durch Vergleich der Expressionsniveaus von miRNA Biomarkern in die entsprechenden Cut-Off-Werten abgeleitet von ROC-Kurve Hoch- oder Niedrigrisikogruppen zugeordnet. In der Phase IV wurde eine retrospektive Studie in 58 GC-Patienten durchgeführt, die in der Langzeit-Follow-up-Studie mindestens einem der förderfähigen Vordiagnose Serumprobe zur Verfügung gestellt, wie oben beschrieben, die zeitliche Entwicklung von potenziellen miRNA-Biomarker zu erforschen und zu bestimmen, ihre Wert in der frühen GC-Detektion. Die gesamte Studie wurde von der Institutional Review Board der Universität Peking Cancer Hospital &genehmigt; Institute, und alle Probanden gaben schriftliche Einverständniserklärung. Bis zu 5 ml Vollblut von jedem Fasten Teilnehmer wurde mit Fällen und Kontrollen im selben Jahr fertig gesammelt. Blutproben wurden für 30-40 min und die Serumtrennung stehen gelassen wurde 15 min bei 965 g durch Zentrifugation erreicht. Der Überstand Serum wurde bei -80 ° C bis zur Analyse zurückgewonnen und gespeichert. RNA wurde aus 100 &mgr; l Serum unter Verwendung des miRNeasy Mini Kit (Qiagen, Deutschland) nach Herstellerprotokoll mit geringen Modifikationen isoliert [20]. und 14 geschlechts- und altersangepassten Kontrollen; Für miRNA Profilierung wurden zwei Pools durch die Kombination der Mischungen aus oberen wässrigen Phase nach der Phasentrennung und Ethanol aus 14 GC-Patienten (Alter, 62 (SD 6,4) Jahre 12 männlich, 2 weiblich) erstellt (Alter, 61 (SD 5,9) Jahre), respectively. Nach der Bindung an die Membran von RNeasy Mini spin column und anschließendes Waschen wurde die RNA mit 40 &mgr; l RNase-freiem Wasser eluiert. Für alle qRT-PCR-Experimente, extrahierte RNA aus 100 &mgr; l Serum mit 100 &mgr; l RNase-freiem Wasser eluiert. Zur Normalisierung wurde die Probe zu Probe Variation in RNA-Isolierung Schritt, synthetische ath-miR-159a auf jede Serumprobe für miRNA Profilierung hinzugefügt, während Cel-miR-39 für qRT-PCR-Analyse hinzugefügt wurde, wie von Mitchell beschrieben et al miRNA- miRNA Profilierung wurde unter Verwendung von TaqMan niedriger Dichte Array A (v2.0) durchgeführt gemäß der empfohlenen Protokoll des Herstellers (Applied Biosystems, Foster City, CA, USA). Diese qRT-PCR Profilierungsplattform besteht aus einem 384-well mikrofluidischen Karte, in der 377 miRNAs zusammen mit drei endogene Kontrollen (U6, RNU44 und RNU48) und eine negative Kontrolle ohne Bezug auf menschliche (ATH-miR-159a) analysiert werden kann. Kurz zusammengefasst die RNA getrennt von 14 GC-Patienten und Kontrollen angepasst, wie oben beschrieben wurde revers transkribiert die TaqMan miRNA reverse Transkription-Kit und RT-Assays Multiplex TaqMan miRNA (human Pool; Applied Biosystems). Für jede RNA-Pool, 3 &mgr; l Gesamt-RNA wurde auf jede der Multiplex-reversen Transkriptionsreaktionen zugesetzt. Um die Menge der cDNA für die Analyse verfügbar, Anreicherung von Zielgenen erhöht wurde mit einer Vorverstärkung Schritt durchgeführt. Die 25-ul-Reaktionsgemisch bestand aus 2,5 ul unverdünntem cDNA kombiniert mit 12,5 ul TaqMan PreAmp Master Mix (2 x), 2,5 ul Megaplex PreAmp Primers (10 x) und 7,5 ul Nuklease-freies Wasser. Die Vorverstärkung Schritt wurde auf einem GeneAmp PCR System 9700 (Applied Biosystems) durchgeführt gemäß dem Protokoll des Herstellers. Für die Echtzeit-PCR-Analyse wurde das Produkt durch Zugabe von 75 ul Nuklease-freies Wasser verdünnt und 9 ul der verdünnten Mischung wurde anschließend mit 450 ul TaqMan 2 × Universal-PCR Master Mix ohne Uracil-N-Glycosylase und 441 ul Nuklease-freies Wasser kombiniert. Nach dem Laden 100 ul jeder Multiplex-Pool Mischung wurde das Array zentrifugiert und versiegelt. Amplifikation wurde auf einem Applied Biosystems 7900 HT Thermocycler (Applied Biosystems) unter Verwendung der vom Hersteller empfohlenen Zyklusbedingungen durchgeführt. Die Daten wurden mittels RQ-Manager-Software Version 1.2 und Dataassist ™ Software Version 2.0 (Applied Biosystems) analysiert. Nach der Normalisierung wurde unter Verwendung der gespickt-in ath-miR-159a, die Falte Änderung von miRNAs Ebenen in GC im Vergleich zur Kontrolle nach der Methode berechnet. die reverse Transkription wurde unter Verwendung des TaqMan miRNA reverse Transkription-Kit und miRNA-spezifische Stem-Loop-Primer (Applied Biosystems) durchgeführt. Die 7,5-ul-Reaktionsgemisch bestand aus 0,75 &mgr; l von 10 × Reverse Transkriptionspuffer, 0,095 &mgr; l RNase-Inhibitor (20 Einheiten /ul), 0,075 &mgr; l 100 mM dNTPs mit dTTP, 0,5 ul MultiScribe Reverse Transkriptase (50 Einheiten /ul), 1,5 ul Primer, 2,5 &mgr; l RNA und 2,08 &mgr; l RNase-freiem Wasser. Die reverse Transkription wurde auf einem T professionellen PCR-System durchgeführt (Biometra, Deutschland) gemäß dem Protokoll des Herstellers. Real-time PCR wurde in zweifacher Ausfertigung und enthalten ohne Matrize Negativkontrollen durchgeführt. Für die 20-ul-Reaktion, cDNA (3 ul) wurde mit 10 ul 2 x TaqMan Universal-PCR Master Mix ohne Uracil-N-Glycosylase kombiniert, 1 ul des TaqMan miRNA Assay-Mischung, und 6 ul Wasser. Die Amplifikation wurde auf einem 7500 Real-Time-PCR-System (Applied Biosystems) nach dem Protokoll des Herstellers durchgeführt. Die Expression von miRNAs wurden dem Stachel-in Cel-miR-39 normalisiert, und wurden nach der Methode berechnet. Die paarweise t
für Trend < 0,05), und dieser Platte konnte Serumproben gesammelt bis zu 5 Jahre vor der klinischen GC Diagnose mit 79,3 klassifizieren Gesamtgenauigkeit%.
Probenverarbeitung und RNA-Extraktion
[11].
miRNA Quantifizierung mittels real-time qRT-PCR
Die statistische Analyse
Test wurde verwendet, um den Unterschied in der mittleren Alter zwischen Gruppen zu untersuchen. Der Wilcoxon-Test oder Mann-Whitney-U-Test wurde verwendet, um die Serum miRNA Niveaus verschiedener Gruppen zu vergleichen, wo angemessen. ROC-Kurven wurden die diagnostischen Wirkung von miRNAs auszuwerten hergestellt und der verallgemeinerten linearen Modell mit wiederholten Messungen wurde die zeitlichen Trends der Serumspiegel während miRNA GC Entwicklung nach logarithmischer Transformation zur Analyse verwendet. Alle P
Werte waren zweiseitig und weniger als 0,05 wurde als statistisch signifikant angesehen. Alle statistischen Analysen wurden die Statistical Package mit für Sozialwissenschaften durchgeführt (SPSS 13.0, Chicago, IL).
Ergebnisse
 Wissenschaftler beweist Rolle des Mikrobioms bei Fettleibigkeit
Wissenschaftler beweist Rolle des Mikrobioms bei Fettleibigkeit
 Starkes Mikrobiom im frühen Leben in Verbindung mit weniger Atemwegsinfektionen
Starkes Mikrobiom im frühen Leben in Verbindung mit weniger Atemwegsinfektionen
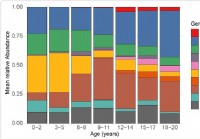 Veränderung des Mikrobioms der oberen Atemwege bei Kindern im Zusammenhang mit der Anfälligkeit für SARS-CoV-2
Veränderung des Mikrobioms der oberen Atemwege bei Kindern im Zusammenhang mit der Anfälligkeit für SARS-CoV-2
 Fischschleim könnte eine potenzielle Quelle für Antibiotika sein
Fischschleim könnte eine potenzielle Quelle für Antibiotika sein
 Patienten mit Immuntherapien sollten mehr Ballaststoffe zu sich nehmen,
Patienten mit Immuntherapien sollten mehr Ballaststoffe zu sich nehmen,
 Studie gibt Aufschluss über die Ursachen von schwächenden Darmschmerzen
Studie gibt Aufschluss über die Ursachen von schwächenden Darmschmerzen
 Ist der Kaiserschnitt gut für die Gesundheit von Kindern?
Eine aktuelle Studie des Instituts für Wirtschaftsforschung VATT legt nahe, dass eine ungeplante CS bei einer Indikation durchgeführt wurde, die vielleicht hätte vermieden werden können, bei Asthma er
Ist der Kaiserschnitt gut für die Gesundheit von Kindern?
Eine aktuelle Studie des Instituts für Wirtschaftsforschung VATT legt nahe, dass eine ungeplante CS bei einer Indikation durchgeführt wurde, die vielleicht hätte vermieden werden können, bei Asthma er
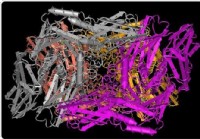 Neues Tool zur Entschlüsselung des Darmmikrobioms
Die Millionen von Bakterien im Darm spielen eine sehr wichtige Rolle für Gesundheit und Krankheit. Jedoch, ein ständiges Thema war das fehlende Verständnis der tatsächlichen Zusammensetzung des gesund
Neues Tool zur Entschlüsselung des Darmmikrobioms
Die Millionen von Bakterien im Darm spielen eine sehr wichtige Rolle für Gesundheit und Krankheit. Jedoch, ein ständiges Thema war das fehlende Verständnis der tatsächlichen Zusammensetzung des gesund
 Obere Endoskopie
Wenn Sie chronisches Sodbrennen haben, Bauch- oder Brustschmerzen, Brechreiz, und Erbrechen, oder andere Probleme, die ihren Ursprung im oberen Verdauungssystem haben, wir können eine obere Endoskopie
Obere Endoskopie
Wenn Sie chronisches Sodbrennen haben, Bauch- oder Brustschmerzen, Brechreiz, und Erbrechen, oder andere Probleme, die ihren Ursprung im oberen Verdauungssystem haben, wir können eine obere Endoskopie