Abstract
Om de patronen van genexpressie in maagkanker te verkennen, een totaal van 26 gepaarde maagkanker en goedaardige weefsels van patiënten werden ingeschreven voor genexpressie microarray analyses. Limma methoden werden toegepast om de gegevens te analyseren en genen werden als significant verschillend tot expressie als de False Discovery Rate (FDR) waarde was < 0,01, P Visum:. Li H Yu B, Li J, Su L, M Yan, Zhang J., et al. (2015) Karakterisering van differentieel tot expressie genen betrokken bij Pathways geassocieerd met maagkanker. PLoS ONE 10 (4): e0125013. doi: 10.1371 /journal.pone.0125013 Academic Editor: Francisco J. Esteban, Universiteit van Jaen, Spanje Ontvangen: 9 november 2014; Aanvaard: 6 maart 2015; Gepubliceerd: 30 april 2015 Copyright: © 2015 Li et al. Dit is een open toegang Artikel gedistribueerd onder de voorwaarden van de Creative Commons Attribution License, die onbeperkt gebruik, distributie en reproductie maakt in elk medium, op voorwaarde dat de oorspronkelijke auteur en de bron worden gecrediteerd Data Beschikbaarheid: Alle relevante gegevens zijn binnen het papier en de Ondersteunende informatie bestanden. Alle microarray-bestanden zijn verkrijgbaar bij de NCBI Gene Expression Omnibus (GEO) database (toetreding nummer "GSE65801"; http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE65801). Financiering: Dit werk werd ondersteund door subsidies voor de analyse van de National Natural Science Foundation of China [No. 81172324, nr 91229106, nr 81272749, en nr 81372231], Wetenschap en Technologie Commissie van Shanghai Gemeente [No. 13ZR1425600], en Key Projecten in de National Science & Technology Pillar Program van China (Nr 2014BAI09B03). De financiers hadden geen rol in de studie design, het verzamelen van gegevens en analyse, besluit te publiceren, of de voorbereiding van het manuscript Competing belangen:.. De auteurs hebben verklaard dat er geen tegenstrijdige belangen bestaan Introductie Maagkanker (GC) is een van de meest voorkomende kankers wereldwijd, en de incidentie is bijzonder hoog in Oost-Azië, met name in China. Ongeveer 952.000 nieuwe gevallen van maagkanker Wereldwijd werden gediagnosticeerd in 2012, en de helft van hen deed zich voor in Oost-Azië (vooral in China) [1]. In China, de meeste patiënten met GC worden gediagnosticeerd in een laat stadium met een slechte prognose. Daarom is het ophelderen van de moleculaire mechanismen die ten grondslag liggen GC progressie is essentieel voor het identificeren van de belangrijkste biomarkers en de ontwikkeling van doeltreffende gerichte therapieën. In de afgelopen tien jaar hebben de genexpressie microarrays een gemeenschappelijk instrument voor de behandeling van gen transcript niveaus in het kankeronderzoek geworden. Microarray gegevens worden gebruikt voor allerlei analyses, zoals ongecontroleerde clustering, classificatie, differentiële expressie analyse en expressie brengen van quantitative trait loci [2]. Het is niet alleen helpt om de belangrijkste disfunctionele genen in kanker te identificeren, maar biedt genoom-brede informatie over de genexpressie op een bepaald moment ook [3,4]. In deze studie hebben we voerden een genoom-brede onderzoek van de expressie van lncRNAs en mRNA van gepaarde monsters van primaire maagkanker weefsels en goedaardige weefsels, de differentieel tot expressie lncRNAs en coderende RNAs profileren. Studie van deze gegevens zal waardevolle informatie verschaffen over het mechanisme van carcinogenese en laat de ontdekking van belangrijke genen die als toekomstig doelwit van anti-kanker therapie kan handelen. ethische verklaring Schriftelijke toestemming werd verkregen van alle deelnemers. De studie werd goedgekeurd door de Human Research Ethics Committee van Ruijin Hospital, Shanghai Jiao Tong University, School of Medicine. De weefsels werden genomen uit primaire maag carcinomen van onbehandelde patiënten die D2 ondergingen radicaal gastrectomy in Shanghai Ruijin Hospital. Voor elke kankerweefsel, werd een gepaarde goedaardig weefselmonster vanuit het nabijgelegen gebied tegelijk. De omvang van elk monster was ongeveer 0,1 cm 3. Alle monsters werden na excisie in RNAlater geplaatst in 15 minuten en opgeslagen in vloeibare stikstof tot RNA-extractie. In deze studie werden 32 gepaarde weefsels verzameld voor de microarray en 26 gepaarde monsters werden ingeschreven voor de volgende stap analyse van GO, route en het netwerk na kwaliteitscontrole met behulp van 3D Principal component analyse (3D-PCA) en Cluster analyse. microarray experimenten Agilent SurePrint G3 Human GE 8x60K Microarray (Design ID: 028.004) werd gebruikt in deze studie. Totaal RNA werd geïsoleerd en versterkt met een lage input Quick Amp Labeling Kit, One-Color (Catȇ0-2305, Agilent Technologies, VS). Vervolgens werden de gemerkte cRNA gezuiverd door RNeasy Mini Kit (Cat˥06, Qiagen, Duitsland). Op basis van aanwijzingen van de fabrikant, werd elke dia gehybridiseerd met 600ng Cy3-gelabeld cRNA met een genexpressie Hybridization Kit (CatȆ8-5242, Agilent Technologies, VS) en gewassen door de Gene Expression Wash Buffer Kit (CatȆ8-5327, Agilent Technologies, VS). Een Agilent Microarray Scanner (Cat # G2565CA, Agilent technologieën, VS) en Feature Extraction software 10,7 (Agilent Technologies, VS) werden toegepast op elke dia met dezelfde instellingen weergegeven als volgt, Dye channel scan: Green, scanresolutie = 3 pm, 20bit. De ruwe gegevens werden genormaliseerd door de kwantiel algoritme, Gene Spring Software 11.0 (Agilent Technologies, VS) (gedetailleerd in S5 tabel). Lineaire modellen en empirische Bayes methoden werden toegepast om te analyseren de gegevens in deze studie. De resulterende P GO categorie We voerden Gene Ontology (GO) analyse van de functies van het differentieel tot expressie van genen te analyseren in onze microarray volgens de belangrijkste functionele indeling van het National Center for Biotechnology Information (NCBI). In het algemeen, exact test en Fisher's de χ Pathway analyses Pathway aantekeningen van de differentiële exressed genen werden verkregen van KEGG (http: //www .genome.jp /KEGG /). Weg categorieën met een FDR < 0,01 werden gemarkeerd. De verrijking van belangrijke trajecten werd gegeven door: verrijking = /, die ons geholpen om meer belangrijke wegen vinden in onze studie ( n Gene-Act netwerk Volgens de KEGG-database, kan een gen worden betrokken bij een aantal wegen of een wisselwerking met een aantal andere genen. Al de gen-gen interacties werden samengevoegd tot de Gene-Act netwerk op basis van het differentieel wegen, die ons helpen om de signaalwegen en belangrijke regulerende genen in GC onthullen bouwen. gen co-expressie netwerk is gebouwd volgens de genormaliseerde signaalintensiteit specifieke expressie genen. Graad centrale wordt gedefinieerd als het aantal verbindingen één knooppunt naar het andere, waarbij het relatieve belang van genen bepaalt. Bovendien werden k-kernen toegepast als een werkwijze voor vereenvoudiging van de grafiek topologie analyses. Core regulerende factoren (genen) dat de hoogste graden verbinden meest naburige genen en bouw de structuur van het netwerk (gespecificeerd in tabel S5). Totaal RNA werd geëxtraheerd uit weefsels met behulp van het Trizol reagens (Invitrogen) volgens de instructies van de fabrikant. De kwantitatieve real-time polymerase-kettingreactie (PCR) werd uitgevoerd met SYBR-groen PCR Master Mix in een Fast real-time PCR System 7500 (Applied Biosystems). De primers van de 10 genen toonde in Tabel S4. PCR reacties werden uitgevoerd bij 50 ° C gedurende 2 minuten, gevolgd door 40 cycli van 95 ° C gedurende 15 s en 60 ° C gedurende 1 min. ACt werd berekend door de Ct van β-actine RNA (controle) van de Ct van het RNA van het monster, respectievelijk. ΔΔCt werd vervolgens berekend door de ACt van de controle van de ACt van het monster. Voudige verandering werd berekend door de vergelijking 2-ΔΔCt. SPSS-software 19 en Microsoft Excel 2010 werd gebruikt om de gegevens te analyseren. Expressieniveaus tussen kanker weefsels en aangrenzende goedaardige weefsels werden geanalyseerd door gepaarde-sample t-test. P Microarray analyses In totaal 42.405 menselijke genen werden in onze studie geprofileerd door met behulp van een Agilent G3 Human GE 8x60K microarray. We hebben onze dataset in de repository van "Gene Expression Omnibus" ingediend, en de toetreding nummer was "GSE65801" (http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE65801). We gebruikten lineaire modellen en empirische Bayes methoden om de gegevens te analyseren (zie Methoden). Er waren 2.371 mRNA en 350 lncRNAs beschouwd als de differentieel tot expressie van genen door limma voor de volgende stap-analyse (figuur 1A). Van alle 2371 differentiële mRNA, zijn er 1142 mRNA down-gereguleerd en 1229 mRNA up- geregeld in onze waarneming op veranderingen van genexpressie tussen maagkanker en controle weefsels (figuur 1C). De meeste mRNA's differentieel bewezen worden gecorreleerd met kanker en metastase in de meeste soorten kanker (tabel 1). De genen zoals GKN2, PGC, MUC6, Chia, PSCA en FBP2 behoorden tot de top 20 down-gereguleerde genen, terwijl KLK8, sFRP4, INHBA, CLDN1, CST1, FAP, SPP1, OLFM4 en KRT17 behoorden tot de top 20 omhoog -gereguleerde genen (Tabel 1). Echter, sommige genen zoals HOXC9, FNDC1, Stra6, KCNE2, PGA3 en KCNJ16 zijn niet gemeld bij maagkanker en hun rol blijven onbekend (zie tabel 1). Daarnaast hebben we 193 down-gereguleerd lncRNAs en 156 opgereguleerd lncRNAs tussen totaal 350 differentiële lncRNAs gebaseerd op de profilering (figuur 1B). Het merendeel van de lncRNAs zijn niet gegeven een officiële namen en hun functies blijven onbekend. Toch hebben sommige gemeld spelen cruciale rol bij kanker, zoals H19, GUCY1B2, MEG3 en AKR7L (tabel 2). In ons vorige rapport [36], de voudige verandering (FC) van H19 in 74 maagkanker versus gepaarde goedaardige weefsels was 6,015, met een P Alle differentieel tot expressie gebrachte genen werden ingedeeld in verschillende functionele categorieën volgens de Gene Ontology (GO) project voor biologische processen. Op basis van onze microarray data, analyses GO aangegeven dat 208 GO termen werden verrijkt ( P Restaurant < 0,01, FDR < 0,01) (S1 tabel). De hoofdcategorieën GO 170 up-gereguleerd GO termen waren gericht op celadhesie, angiogenese, meercellig organisme ontwikkeling, axon begeleiding, ontwikkeling van het skelet systeem, collageen fibrillen organisatie, positieve regulatie van angiogenese, verwonding en negatieve regulatie van celproliferatie (Fig 2A) . De belangrijkste GO categorieën voor-down gereguleerde genen waren spijsvertering, lichaamsvreemde stofwisseling, transmembraan transport, ionentransport, klein molecuul stofwisseling, negatieve regulatie van de groei, glutathion metabolisme, cellulaire respons op cadmium ion en stofwisseling (figuur 2B). Volgens het differentieel genen en functies, bouwden we een GO Boom om de interacties tussen al het differentieel GO categorieën te verkennen. De diversiteit in deze categorieën bij het vergelijken van kankerachtige en controle weefsels voorgesteld maagkanker kan worden geassocieerd met significant opgereguleerd celmigratie, celproliferatie, angiogenese, cel-cel adhesie en celoppervlak receptor signaalroutes, terwijl celmetabolisme processen en ionen transmembraantransport zijn downgereguleerd (figuur 3). Pathway analyses Pathway analyses werden gebruikt om de betekenis die geassocieerd zijn met differentieel tot expressie gebrachte genen volgens KEGG identificeren. Er waren 32 opgereguleerd paden en 31 neerwaarts gereguleerd trajecten voor onze gegevens (figuur 4). Bovendien is de route profilering kwam overeen met de resultaten van de GO categorieën kankergerelateerde biologische functies. Onze data toonde enkele differentiële genen sterk up-gereguleerd, die suggereerde de betrokken trajecten werden activiated. Zo werden sFRP4, WNT11, FZD2, MYC hoog tot expressie in kankerweefsel overeenkomende met de Wnt route werd activiated en BCL2A1, ICM1, TNFSF14 NF-kB route waren sterk tot expressie ook. De meeste kankergerelateerde signaalwegen zoals JAK /STAT, Wnt, NF-KB, PI3K, mTOR, Hedgehog en Notch pathways werden geactiveerd bij maagkanker opzichte van kankerachtige weefsels op basis van onze gegevens (tabel S2). De opgereguleerd routes die waren gericht op celadhesie, transcriptionele ontregeling, carcinogenese en differentiatie werden gecorreleerd met tumorogenese en metastase (figuur 4A). Echter, de-down gereguleerd paden waren over het algemeen verantwoordelijk voor het metabolisme (figuur 4B). Op basis van de GO categorieën en pathway-analyse, kan een gen worden betrokken bij een aantal wegen of interactie met verscheidene andere genen. We gepoolde het differentieel genen en is een netwerk van de interacties van differentieel tot expressie gebrachte genen. Een hoge proteïne regelt of wordt gereguleerd door vele andere eiwitten, die een belangrijke rol in de Gene-Act netwerk (S3 tabel) impliceert. De glutathion S-transferase (GST) familie, cytochroom P450 (CYP) familie, UDP glucuronosyltransferase 2 (UGT2) familie, epidermale groeifactorreceptor (EGFR) familie en cAMP-afhankelijke proteïne kinase katalytische beta (PRKACB) waren in de kern van de gen-gen interactie netwerk. Zij kunnen een belangrijke rol spelen in het netwerk, omdat zij bezaten de sterkste mate (graad > 25) centraliteiten (gen-gen interacties) (Figuur 5). Er werd gemeld dat GST, EGFR en PRKACB verantwoordelijk zijn voor signaaltransductie routes betrokken bij tumorgroei en differentiatie in verschillende kankersoorten [42,43]. produceerden we een gen co-expressie netwerk op basis van de differentieel tot expressie gebrachte genen, eiwitten en eiwit complex in kankerweefsel en goedaardige (controle) weefsels, respectievelijk. In vergelijking met de controlegroep, de verbindingen tussen genen in kanker weefsels minder, waaruit bleek dat de meeste fysiologische gen-gen interacties en interrelaties normale weefsels was gebroken of verloren in de kankerweefsels (Fig 6A en 6B). De genen met een hoge graad en k-core wat betekent dat ze het grootste deel van de interacties met andere geneswere bekend als belangrijkste genen in de interactie netwerk (figuur 6B), waaronder TRO, GPR124, TIMP2, EMCN, SLIT3, HTRA1, SPARC, LAMA4 en MEOX2 bezat (Tabel 3). Zij waren verantwoordelijk voor celsignalering, adhesie, angiogenese, migratie, groei en metastase. We voerden kwantitatieve real-time PCR (qPCR) op 6 opwaarts gereguleerd genen (COL1A, BGN, SPP1, MELK, IGFBP4, SPARC) en 4 down-gereguleerde genen (PGC, SST, MT1X, S100P) om onze gegevens te controleren bij maagkanker weefsels (Tumor) en goedaardige weefsels (Normaal). De expressie verhoudingen van deze 10 genen (Tumor /Normal) vanaf qPCR zijn consistent met die van microarray (Tabel S4). Zij stelde de gegevens van differentiële genen expressie van microarray was betrouwbaar. Wat meer is, heeft ons team gewerkt aan een aantal van de differentiële genen zoals PHF10 [55], CEACAM6 [56], sFRP1 [57], SOX11 [58], CLDN1 [59] om hun expressie en functies te onderzoeken bij maagkanker en de resultaten bleek perfecte onze microarray data. Discussie microarray genexpressie analyses maagkanker zijn eerder gebruikt om diagnostische merkers [60] en gen-expressiepatronen geassocieerd met prognose identificeren voorspellen [ ,,,0],61,62], maar het is niet gebruikt voor moleculaire interacties tussen lncRNAs en mRNAs in GC onthullen. In deze studie analyseerden we 26 maagkanker weefsels met gekoppelde kankerachtige weefsels en geprofileerd de genen die differentieel tot expressie naar hun GO categorieën, wegen, Gene-Act netwerk en Co-expressie netwerk. De resultaten gen expressie werden verkregen met behulp van een Agilent G3 Human GE 8x60K microarray, die niet alleen betrekking op de transcriptoom databases mRNA targets maar omvat ook probes voor lncRNAs (lange niet-coderende RNA's). Met de combinatie van mRNA en lncRNAs kan twee experimenten op één microarray en lncRNA functie en interactie met mRNAs voorspellen. Uit de analyses bleek een set van genen die differentieel tot expressie werden gebracht tussen maagkanker en normaal weefsel. Sommigen van hen zijn eerder gerapporteerd in de maag of andere kankers. Expressie van gastrokine-2 (GKN2) significant neerwaarts gereguleerd of afwezig bij maagkanker cellijnen, maag- intestinale metaplasie en tumorweefsels. Overexpressie van GKN2 bijgedragen aan celproliferatie, migratie en invasie van maagkanker en gearresteerd de celcyclus in de G1-S overgangsfase [6]. Daarentegen expressieniveaus van inhibine A beta (INHBA) waren in kankerweefsel significant hoger dan in aangrenzend normaal mucosa en het wordt beschouwd als een onafhankelijke prognostische factor bij maagkanker [22]. Bovendien ontdekten we enkele nieuwe genen, zoals TMEM184A, PSAPL1, KIAA1199, CLRN3 en FNDC1, die niet zijn vermeld in maagkanker vroeger, en hun rol in kanker blijft onbekend. Een van de voordelen van onze genexpressie microarray analyse is dat vertegenwoordigde de expressie van mRNA en lncRNAs zodat beide samen kunnen worden onderzocht. Onze eerder verslag uit over de rol van lncRNA H19 en het netwerk GC [36] is gebaseerd op de microarray. De meeste van de lncRNAs zoals DRD5, FMO6P, SNAR-A3 en TPRXL toonde in onze microarray zijn niet geïdentificeerd en moeten verder onderzoek naar hun rol verduidelijken maagkanker. Op basis van genexpressie profilering data, de genen en hun functies geactiveerd maagkanker waren verantwoordelijk voor proliferatie, adhesie, migratie en metastase, die in overeenstemming met de resultaten van analyses route was. Interessant genoeg ontdekten we dat de meeste kankergerelateerde signaalwegen eerder gerapporteerd zoals Notch, mTOR en Egel in GC werden geactiveerd op basis van onze gegevens. Deze resultaten ondersteunen het standpunt dat heterogeniteit is het kenmerk van GC. Vergelijking van de co-expressie netwerk tussen normale weefsels en kanker gesuggereerd dat de expressie, functies en interacties van de meeste fysiologische gen verloren of beschadigd maagkanker, dat proliferatie, migratie en metastase abnormaal zijn verhoogd. Deze interessante bevindingen overeenkomen met de kenmerken van kanker, zoals anaplasie en dedifferentiatie. Deze differentieel tot expressie van genen die betrokken zijn bij signaalwegen trad op als belangrijkste genen in co-expressie netwerk zou kunnen zijn van de potentiële doelwitten van anti-kankertherapie of diagnostische markers in de toekomst. Dankwoord We willen graag bedanken Dr. Fred Bogott bij de Austin Medisch Centrum , University of Minnesota, en Dr. Joshua Liao op de Hormel Instituut, Austin van Minnesota, voor hun Engels redactie van dit manuscript. We willen graag de financiers te danken aan dit onderzoek te ondersteunen. Dit werk werd ondersteund door subsidies van de National Natural Science Foundation of China [No. 81172324, nr 91229106, nr 81272749, en nr 81372231], Wetenschap en Technologie Commissie van Shanghai Gemeente [No. 13ZR1425600], en Key Projecten in de National Science & Technology Pillar Program van China (Nr 2014BAI09B03).
-waarde WAS < 0,01 en de vouw change (FC) was > 2. Vervolgens werden Gene Ontology (GO) categorieën gebruikt om de belangrijkste functies van het differentieel tot expressie van genen te analyseren. Volgens het Kyoto-Encyclopedie van Genen en Genomen (KEGG) databank hebben wij trajecten significant geassocieerd met het differentieel genen. Gen-Act netwerk en co-expressie netwerk respectievelijk opgebouwd op basis van de relatie tussen de genen, eiwitten en verbindingen in de database. 2371 mRNAs en 350 lncRNAs beschouwd als significant differentieel tot expressie gebrachte genen werden geselecteerd voor verdere analyse. De GO categorieën, wandelpad analyses en de Gen-Act netwerk liet een consistente gevolg dat up-gereguleerde genen verantwoordelijk waren voor het ontstaan van tumoren, migratie, angiogenese en de vorming van micro-omgeving, terwijl down-gereguleerde genen betrokken waren bij de stofwisseling. De resultaten van dit onderzoek bieden een aantal nieuwe bevindingen over coderende RNAs, lncRNAs, paden en de co-expressie netwerk bij maagkanker die nuttig zal zijn om nader onderzoek te begeleiden en te richten therapie voor deze ziekte
Methods and Materials
weefselmonsters
Limma
-waarden werden aangepast met de BH FDR algoritme. Er waren drie normen voor ons om te oordelen dat een gen significant verschillend tot expressie werd gebracht, de FDR waarde was < 0,01, P
-waarde WAS < 0,01 en de vouw verandering was > 2. (Beschreven in S5 tabel)
2-test werden toegepast op classificeren van de GO-categorie, en de valse ontdekking tarief (FDR,) werd berekend om te corrigeren de P
-waarde ( N
k
verwijst naar het nummer van de toets van Fisher P
-waarden kleiner dan de χ
2-test P
-waarden). De verrijking Re werd gegeven door: Re = ( n
f Twitter / n
) /( N
f Twitter / N
) in de belangrijke categorieën ( N
f
is het aantal differentiële genen in de betreffende categorie, n
is het totale aantal genen in dezelfde categorie, n
f
is het aantal differentiële genen in het gehele microarray, en N
het totale aantal genen in de microarray) (gespecificeerd in tabel S5)
g
is het aantal differentiële genen binnen de bijzondere route, n
een
is het totale aantal genen in dezelfde route, N
g
is de aantal differentiële genen waarvan tenminste één traject annotatie N
een
het aantal genen waarvan tenminste één traject aantekening in de gehele microarray hebben.) (gedetailleerd zijn, en in S5 tabel).
Co-expressie netwerk
Real-time kwantitatieve PCR
Statistische analyse
-waarden lager dan 0,05 werden beschouwd als statistisch significant.
Resultaten
-waarde van 0,017. Dit resultaat was consistent met de gegevens van H19 (Absolute FC = 6,06) in deze microarray analyses. Bovendien overexpressie van H19 bijdraagt aan de proliferatie, migratie, invasie en metastase van maagkanker.
Gene Ontology categories
Gene-Act netwerk
gen co-expressie netwerk
Bevestiging van microarray-resultaten op qPCR
Ondersteunende informatie
S1 Table. Pathway analyse van differentiële genen
doi: 10.1371 /journal.pone.0125013.s001
(XLSX)
S2 Table. GO analyses van differentiële genen
doi: 10.1371 /journal.pone.0125013.s002
(XLSX)
S3 Table. Gene-Act netwerk van differentiële genen
doi: 10.1371 /journal.pone.0125013.s003
(XLSX)
S4 Table. Primers en verificatie
doi: 10.1371 /journal.pone.0125013.s004
(docx)
S5 Table. Methods and Materials
doi: 10.1371 /journal.pone.0125013.s005
(docx)
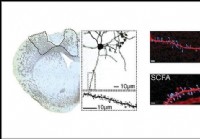 Suppletie met korte keten vetzuren verbetert het herstel van een beroerte,
Suppletie met korte keten vetzuren verbetert het herstel van een beroerte,
 Onderzoekers hopen dat een bloedtest die fibromyalgie nauwkeurig diagnosticeert, binnen vijf jaar beschikbaar kan zijn
Onderzoekers hopen dat een bloedtest die fibromyalgie nauwkeurig diagnosticeert, binnen vijf jaar beschikbaar kan zijn
 Veelvoorkomende genetische variant verklaart waarom immunotherapie vaak faalt bij de ziekte van Crohn
Veelvoorkomende genetische variant verklaart waarom immunotherapie vaak faalt bij de ziekte van Crohn
 Verlies van darmepitheelbarrière die verantwoordelijk is voor COVID-19-gerelateerde MIS-C bij kinderen,
Verlies van darmepitheelbarrière die verantwoordelijk is voor COVID-19-gerelateerde MIS-C bij kinderen,
 Een fysieke en genetische kaart van Cannabis sativa maken
Een fysieke en genetische kaart van Cannabis sativa maken
 Landen met een oudere bevolking hebben hogere SARS-CoV-2-infecties en sterfgevallen,
Landen met een oudere bevolking hebben hogere SARS-CoV-2-infecties en sterfgevallen,
 De eerste bevindingen van het Human Microbiome Project leidden tot 'honderden vervolgonderzoeken'
Het Human Microbiome Project (HMP) is een initiatief ontwikkeld door de National Institutes of Health om het menselijke microbioom te karakteriseren bij zowel gezonde volwassenen als bij mensen met sp
De eerste bevindingen van het Human Microbiome Project leidden tot 'honderden vervolgonderzoeken'
Het Human Microbiome Project (HMP) is een initiatief ontwikkeld door de National Institutes of Health om het menselijke microbioom te karakteriseren bij zowel gezonde volwassenen als bij mensen met sp
 Risico op tandvleesaandoeningen en slokdarm- en maagkanker
Amerikaanse onderzoekers hebben hun nieuwe bevindingen over tandvleesaandoeningen gepubliceerd in een onderzoeksbrief in het laatste nummer van het tijdschrift Darm getiteld, Tandvleesziekte, tandve
Risico op tandvleesaandoeningen en slokdarm- en maagkanker
Amerikaanse onderzoekers hebben hun nieuwe bevindingen over tandvleesaandoeningen gepubliceerd in een onderzoeksbrief in het laatste nummer van het tijdschrift Darm getiteld, Tandvleesziekte, tandve
 Onderzoekers identificeren in vitro een bacterie met anti-SARS-CoV-2-activiteit:Dolosigranulum pigrum
Het ernstige acute respiratoire syndroom coronavirus 2 (SARS-CoV-2) heeft wereldwijd meer dan 173,3 miljoen mensen besmet. Van deze, een significante minderheid is ernstig of kritisch geweest, leidend
Onderzoekers identificeren in vitro een bacterie met anti-SARS-CoV-2-activiteit:Dolosigranulum pigrum
Het ernstige acute respiratoire syndroom coronavirus 2 (SARS-CoV-2) heeft wereldwijd meer dan 173,3 miljoen mensen besmet. Van deze, een significante minderheid is ernstig of kritisch geweest, leidend