Hintergrund
Magenkrebs ist weiterhin einer der tödlichsten Krebsarten in der Welt zu sein und damit Identifizierung neuer Medikamente dieser Art von Krebs-Targeting ist daher von großer Bedeutung. Der Zweck dieser Studie ein therapeutisches Mittel zur Identifizierung und Validierung war, die die Ergebnisse für Patienten mit Magenkrebs in der Zukunft verbessern könnte.
Microarray-Technologie, erzielten wir ein Gen Expressionsprofil von menschlichen Magenkrebs-spezifische Gene aus humanen Proben Magenkrebsgewebe. Wir haben dieses Profil in der Connectivity Map Analyse des Broad Institute Kandidat therapeutischen Verbindungen für Magenkrebs zu identifizieren. Wir fanden das Histondeacetylaseinhibitors Vorinostat als Leitsubstanz und damit ein potenzielles therapeutisches Arzneimittel für Magenkrebs. Vorinostat sowohl Apoptose und Autophagie bei Magenkrebs-Zelllinien induziert. jedoch pharmakologischen und genetischen Hemmung der autophagy, erhöht die therapeutische Wirksamkeit von vorinostat, was anzeigt, dass eine Kombination von vorinostat mit autophagy Inhibitoren therapeutisch vorteilhafter sein kann. Darüber hinaus identifiziert die Genexpressionsanalyse von Magenkrebs eine Sammlung von Genen ( ITGB5, TYMS, MYB, APOC1, CBX5, PLA2G2A, Poster und KIF20A Wir haben gezeigt, dass die Analyse von Genexpressionssignatur kann ein aufstrebendes Ansatz darstellen, um therapeutische Mittel für Magenkrebs, wie Vorinostat entdecken. Die Beobachtung der veränderten Genexpression nach vorinostat Behandlung kann den Schlüssel zur Verfügung stellen, den molekularen Mechanismus von Vorinostat und die Patienten würden profitieren von Vorinostat Behandlung Citation zu identifizieren. Claerhout S, Lim JY, Choi W, Park YY, Kim K, Kim SB, et al. (2011) Gene Expression Signaturanalyse Identifiziert Vorinostat als Kandidat Therapie für Magenkrebs. PLoS ONE 6 (9): e24662. doi: 10.1371 /journal.pone.0024662 Herausgeber: David L. McCormick, IIT Research Institute, Vereinigte Staaten von Amerika Empfangen: 29. März 2011; Akzeptiert: 16. August 2011; Veröffentlicht: 9. September 2011 © 2011 Claerhout et al. Dies ist eine Open-Access-Artikel unter den Bedingungen der Lizenz Creative Commons, die uneingeschränkte Nutzung erlaubt, die Verteilung und Vervielfältigung in jedem Medium, vorausgesetzt, der ursprüngliche Autor und Quelle genannt werden Finanzierung:. Die Finanzierung zu SC als Odyssey Fellow wurde von der Odyssey-Programm und dem Theodore N. Law Stiftung für wissenschaftliche Leistungen an der University of Texas MD Anderson Cancer Center unterstützt. Diese Arbeit wurde auch durch Mittel aus dem 2009 Innere Medizin Academic Research Fund und Basic Science Research Program durch die National Research Foundation of Korea gefördert durch das Ministerium für Bildung, Wissenschaft und Technologie (Nr 2010-0.024.248) unterstützt. Die Geldgeber hatten keine Rolle in Studiendesign, Datenerfassung und Analyse, Entscheidung oder Vorbereitung des Manuskripts zur Veröffentlichung Konkurrierende Interessen:.. Die Autoren haben erklärt, dass keine Interessenkonflikte bestehen Einführung Magenkrebs ist die vierthäufigste Krebserkrankung und die zweithäufigste Ursache für Krebstod in der Welt [1], mit einem Gesamtüberlebenszeit von etwa 10 Monate [2] - [4]. Die Behandlung von Magenkrebs kann eine Chemotherapie, Operation und Strahlentherapie umfassen. Leider aktuellen Chemotherapie-basierten Behandlungen für fortgeschrittenem Magenkrebs zeigen enttäuschende Ergebnisse [2] - [4]. Tatsächlich sind komplette Remissionen selten oder nur sehr kurz dauern. Mehrere gezielte Mittel, die das Überleben Vorteile bei anderen Krebsarten verleihen haben bei Magenkrebs untersuchten gewesen. Während einige frühe klinische Studien vaskulären endothelialen Wachstumsfaktor-Rezeptor (VEGFR) und epithelialen Wachstumsfaktor-Rezeptor (EGFR) -1-Inhibitoren, wie Cetuximab und Bevacizumab verwenden, haben etwas Aktivität gezeigt, gibt selten eine tatsächliche Überlebensvorteil für den Patienten ist [5] [6]. Einer der Gründe kann sein, dass diese Studien nicht die Patienten nach dem Vorhandensein von Biomarkern ausgewählt haben. Vor kurzem stellte die Trastuzumab für Magenkrebs (ToGA) -Studie, dass die Zugabe von Trastuzumab zur Chemotherapie zu einer statistisch signifikanten Verbesserung des progressionsfreien Überlebens (PFS) geführt und das Gesamtüberleben (OS) der etwa 20% der Patienten mit metastasierenden Magen- und gastroösophagealen (GE) Kreuzung Tumoren überexprimiert, HER2 [7]. Dies unterstreicht die Notwendigkeit einer gezielten biologischen Therapie und die Suche nach Biomarkern Patienten für klinische Studien zu wählen, die das Überleben profitieren können. Trotz einiger Hinweise auf potentielle Ziele, einschließlich HER2 [8], [9], die Wirksamkeit dieser biologisch zielgerichtete Therapien ist nicht bekannt, und es besteht ein Mangel an einer Standard-zielgerichtete Therapie für Magenkrebs. Aufgrund der biologischen Heterogenität von Magenkrebs, ist es unwahrscheinlich, dass es eine einzige "Wundermittel" Heilung. Molekulare Marker werden in Zukunft so wichtig sein, Patienten "Ergebnisse und Schneiderei Behandlungen nach individuellen Biologie zu prognostizieren. Auf der Suche nach Biomarkern, die Genexpression Signaturanalyse hat in verschiedenen Anwendungen, wie zum Beispiel für die Aufklärung der verwendet worden Mechanismen der biologischen Wege [10], Subtypen einer Krankheit [11], die Vorhersage Krebsprognose [12] und Profilierungs Genexpression in Reaktion auf bestimmte Medikamente [13], [14] zu klassifizieren. Die Genexpression Signaturanalyse kann unter Verwendung des Broad Institute Connectivity Map (http://www.broadinstitute.org/cmap) erfolgen. Die Connectivity Map zielt darauf ab, eine Karte zu generieren, die von Arzneimittelkandidaten und genetische Manipulationen erzeugten Muster der Genexpression im Zusammenhang mit der Krankheit zu entsprechenden Muster verknüpft [15], [16]. Dieser Systemansatz ermöglicht Verbindungen gegen genomweite Krankheit Unterschriften aussortiert zu werden, anstatt einer vorgewählten Satz von Zielgenen. Die Medikamente werden mit Krankheiten mit ausgefeilten Pattern-Matching-Verfahren mit einem hohen Grad an Auflösung und Spezifität gepaart. Obwohl es viele offene Fragen hinterlässt, hat die Connectivity Map dass genomische Signaturanalyse gezeigt, mit gemeinsamen Wirkungsmechanismen Drogen zu erkennen, verwendet werden, entdecken unbekannte Wirkmechanismen und mögliche neue Therapeutika [15], [16]. der Zweck dieser Studie war es potentielle neue Therapeutika für die Behandlung von Magenkrebs zu identifizieren. Dazu analysierten wir zunächst die genomische Unterschrift des menschlichen Magenkrebs. Die resultierende Signatur wurde Magen-Krebs-Gen dann verwendet, in silico und Videos Connectivity Map-Analyse unter Verwendung therapeutischer Wirkstoffe zu identifizieren, die potentiell wirksam gegen diese Art von Krebs sein könnte. Wir validiert weiterhin die Top-Medikament für seine Wirksamkeit bei Magenkrebs-Zelllinien-Targeting. Wir fanden, dass Vorinostat, als ein potenzielles neues Medikament, induzierte sowohl Apoptose und Autophagie in Magenkrebszellen. Zusammen zeigt diese Studie, dass die Anschlusskartenanalyse kann zur Identifizierung von therapeutischen Mitteln verwendet werden, die in der Behandlung einer Untergruppe von Magenkarzinome erfolgreich sein kann. Analyse von Microarray-Daten Für die Connectivity Map Analyse verwendeten wir die Microarray-Daten von 65 Patienten mit Magenkrebs, darunter 65 Krebserkrankungen und 19 normalen Magen-Gewebe, die aus unserer bisherigen Arbeit, Yonsei Daten [17] erhalten wurden. Tumorproben wurden von Patienten mit Magenkrebs Gastrektomie als primäre Behandlung gesammelt. Gewebeproben wurden durch Pathologen bei der Sammlung und gespeichert in -80 ° C an der Gewebebank bis zum Beginn des Experiments untersucht. Gesamt-RNA wurde aus den frischen gefroren Gewebe extrahiert durch eine Mirvana RNA-Isolierung Kennzeichnung Kit (Ambion, Inc.). Primäre Microarray-Daten ist in NCBI Gene Expression Omnibus öffentliche Datenbank (Microarray-Plattform, GPL6884; Microarray-Daten, GSE 13861) zur Verfügung. Ein weiteres Genexpressionsprofil wurde von 69 Magen-Gewebeproben erhalten, darunter 38 Krebs und 31 nicht Krebs Stroma, der Stanford Microarray-Datenbank (http://smd.stanford.edu, GSE13911), Stanford-Daten. Magen-Krebs-spezifische Gene wurden von BRB-ArrayTools Version 3.6.1 (Biometric Research Branch, National Cancer Institute, Bethesda, MD) ausgewählt. Klasse Vergleich mit zwei Stichproben-t-Test (Signifikanz < 0,001, 10.000 zufällige Permutation), deren mittlere Magenkrebs spezifische Gene und Gene identifiziert Expressionsintensitäten wurden geändert durch mindestens zweifach verglichen Ausdruck normalem Gewebe Gen verstanden wurden ausgewählt Connectivity Map Analyse Um potenzielle Medikamente Targeting Magenkrebs, die Gen-Listen der Top-500 bis regulierbar und die Top-500 nach unten regulierte Gene aus den Magen-Krebs-spezifische Gene verwendet wurden (Tabelle S1) zu identifizieren. Die Connectivity Map-Analyse wurde über die Web-Schnittstelle durchgeführt (http://www.broadinstitute.org/cmap) Version, 02 bauen, die mehr als 7000 enthält Expressionsprofile Effekte von 1.309 Verbindungen auf mehreren kultivierten menschlichen Zellen darstellen [15], [16]. Die Connectivity Karte zeigt funktionelle Verbindungen zwischen Arzneimitteln, Genen und Krankheiten. Medikamente, die krankheits nachahmt Gensignaturen produzieren kann Wege identifizieren, die potenzielle therapeutische Ziele für diese Krankheit darstellen. Umgekehrt, Medikamente, die entgegengesetzt zu derjenigen in dem Krankheitszustand beobachtet einen "umgekehrten" Signatur, d.h. Veränderungen in der Genexpression in einer Richtung, induzieren könnten neue therapeutische Mittel darstellen. Kandidatenmittel gegen eine bestimmte Krankheit durch Anwendung krankheitsspezifische Genexpressionsprofil dem Connectivity Map Analyse erkannt werden. Wir ausgewählten Kandidaten Medikamente für die Validierung In-vitro- Vorinostat wurde von Merck erhalten und als Stammlösung in Dimethylsulfoxid (DMSO) hergestellt. Chloroquin und Bafilomycin A1 (Sigma) wurden in jeweils Wasser und DMSO gelöst. Menschliche Zelllinien Magenkrebs AGS, NCI-N87 und KATO-III wurden von der American Type Culture Collection erhalten und wurden nach ihren Empfehlungen gehalten. Zellen wurden in RPMI 1640, ergänzt mit 10% fötalem Rinderserum, 100 U Penicillin und 100 ug /ml Streptomycin bei 37 ° C in 5% CO 2. Für das 3- (4,5-Dimethylthiazol-2-yl) -2,5-diphenyl Tetrazolium Bromid (MTT) -Assay, MTT wurde in PBS mit 5 mg /ml gelöst. Etwa 5 × 10 3 Zellen in 96-Well-Platten ausgesät und über Nacht anheften gelassen. Das Kulturmedium wurde dann mit frischem Medium, enthaltend die angegebenen Konzentrationen von vorinostat oder DMSO ersetzt. Nach 72 h wurden 20 &mgr; l MTT-Lösung zugegeben, und die Platten wurden bei 37 ° C für 4 h inkubiert. Nach der Inkubation wurden 100 &mgr; l DMSO zugegeben, um das Formazan zu lösen, und die Extinktion wurde bei 570 nm unter Verwendung eines spektrophotometrischen Mikrotiterplatten-Lesegerät (Vmax kinetischen Mikroplattenlesegerät, Molecular Devices) abgelesen. Die Versuche wurden dreifach durchgeführt. Kristallviolettfärbung, Zellen für 12 h behandelt wurden, wurde das Medium entfernt und die Zellen wurden mit PBS gewaschen und dann für 30 min mit 0,5% Kristallviolett inkubiert (in 20% Methanol und 80% doppelt destilliertem Wasser). Die Zellen wurden dann dreimal mit PBS gewaschen. Das verbleibende Kristallviolett wurde 5 min in Essigsäure extrahiert, und die Extinktion wurde bei 595 nm gemessen, um eine spektrophotometrische Mikrotiterplatten-Lesegerät verwenden. die Lebensfähigkeit der Zellen zu bestimmen, wurden die Zellen für 72 h mit vorinostat inkubiert. Adhärente Zellen wurden dann aus Kulturplatten durch Trypsinisierung und in Kombination mit schwimmenden Zellen abgelöst, abzentrifugiert und in 500 ul Propidiumiodid (PI) -exclusive Lösung suspendiert, für 15 min bei 4 ° C (nicht Membran eindringende Zelle). Gefärbte Zellen wurden mittels Durchflusszytometrie überwacht (Beckman Coulter Cytomics FC 500). Für die Zellzyklusanalyse wurden die Zellen inkubiert mit vorinostat für 24 h gesammelt und in 500 &mgr; l der hypotonischen Lösung (0,1% Natriumcitrat suspendiert, 0,1% Triton X-100, 100 ug /ml RNase und 50 &mgr; g /ml PI) für 15 min bei 4 ° C. PI-gefärbten Zellen wurden mittels Durchflusszytometrie überwacht. Zellzyklus wurde durch Multicycle AV-Software analysiert. RNA Isolation und Microarray-Experimente wurden durchgeführt nach dem Protokoll, wie zuvor beschrieben [17]. Gesamt-RNA wurde aus Magenkrebs-Zelllinien, mit oder ohne vorinostat Behandlung extrahiert eine Mirvana RNA Isolation Kit (Ambion, TX, USA). Die Integrität der großen RNA-Fraktion wurde mit einem Experion Bioanalyzer (Bio-Rad, CA, USA) als Surrogat für mRNA Qualitätskontrolle bestimmt. Gesamt-RNA wurde gemäß den Herstellerprotokollen (Illumina, CA, USA) mit humanen HT12 v.3 Ausdruck BeadChips markiert und hybridisiert. Nachdem die BeadChips mit einem Illumina BeadArray Reader gescannt wurden, wurden die Microarray-Daten normalisiert mit dem Quantil Normierungsverfahren in den Linear Models für Microarray-Daten-Paket in der R Sprachumgebung [18]. Das Expressionsniveau von jedem Gen wurde in ein Protokoll 2 Base vor der weiteren Analyse transformiert. Die Clusteranalyse wurde mit Cluster durchgeführt und Treeview [19]. Die Zellen wurden in Medium und abzentrifugiert geschabt und Proteine wurden mit Lysepuffer isoliert (50 mM HEPES, 150 mM NaCl, 1 mM EGTA und 10 mM Natriumpyrophosphat (pH 7,4)), enthaltend 100 mM NaF, 10% Glycerin, 1,5 mM MgCl 2, 1% Triton X-100 und Protease-Inhibitor (Roche). Extrakte wurden für 20 min auf Eis inkubiert und bei 20800 g für 20 min abzentrifugiert. Die Proteinkonzentration wurde unter Verwendung von BCA-Protein-Assay-Reagenz (Pierce) bestimmt. Gleiche Mengen von Protein aus jeder Probe wurden durch Elektrophorese durch SDS-PAGE getrennt und auf Hybond-C Super-Membran (Amersham Pharmacia Biotech). Membranen wurden für 1 h bei Raumtemperatur in Tris-gepufferter Salzlösung, enthaltend 0,1% Tween-20 und 5% fettfreier Trockenmilch blockiert. Die Membranen wurden über Nacht bei 4 ° C mit dem primären Antikörper, verdünnt in 5% fettfreier Trockenmilch oder 5% BSA in 1 × Tris-gepufferter Kochsalzlösung plus 0,1% Tween-20. Antikörper gegen LC3 (Novus Biologicals), aktive Caspase-3 (Epitomics) und p62 (BD Biosciences) verwendet wurden. Antikörper gegen alpha-Tubulin und Beclin-1 waren von Cell Signaling Technology; Antikörper gegen &bgr; -Actin wurde von Sigma. Membranen wurden dann für 1 h bei Raumtemperatur mit Peroxidase-konjugierten sekundären Antikörper (Cell Signaling Technology) gewaschen und inkubiert. Die Proteinbanden wurden mittels verstärkter Chemilumineszenz sichtbar gemacht, wie vom Hersteller beschrieben (GE Healthcare). Die Ziel siRNA-Sequenz für Beclin-1, nontargeting siRNA (Risc Free) und Dharmafect 1 waren von Dharmacon gekauft. Die Zellen wurden in 10-cm-Schalen und transfiziert mit siRNA 24 h später ausgesät gemäß dem Protokoll des Herstellers. Am nächsten Tag wurden die Zellen mit Trypsin behandelt und ausgesät in 6-cm oder 96-Well-Platten die gleiche Transfektionseffizienz zu erhalten. Protein-Expressionsniveaus wurden durch Western-Blot-Analyse bestimmt. Die Proben wurden mit einer Lösung fixiert, die 3% Glutaraldehyd plus 2% Paraformaldehyd in 0,1 M Cacodylatpuffer, pH 7,3, für 1 Stunde. Nach der Fixierung wurden die Proben gewaschen und mit 0,1% Millipore-filtrierte Cacodylat gepuffertem Gerbsäure behandelt, nachfixiert und mit 1% gepuffertem Osmiumtetroxid 30 min und en bloc mit 1% Millipore-filtrierte Uranylacetat gefärbt. Die Proben wurden dehydriert in Konzentrationen von Ethanol, infiltriert und eingebettet in LX-112 Medium zu erhöhen. Die Proben wurden in einem Ofen 70 ° C polymerisiert, für 2 Tage. Ultradünne Schnitte wurden in einem Leica Ultracut Mikrotom geschnitten (Leica, Deerfield, IL), gefärbt mit Uranylacetat und Bleizitrat in einem Leica EM Stainer und untersuchte in einem JEM 1010 Transmissions-Elektronenmikroskop (JEOL, USA, Inc., Peabody, MA ) bei einer Beschleunigungsspannung von 80 kV. Digitale Bilder wurden mit AMT Imaging System (Advanced Mikroskopietechniken Corp, Danvers, MA) erhalten.
), deren Expression in Magentumorgewebe erhöht und mehr als 2-fach durch vorinostat Behandlung bei Magenkrebs-Zelllinien nach unten reguliert. Im Gegensatz dazu SCGB2A1, TCN1, CFD, APLP1, Poster und NQO1
ein umgekehrtes Muster manifestiert.
Schlussfolgerungen /Bedeutung
Methods
.
auf der Grundlage der Connectivity-Score, Korrelation und P-Wert.
Chemikalien und Zellkultur
Zellwachstum, Lebensfähigkeit und Zellzyklus Assays
RNA Isolation und Microarray-Experimenten
Western-Blot-Analyse
siRNA-Transfektion
Transmissionselektronenmikroskopie
Ergebnisse
 Antibiotikum X-206 wirksam gegen SARS-CoV-2 in vitro
Antibiotikum X-206 wirksam gegen SARS-CoV-2 in vitro
 Milchsäurebakterien und Darmbakterien tragen zu den gesundheitlichen Vorteilen von Roggen bei,
Milchsäurebakterien und Darmbakterien tragen zu den gesundheitlichen Vorteilen von Roggen bei,
 Thiopurine könnten helfen, die Virusreplikation bei menschlichen Coronaviren zu stoppen
Thiopurine könnten helfen, die Virusreplikation bei menschlichen Coronaviren zu stoppen
 Bakteriophagen könnten E. coli behandeln, ohne den Darm zu schädigen.
Bakteriophagen könnten E. coli behandeln, ohne den Darm zu schädigen.
 Infliximab kann die Wirksamkeit einiger COVID-19-Impfstoffe beeinträchtigen
Infliximab kann die Wirksamkeit einiger COVID-19-Impfstoffe beeinträchtigen
 Was Sie essen, kann die Wirkung von Antibiotika auf Ihren Darm verändern
Was Sie essen, kann die Wirkung von Antibiotika auf Ihren Darm verändern
 Studie zeigt antivirale Wirkung von Curcumin
Kurkumin, ein natürlicher Bestandteil des Gewürzes Kurkuma, könnte helfen, bestimmte Viren zu eliminieren, Forschung gefunden hat. Eine im veröffentlichte Studie Zeitschrift für Allgemeine Virologi
Studie zeigt antivirale Wirkung von Curcumin
Kurkumin, ein natürlicher Bestandteil des Gewürzes Kurkuma, könnte helfen, bestimmte Viren zu eliminieren, Forschung gefunden hat. Eine im veröffentlichte Studie Zeitschrift für Allgemeine Virologi
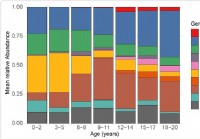 Veränderung des Mikrobioms der oberen Atemwege bei Kindern im Zusammenhang mit der Anfälligkeit für SARS-CoV-2
Die Forscher testeten das nasopharyngeale Mikrobiom von Kindern bis zu 21 Jahren und fanden heraus, dass sich das Mikrobiom mit dem Alter verändert. Spezifische Bakterien, deren Fülle sich auch mit de
Veränderung des Mikrobioms der oberen Atemwege bei Kindern im Zusammenhang mit der Anfälligkeit für SARS-CoV-2
Die Forscher testeten das nasopharyngeale Mikrobiom von Kindern bis zu 21 Jahren und fanden heraus, dass sich das Mikrobiom mit dem Alter verändert. Spezifische Bakterien, deren Fülle sich auch mit de
 Fast Food kann ein Hauptgrund für Depressionen im Teenageralter sein
Warum ist Depression unter Teenagern in Amerika ein so wachsendes Problem? Eine Antwort ist die Art der Nahrung, die sie essen, Das geht aus einer neuen Studie von Forschern der University of Alabama
Fast Food kann ein Hauptgrund für Depressionen im Teenageralter sein
Warum ist Depression unter Teenagern in Amerika ein so wachsendes Problem? Eine Antwort ist die Art der Nahrung, die sie essen, Das geht aus einer neuen Studie von Forschern der University of Alabama