Résumé
Chitinases hydrolyser les β-1-4 liaisons glycosidiques de chitine, un importante composante structurelle de champignons, de crustacés et d'insectes. Bien que les mammifères ne produisent pas de chitine ou de son synthase, ils expriment deux chitinases actifs, chitotriosidase (Chit1) et chitinase mammifère acide (AMCase). Ces chitinases de mammifères ont attiré une attention considérable en raison de leur expression accrue chez des individus ayant un certain nombre d'états pathologiques, y compris la maladie de Gaucher, la maladie d'Alzheimer et l'asthme. Toutefois, la contribution de ces enzymes dans la physiopathologie de ces maladies reste à déterminer. La quantification des niveaux Chit1 et AMCase ARNm et la comparaison de ces niveaux avec les niveaux de gènes de référence bien connus peuvent générer des informations utiles et pertinentes biomédical. Au début, nous avons établi un système de PCR quantitative en temps réel, qui utilise l'ADN standard produit par ligature des fragments d'ADNc des gènes cibles. Ce système nous a permis de quantifier et de comparer les niveaux d'expression des chitinases et les gènes de référence sur la même échelle. Nous avons constaté que AMCase ARNm est synthétisé à des niveaux extraordinairement élevés dans l'estomac de la souris. Le niveau de cet ARNm dans l'estomac de la souris était de 7 à 10 fois plus élevé que les niveaux des gènes d'entretien et était comparable à celle du niveau de l'ARNm pour le pepsinogène C (progastricsin), un composant majeur de la muqueuse gastrique. Ainsi, AMCase ARNm est un transcrit majeur dans l'estomac de la souris, ce qui suggère que les fonctions de Amcase comme une enzyme digestive qui décompose la chitine polymère et dans le cadre de la défense de l'hôte contre les agents pathogènes contenant de la chitine dans le contenu gastrique. Notre méthodologie est applicable à la quantification des ARNm pour plusieurs gènes sur plusieurs spécimens en utilisant la même échelle
Citation:. Ohno M, Tsuda K, Sakaguchi M, Sugahara Y, Oyama F (2012) chitinase niveaux d'ARNm par Quantitative PCR en utilisant l'ADN simple standard: Acidic Mammalian chitinase est un major Transcript dans le ventre de la souris. PLoS One 7 (11): e50381. doi: 10.1371 /journal.pone.0050381
Editeur: Dominik Hartl, Université de Tübingen, Allemagne
Reçu: 6 Août 2012; Accepté le 19 Octobre 2012; Publié: 21 Novembre 2012 |
Droit d'auteur: © 2012 Ohno et al. Ceci est un article en accès libre distribué sous les termes de la licence Creative Commons Attribution, qui permet une utilisation sans restriction, la distribution et la reproduction sur tout support, à condition que l'auteur et la source originelle sont crédités
Financement:. Ce travail a été soutenue par la subvention du projet de recherche de l'Institut de recherche en sciences et technologie, Université Kogakuin. Les bailleurs de fonds ont joué aucun rôle dans la conception de l'étude, la collecte et l'analyse des données, la décision de publier, ou de la préparation du manuscrit
Intérêts concurrents:.. Les auteurs ont déclaré aucun conflit d'intérêts existent
Introduction
chitine, un polymère linéaire de β-1-4-liés N Le niveau Chit1 est nettement élevée dans le plasma des patients atteints de la maladie de Gaucher, un trouble récessif du stockage lysosomal autosomique [4]. Chit1 a été le premier mammifère chitinase à purifier et clone [5], [6]. Une carence héréditaire récessive de l'activité Chit1 est couramment observée chez les Caucasiens [7]. AMCase a été découvert en raison de son rôle compensatoire et a été nommé pour son pH acide optimal [8]. Ces chitinases de mammifères sont considérées comme faisant partie du mécanisme de défense de l'hôte contre les agents pathogènes et les parasites contenant de la chitine [3], les protéines [9]. Les deux Chit1 et AMCase sont sécrétées avec des poids moléculaires d'environ 50 kDa. Les deux protéines contiennent un domaine catalytique N-terminal, une région charnière et un domaine de liaison à la chitine C-terminale [6], [8]. AMCase souris montre une homologie de séquence Chit1, avec une identité de 52% et une similarité de 60% [10]. En dépit de ces similitudes structurales, ces enzymes diffèrent de manière significative par rapport à leur comportement enzymatique à un pH acide. AMCase montre un optimum de pH marquée à pH 2 et un optimum moins évident à pH 4-7 [8], alors que Chit1 ne montre qu'une large optimum de pH à un pH d'environ 5 [5], [11]. Mammifères chitinases ont attiré une attention considérable en raison de leur expression accrue chez les personnes ayant des conditions pathologiques. Chit1 est augmentée chez les sujets atteints de la maladie de Gaucher [4], la broncho-pneumopathie chronique obstructive (BPCO) [12] et la maladie d'Alzheimer [13] et chez les fumeurs [14]. l'expression et l'activité AMCase sont également régulés à la hausse au cours des réponses des voies respiratoires allergiques chez les modèles murins d'asthme [15]. En outre, la chitine polymère induit l'expression de AMCase et le recrutement de cellules immunitaires associées à l'allergie et l'asthme [16]. Ces résultats suggèrent fortement que les enzymes chitinolytiques jouent un rôle important dans de nombreuses conditions physiopathologiques. Toutefois, la contribution de ces enzymes à la pathophysiologie de ces maladies reste à déterminer. La quantification des niveaux Chit1 et AMCase ARNm et la comparaison de ces niveaux avec les niveaux de gènes de référence bien connus sont importants étapes de gagner un aperçu de la in vivo Dans la présente enquête, nous avons développé un système RT-PCR quantitative en temps réel qui utilise l'ADN standard produit en ligaturant le des fragments d'ADNc des gènes cibles. Ce système nous a permis de quantifier et de comparer les niveaux d'expression des chitinases et les gènes de référence sur la même échelle. Nos résultats indiquent que AMCase est un transcrit majeur dans l'estomac de la souris, ce qui suggère que les fonctions des protéines correspondantes comme une enzyme digestive qui décompose les aliments contenant de la chitine et dans le cadre de la défense de l'hôte contre les agents pathogènes contenant de la chitine dans le contenu gastrique. Matériaux et ARN, ARN Isolation de méthodes et d'ADNc Préparation la souris ARN total master Panel (Clontech Laboratories) a été utilisé pour examiner la distribution tissulaire des transcrits. Nous avons analysé quatre stades embryonnaires différents et huit tissus adultes. En outre, l'ARN a été isolé à partir des poumons et des estomacs des souris mâles de 3 mois. C57BL /6J (EFFACER Japon) ont été élevés à l'installation de RIKEN Brain Science Institute animaux. Toutes les expériences sur les animaux ont été effectuées en conformité avec les directives institutionnelles. Le protocole a été approuvé par le Comité sur l'éthique de l'expérimentation animale de l'Institut des sciences du cerveau RIKEN (Approbation n ° H19-2B013). Toute opération chirurgicale a été réalisée en utilisant de l'éther diéthylique comme anesthésique, et tous les efforts ont été faits pour minimiser la souffrance. Ces tissus pour la préparation d'ARNm ont été fournis par les Drs. Miyazaki et Nukina à RIKEN Brain Science Institute. L'ARN total a été préparé à partir de poumons et l'estomac à l'aide du réactif Trizol (Invitrogen) selon les instructions du fabricant. Pour éliminer les traces d'ADN génomique contaminant les échantillons d'ARN totaux ont été traités avec DNase RQ1 Rnase-Free (Promega) selon le protocole recommandé par le fabricant. Les concentrations des acides nucléiques ont été déterminées en mesurant l'absorbance à 260 nm en utilisant un BioPhotometer Plus (Eppendorf). Chacun des échantillons d'ARN total (3 pg) a été soumis à une transcription inverse avec des hexamères aléatoires comme amorces. Le mélange réactionnel (15 ul) contenait le tampon d'enzyme [50 mM de Tris-HCl (pH 8,3), 75 mM de KCl, et mM MgCl 3 2], 100 ng d'hexamères aléatoires, dithiothréitol 10 mM, et 0,5 mM de désoxynucléotide triphosphates (dNTP). Après avoir chauffé la solution à 60 ° C pendant 5 min et l'incubation du mélange à 37 ° C pendant 5 min, 200 U de virus de la leucémie murine recombinante transcriptase inverse (Invitrogen) a été ajouté et le mélange a été mis en incubation à 37 ° C pendant 45 min . La transcription inverse a été arrêtée par chauffage à 95 ° C pendant 5 min. Amorces pour en temps réel RT-PCR ont été conçus sur la base Primer Software Express (Applied Biosystems) et ont été synthétisés dans le commerce (Sigma-Genosys, Sigma-Aldrich). Les réactions PCR ont été réalisées dans un volume final de 13 pi contenant 2 × SYBR Green Master Mix (Brilliant II SYBR Green QPCR Master Mix, Agilent), 2,7 ng d'ADNc de souris ou des dilutions appropriées des étalons externes (voir ci-dessous), et le les concentrations appropriées des amorces pour l'chitinases, le pepsinogène C, la glycéraldéhyde-3-phosphate déshydrogénase (GAPDH) ou β-actine. Les conditions de PCR en temps réel standard pour le système (Mx3005P, Agilent) ont été utilisées: une étape initiale de dénaturation et de la polymerase d'activation pendant 10 min à 95 ° C, suivie de 40 cycles de dénaturation à 95 ° C pendant 1 min, 55 ° C pendant 30 s et 72 ° C pendant 1 min. courbes de fusion ont été générés après amplification. Les produits de PCR ont été soumis à une électrophorèse sur un gel à 10% de Polyacrylamide et analysées en utilisant l'analyseur d'image luminescents (ImageQuant LAS 4000, GE Healthcare). La solution PCR a été traité avec ExoSAP-IT (USB Produits) selon les instructions du fabricant pour éliminer les amorces et les dNTP non constituées en société, et les produits ont été séquences en utilisant le kit de séquençage de cycle v3.1 ABI PRISM Big-Dye Terminator et un analyseur génétique 3130 ( Applied Biosystems). Les séquences nucléotidiques des amorces choisies pour la PCR en temps réel sont présentés dans le tableau S1. L'ADNc de modèle standard (913 bases) pour la quantification des niveaux de transcription par PCR en temps réel a été construit comme suit. Les fragments d'ADNc couvrant la région PCR cible plus 9-120 bases des régions adjacentes de AMCase, Chit1, le pepsinogène C, GAPDH, et β-actine ont été amplifiés à partir d'ADNc d'estomac de souris par PCR en utilisant KOD ADN plus polymerase (Toyobo) et des oligonucléotides amorce contenant les sites de Bgl Pour valider notre méthode absolue PCR en temps réel, nous a préparé plein codant des ADNc par PCR en utilisant les jeux d'amorces énumérées dans le tableau S3. Cinq ADNc couvrant l'ensemble des régions codantes de deux chitinases (Chit1 et AMCase) et les gènes de référence (GAPDH, bêta-actine et de pepsinogène C) ont été amplifiés par PCR à partir d'ADNc d'estomac de souris en utilisant KOD ADN plus polymerase et ont été sous-clonés dans pGEM-T vecteur facile par clonage TA, tel que décrit ci-dessus. Les séquences des ADNc ont été vérifiées par séquençage, comme décrit dans la figure S1. Les fragments sous-clonés ont été réamplifiés à partir des ADN plasmidiques avec les mêmes amorces et ont ensuite été utilisés comme l'ensemble de la région codante d'ADNc. La concentration molaire de l'étalon d'ADN contenant des gènes multiples, on a calculé en fonction de la concentration et du poids moléculaire. Des dilutions en série ont été préparées à partir de la concentration de matrice standard, ce qui a abouti à un Ct d'environ 13 (Ct = valeur du cycle de seuil fractionnaire). L'ADN standard a été soumis à 10 fois des dilutions en série, allant de 10 0 à 10 7 molécules, et les aliquotes ont été maintenus congelés à -20 ° C jusqu'à utilisation. chaque échantillon a été amplifié en trois exemplaires, et chaque expérience a été répétée au moins deux fois. En utilisant la courbe standard, le nombre de Chit1, AMCase, GAPDH, β-actine et pepsinogène molécules d'ARNm C ont été extrapolés automatiquement en utilisant la version MxPro QPCR Software 4.10 (Agilent). Toutes les valeurs ont été exprimées en molécules par 10 ng d'ARN total. Dans certains cas, les valeurs ont été normalisées par rapport aux niveaux de GAPDH ou β-actine. Pour évaluer notre méthodologie actuelle, nous avons également eu recours à la méthode ΔΔ Ct [21], ce qui nécessite la mesure du seuil de cycle (Ct) de gènes cibles. Les valeurs de Ct ont été calculées par le logiciel QPCR MxPro utilisant GAPDH ou β-actine en tant que normalisateur. Résultats de création de la PCR en temps réel Assay La mise en place d'un méthode fiable pour la mesure de l'expression des transcrits chitinase est une étape importante dans les enquêtes sur l'importance des chitinases de souris. En temps réel RT-PCR est, à l'heure actuelle, la méthode quantitative la plus sensible pour la détection des deux ARNm de faible abondance et relevés de notes abondantes. Nous avons d'abord comparé les niveaux des gènes Chit1 et Amcase expression génique (voir Figure 1). Pour évaluer les niveaux de chitinase, nous avons utilisé deux gènes de ménage, GAPDH et β-actine, comme des gènes de référence parce qu'ils sont constitutivement exprimés à des niveaux élevés dans la plupart des tissus et des cellules [22]. En outre, nous avons choisi le pepsinogène C (également connu sous le nom progastricsin) en tant que gène de référence dans l'estomac. Pepsinogène C est une protéase aspartique qui fonctionne comme une enzyme digestive et est produite dans l'estomac. Cette enzyme constitue une composante essentielle de la muqueuse gastrique [23]. L'utilisation de ces trois gènes de référence, nous avons évalué l'expression génique de Chit1 et AMCase dans les tissus de souris (Figure 1). Nous avons d'abord conçu plusieurs jeux d'amorces pour la PCR quantitative et évalué leur aptitude selon qu'ils ont donné des produits simples comme reflété par une seule température de fusion (Tm) et d'une bande unique sur un gel de polyacrylamide à 10%. Les séquences nucléotidiques des produits ont également été vérifiées. Pour examiner la spécificité des amorces, chacun des produits de PCR a été amplifié dans des conditions de PCR en temps réel en utilisant un mélange d'ADNc de tissu de souris comprenant des tissus provenant de quatre stades embryonnaires et huit tissus adultes et a été dosée en combinaison avec des méthodes différentes. Comme on le voit sur la figure 2A-E, un seul pic est apparu dans les courbes de dissociation pour Chit1 (Tm = 79,7 ° C), AMCase (Tm = 79,1 ° C), GAPDH (Tm = 81,4 ° C), β-actine (Tm = 82,0 ° C) et le pepsinogène C (TM = 81,4 ° C). A la fin de la PCR, nous avons analysé les produits sur un gel de polyacrylamide à 10%. L'électrophorèse sur gel a montré des bandes uniques claires aux tailles attendues de Chit1 (69 pb), AMCase (81 pb), GAPDH (77 pb), β-actine (71 pb) et pepsinogène C (82 pb) (Figure 2F). Les séquences de nucleotides des produits de PCR ont été déterminées directement comme décrit dans la section Matériels et Méthodes. Nous avons confirmé que les produits de PCR ont été amplifiés à partir d'ADNc cibles (voir la figure 3B). Ces résultats indiquent que les produits de PCR sont amplicons spécifiques des ADNc cibles et que mésamorçage est négligeable dans nos conditions expérimentales. Nous avons cherché à comparer les niveaux d'expression de deux chitinases et trois gènes de référence sur la même échelle (Figure 1). A cet effet, nous avons mis en place un système de PCR quantitative en temps réel pour lequel un modèle standard était nécessaire pour la quantification précise (figure 3A). Nous avons construit un ADN matrice standard pour la PCR en temps réel en ligaturant les cinq fragments cibles dans un rapport un-à-un, puis ce fragment d'ADN ligaturé a été clone dans le vecteur plasmidique tel que décrit dans la section Matériaux et procédés (figure 3A) . Le 913-nucléotides de long ADN matrice contenait cinq fragments d'ADNc couvrant la région PCR-cible ainsi 9-120 bases des régions adjacentes et contenait les sites de Bgl validation de la courbe standard et quantitative en temps réel du système La quantification des deux chitinases et les ARNm de référence repose sur des courbes d'étalonnage. Des dilutions en série de l'ADN de modèle standard ont été utilisés pour construire une courbe standard pour comparer et évaluer en temps réel des stratégies de quantification RT-PCR qui ont été utilisées pour analyser les cinq ARNm. Chaque courbe standard a été généré en utilisant 10 fois des dilutions en série de l'ADN standard et les cinq paires différentes d'amorces (figure 4A-E, à gauche). l'amplification exponentielle a été maintenue sur une large gamme de cycles, ce qui donne une gamme dynamique de sept ordres de grandeur (figure 4A-E, à gauche). En utilisant l'ADN matrice standard contenant les cinq fragments d'ADNc, des quantités égales peuvent être assignées à tous les cinq gènes dans chaque utilisation de dilution pour construire les courbes standard (figure 4A-E, à droite). Pour tester l'égalité absolue des courbes, une concentration connue de l'ADNc codant complet a été amplifié et analysé par la suite comme un échantillon inconnu. Cet essai a été effectué pour vérifier que chaque dilution testée a donné lieu à la quantité attendue. Comme cela est représenté sur la figure 4A-E, à droite, des quantités égales ont été observées pour chaque dilution testée utilisée pour établir la courbe d'étalonnage (voir figure S2). La quantification des transcrits de faible abondance et les transcriptions abondantes nous permet de valider la sensibilité et la fiabilité de la PCR en temps réel, ce qui indique que notre méthode PCR en temps réel offre une large gamme dynamique de quantification, de grande précision, et une sensibilité élevée (Figure 4A- E, à droite). Ainsi, notre méthode PCR en temps réel fournit des valeurs fiables pour deux gènes chitinase et pour les gènes de référence sur la même échelle. Pour étudier le dans la régulation in vivo Nous avons ensuite comparé les ratios de AMCase à Chit1. Le nombre de copies de chaque ARNm a été déterminée en utilisant les mêmes dilutions standards. Nous avons constaté que l'estomac et de la glande sous-maxillaire principalement exprimé AMCase ARNm (Figure 5C, panneau supérieur). Les autres tissus examinés dans cette étude ont produit plus AMCase que Chit1 et Chit1 était répandue seulement dans les yeux (Figure 5C, panneau inférieur). Nous avons ensuite examiné la quantification relative dans les données originales présentées dans la figure 5, qui a été exprimée en molécules par 10 ng d'ARN total, en utilisant des gènes de ménage, comme décrit dans Matériels et méthodes. Les résultats ont été présentés comme appui de l'information sur la figure S3 (les données ont été normalisées par GAPDH) et Figure S4 (normalisée par β-actine). Ensuite, nous avons analysé les données représentées sur la figure 5 par la méthode ΔΔ Ct [21] et représentés comme des informations complémentaires sur la figure S5 (normalisée par GAPDH) et Figure S6 (normalisée par β-actine). Lorsque l'on compare l'expression relative détectée par les méthodes actuelles de quantification absolue et relative, il n'y avait pas de différence significative entre les données à l'exception de l'expression Chit1 ARNm, qui a été le plus abondamment exprimé dans les yeux quand normalisé par β-actine (voir la Figure 5, Figure S3 et la figure S4). Des résultats similaires ont également été obtenus par quantification relative en utilisant la méthode ΔΔ Ct [21] (voir la figure 5, la figure et la figure S5 S6). Cependant, nous ne pouvions pas évaluer directement les niveaux d'expression mutuels de Chit1 et AMCase lorsqu'il est analysé par la méthode ΔΔ Ct. de nombreuses études ont été menées sur la pathophysiologie de chitinases de mammifères dans les tissus pulmonaires. Dans cette étude, nous avons montré que AMCase ARNm est exprimé de manière prédominante dans les tissus de l'estomac de la souris (Figure 5). Nous avons également comparé les niveaux d'expression des gènes et des chitinases de référence en utilisant les ADNc préparés à partir des tissus pulmonaires et de l'estomac de souris 3-month-old (n = 5). Les données quantitatives sont présentés sur la figure 6. Lorsque les niveaux Chit1 ont été fixées à 1,0, les niveaux d'expression relatifs des ADNc ont été AMCase, 14; GAPDH, 196; et β-actine, 681 dans le tissu pulmonaire de souris (figure 6A). Ce résultat indique que les tissus pulmonaires expriment plus AMCase que Chit1, bien que le niveau d'expression de AMCase était inférieur à celui des deux gènes de ménage. Dans les tissus de l'estomac, lorsque le niveau Chit1 a été fixé à 1,0, les niveaux d'expression relatifs étaient AMCase, 721; pepsinogène C 2261; GAPDH, 61; et β-actine, 127 (figure 6B). Les deux GAPDH et les gènes de ß-actine sont bien connus gènes de ménage et sont constitutivement exprimés à des niveaux élevés dans la plupart des tissus et des cellules. Pepsinogène C (progastricsin) est une protéase aspartique qui fonctionne comme une enzyme digestive et est produite dans l'estomac. Cette enzyme est une composante majeure de la muqueuse gastrique [23]. Le niveau de AMCase d'expression était beaucoup plus élevé que ceux de la GAPDH et la β-actine et a été comparable à celui du pepsinogène C. Ces résultats indiquent que l'estomac AMCase est un transcrit majeur dans la muqueuse gastrique et suggèrent que cette enzyme est susceptible de jouer importants rôles physiologiques dans l'estomac. Nous avons également réexaminé l'expression relative de Chit1, AMCase et pepsinogène C représenté sur la figure 6, en utilisant des gènes domestiques par présents procédé tel que décrit ci-dessus. Les résultats ont été présentés comme appui de l'information sur la figure S7 (normalisée par GAPDH) et Figure S8 (normalisée par β-actine). Il n'y avait pas de différence significative entre les Figure 6, Figure S7 et S8 Figure. Les deux Chit1 et AMCase sont pensés pour aider à la défense de l'hôte contre les agents pathogènes contenant de la chitine [3], [ ,,,0],9]. En outre, AMCase est une molécule effectrice dans l'inflammation allergique. Ces enzymes jouent un rôle important chitinolytiques dans la physiopathologie des réactions allergiques des voies aériennes dans des modèles murins d'asthme. On sait relativement peu, cependant, sur les in vivo quantitative en temps réel RT-PCR représente une avancée importante dans la quantification de l'ARNm et permet la détection de l'expression d'un gène particulier d'intérêt au niveau moléculaire, . Il existe de nombreux rapports sur la détection de très faibles niveaux d'ARNm à l'aide de cette technologie. Cependant, en temps réel RT-PCR est pas largement utilisé, car il a encore des limites dans la quantification de multiples ARNm en utilisant la même échelle. Nous avons construit un ADN modèle standard comprenant cinq fragments d'ADNc (chaque -200 bases longues). Ces fragments inclus fragments pour deux chitinases, un marqueur et deux gènes de ménage et ont été combinés en un-à-un des ratios (figure 3A). GAPDH et β-actine, les gènes les ménagères les plus couramment utilisés, ont été inclus dans les contrôles internes et de références pour l'amplification par PCR. En outre, nous avons utilisé le pepsinogène C en tant que gène marqueur, car il est produit à un niveau élevé dans l'estomac [23]. Cette méthode a permis la quantification absolue du nombre de molécules d'ARNm Chit1 et AMCase, ainsi que le nombre de molécules d'ARNm de référence, par 10 ng d'ARN total (mole /10 ng) (figure 5 et figure 6). En outre, nous pourrions examiner la quantification relative des niveaux Chit1 et AMCase ARNm en utilisant deux gènes de ménage, GAPDH et β-actine (informations à l'appui Figure S3 et S4 Figure). quantification relative est plus facile à réaliser que la quantification absolue parce qu'un courbe d'étalonnage ne sont pas nécessaires. Dans une quantification relative, les taux d'ARNm du gène d'intérêt sont comparés à ceux des gènes de ménage [21]. Cependant, cette méthode de quantification ne parvient pas à comparer les niveaux des différents produits de transcription de gènes à la même échelle. Bien que notre procédé nécessite plusieurs étapes associées à la construction de l'ADN standard et avec les processus de validation, ce procédé peut fournir des données d'expression des gènes qui sont directement comparables entre les gènes (figure 5 et figure 6). Notre PCR en temps réel fourni une large gamme dynamique de quantification et une sensibilité élevée, et la quantification des transcrits de faible abondance nous a permis de valider la sensibilité et la fiabilité de notre méthode (Figure 4). Cette technique est très bien adapté à la quantification et la comparaison des niveaux d'ARNm à travers de multiples gènes en utilisant la même échelle. Notre méthode actuelle est applicable au génie biomédical, ainsi que pour des utilisations cliniques et pratiques. Dans la présente enquête, nous avons constaté que AMCase ARNm est synthétisé à des niveaux extraordinairement élevés dans l'estomac de la souris par rapport aux niveaux de gènes de ménage (figure 5 et figure 6). Le niveau d'ARNm AMCase est comparable à celle du pepsinogène C (progastricsin) ARNm, une composante majeure de la muqueuse gastrique dans l'estomac (figure 6B). En plus de l'estomac, glandes sous-maxillaires ont également été trouvés pour exprimer de grandes quantités de AMCase (figure 5). En ce qui concerne la synthèse d'ARNm chitinase de mammifères, à la fois l'estomac et glandes sous-maxillaires sont considérés comme des tissus notables pour la production énorme quantité de AMCase chez la souris. L'acide chlorhydrique est sécrétée dans l'estomac, ce qui crée des conditions acides pour la digestion des protéines par la pepsine à environ pH 2 [23], [24]. AMCase souris montre une stabilité de l'acide profonde et est le plus actif à un pH de 2,0, ce qui le distingue des autres enzymes de la souris [8]. La stabilité du pH et de la dépendance inhabituelle de la AMCase de la souris dans des conditions acides permet la digestion efficace des matières chitine dans des conditions acides. L'observation selon laquelle AMCase est exprimé de manière prédominante dans les points de l'estomac à sa fonction possible dans la transformation des aliments. souris sauvages mangent des aliments contenant de la chitine tels que les insectes, alors que les souris gardés dans le laboratoire mangent des aliments artificiels contenant la levure de bière séchée, qui contient également la chitine dans la paroi cellulaire. Ainsi, AMCase peut fonctionner comme une enzyme digestive qui décompose la chitine polymère dans le contenu gastrique. Le plus haut niveau de Chit1 a également été exprimé dans l'estomac de la souris. Cependant, le niveau d'expression Chit1 dans l'estomac est 1/721 à celle du niveau d'expression de AMCase et était inférieur aux niveaux de deux gènes de ménage, GAPDH et β-actine (figure 6A) expression. En outre, Chit1 ne possède aucune activité chitinolytique au pH du suc gastrique, ce qui est un pH d'environ 2 [5], [11]. Il a été démontré que Chit1 est produite au niveau des sites de pH presque neutre, tel que la partie non glandulaire de l'estomac et de l'intestin grêle [25]. Ainsi, Chit1 ne contribue pas à l'activité chitinase dans l'estomac de la souris. Bien que AMCase est exprimé principalement dans l'estomac et les glandes sous-maxillaires, tous les autres tissus examinés dans cette étude ont exprimé des niveaux de Chit1 et AMCase faibles, mais détectables, . Les niveaux d'expression Chit1 et AMCase étaient inférieurs à ceux des gènes de ménage dans ces tissus. Chit1 montre un large optimum de pH à un pH d'environ 5 [5], alors que le premier pH optimal de AMCase est un pH de 2 et le pH optimal secondaire est à un pH de 4-7, avec AMCase conservant moins de 30% de son activité à un pH de 2 à cette gamme de pH [8]. Et les deux Chit1 AMCase digèrent la chitine et le chitosan naturel via l'activité endo-chitinase et produisent chitobiose [8], [26]. Bien que AMCase est dominant sur Chit1 dans les tissus de souris beaucoup, il ne peut pas y avoir de différences significatives dans leur activité chitinolytiques dans les tissus de la souris. Plus AMCase ARNm de Chit1 ARNm a été observée dans la plupart des tissus de souris, sauf pour les yeux. Nos résultats suggèrent également que l'expression du gène de Chit1 peut être régulé de manière développementale, ce qui suggère qu'elle joue un rôle dans l'ontogenèse. L'étude de la régulation de l'expression des chitinases de mammifères pourrait donner un aperçu des rôles physiologiques de ces enzymes. Une caractérisation détaillée des régions promotrices de gènes Chit1 et Amcase ainsi que l'identification de cis Bien que les mammifères ne produisent pas de chitine ou de chitine synthase, ils sont continuellement exposés à ce polymère par exposition aux parasites et pathogènes contenant de la chitine. Le substrat pour les chitinases de mammifère est vraisemblablement chitine environnementale, telle que celle trouvée dans des champignons ou des nématodes parasitaires. Dans les poumons, les deux enzymes peuvent agir en tant que partie du mécanisme de défense de l'hôte contre les agents pathogènes contenant de la chitine, comme les moisissures et les acariens de l'environnement, qui induisent des voies respiratoires allergiques.
acétyl-D-glucosamine, est le deuxième polysaccharide le plus abondant dans la nature. Il fonctionne en tant que composant structurel majeur de champignons, crustacés et insectes mais ne se trouve pas chez les mammifères [1]. Chitinases hydrolyser le β-1-4 liaisons glucosidiques du polymère de la chitine. Bien que les mammifères ne produisent pas de chitine ou de son synthase, ils expriment deux chitinases actifs, chitotriosidase (Chit1) et chitinase mammifère acide (AMCase) [2], [3].
régulation des chitinases de mammifères. Récemment, en temps réel, une RT-PCR a été utilisée pour quantifier les taux d'ARNm dans de nombreuses études d'expression génique [17] - [20], car ce procédé est suffisamment sensible pour détecter l'ARNm de même une seule cellule. PCR en temps réel implique généralement la normalisation des niveaux du gène d'intérêt avec ceux des gènes de ménage qui sont censés être exprimé de façon constante dans tous les échantillons d'expression. Cependant, cette méthode de quantification ne parvient pas à comparer les niveaux des différents transcrits du gène sur la même échelle.
PCR en temps réel
Construction de l'ADN standard
II, Xho restriction
I, Pst
I, ou Non
I (à 5 'et 3' - fin) selon le protocole du fabricant. Les amorces avant et inverse sont énumérés dans le tableau S2. Les produits de PCR ont été purifiés en utilisant le SV Gel Wizard et PCR système Clean-Up (Promega), puis digérés par les enzymes de restriction correspondantes. Les fragments d'ADN ont été purifiés par électrophorèse sur gel d'agarose et le système Clean-Up, puis ligaturés ensemble avec l'ADN ligase T4 (Toyobo). Les fragments ligaturés ont été amplifiés en utilisant l'amorce sens 5'-GTGGATTCTGTGCCGACAAAGCAGATGGCC-3 'et l'amorce inverse 5'-TGGGTACATGGTGGTACCACCAGACAGCAC-3' avec KOD ADN polymerase plus. 3'-dA a été ajouté à l'ADN amplifié à l'aide Takara Taq HS (Takara Bio), et le produit a été purifié par électrophorèse sur gel comme décrit ci-dessus. L'ADN résultant a été clone dans le vecteur pGEM-T Easy (Promega) par clonage TA selon les instructions du fabricant. La séquence de nucleotides du plasmide résultant a été confirmé par séquençage. Le fragment d'ADN contenant multigénique linéarisé a été préparé par ré-amplification de l'ADN du plasmide avec les mêmes amorces par PCR en utilisant KOD ADN polymerase plus. Les fragments ont été purifiés et quantifiées comme décrit ci-dessus et utilisé comme ADN standard.
Préparation de cinq ADNc couvrant la région codante entière
Courbes standard
Quantification de l'ARNm par PCR en temps réel en utilisant les courbes standard ou le Ct Méthode ΔΔ
Construction de l'ADN modèle standard pour la quantification et la comparaison de l'expression génique entre cinq gènes
II, I
Xho restriction , Pst
I et Non
I (voir les détails dans la figure 3B).
PCR
Expression de Chit1 et AMCase dans les tissus
souris
Chit1 et AMCase l'expression des gènes, des échantillons d'ARN total extrait à partir de quatre stades embryonnaires et de divers tissus adultes, ont été analysées avec un test de PCR quantitative en temps réel en utilisant l'ADN standard unique (figure 3). Les résultats ont été exprimés sous forme de molécules par 10 ng d'ARN total (figure 5 et figure 6). Tant le Chit1 et AMCase ARNm ont été largement exprimés dans les tissus de souris (figure 5A et 5B). spécificités tissulaires claires ont été observées dans les motifs d'expression des deux ARNm chitinase. Des niveaux élevés d'ARNm Chit1 ont été détectés dans l'estomac de la souris (figure 5A, panneau supérieur), suivie par les yeux et les poumons. Chit1 ARNm a été détecté à des niveaux faibles, mais facilement détectables, dans d'autres tissus (figure 5A, panneau inférieur). AMCase ARNm a été principalement détectée dans l'estomac, suivie par la glande sous-maxillaire (figure 5B, panneau supérieur), mais était également présent dans d'autres tissus (figure 5B, panneau inférieur).
Analyse des Chit1, AMCase, Pepsinogène C, GAPDH, et β-actine dans les poumons et les tissus de l'estomac
Discussion
niveaux de Chit1 et AMCase d'expression mutuelle. La quantification des ARNm peut générer des informations utiles et pertinentes biomédical. Dans cette étude, nous avons établi un système de PCR quantitative en temps réel capable de déterminer les taux d'ARNm de deux chitinases de mammifères et de comparer ces niveaux avec ceux des gènes de référence en utilisant la même échelle. Nos résultats indiquent que AMCase est exprimé de manière prédominante dans l'estomac de la souris.
- et Les facteurs de
trans seront nécessaires pour comprendre l'expression sélective de ces enzymes <. br>
 Torsions des organes abdominaux – Diagnostic de l'abdomen aigu
Torsions des organes abdominaux – Diagnostic de l'abdomen aigu
 Rémission de Crohn
Rémission de Crohn
 Après la réouverture de la clinique COVID-19
Après la réouverture de la clinique COVID-19
 Les propriétés uniques et les avantages pour la santé des aliments fermentés
Les propriétés uniques et les avantages pour la santé des aliments fermentés
 Des scientifiques repèrent des gènes derrière la maladie de Crohn et la colite ulcéreuse
Des scientifiques repèrent des gènes derrière la maladie de Crohn et la colite ulcéreuse
 Combien de temps faut-il pour se remettre d'une appendicectomie ?
Combien de temps faut-il pour se remettre d'une appendicectomie ?
 Signes et symptômes précoces du syndrome pulmonaire à hantavirus (SPH)
Faits sur lhantavirus et le syndrome pulmonaire à hantavirus (SPH) Figure 1 :Image des particules dhantavirus Sin Nombre ; SOURCE :CDC/D. Loren Ketai, MD Les hantavirus sont des virus à ARN transmis
Signes et symptômes précoces du syndrome pulmonaire à hantavirus (SPH)
Faits sur lhantavirus et le syndrome pulmonaire à hantavirus (SPH) Figure 1 :Image des particules dhantavirus Sin Nombre ; SOURCE :CDC/D. Loren Ketai, MD Les hantavirus sont des virus à ARN transmis
 Les troubles gastro-intestinaux fonctionnels
Les troubles gastro-intestinaux fonctionnels (DFG) sont des troubles du système digestif dans lesquels les symptômes ne peuvent être expliqués par la présence dune anomalie structurelle ou tissulaire.
Les troubles gastro-intestinaux fonctionnels
Les troubles gastro-intestinaux fonctionnels (DFG) sont des troubles du système digestif dans lesquels les symptômes ne peuvent être expliqués par la présence dune anomalie structurelle ou tissulaire.
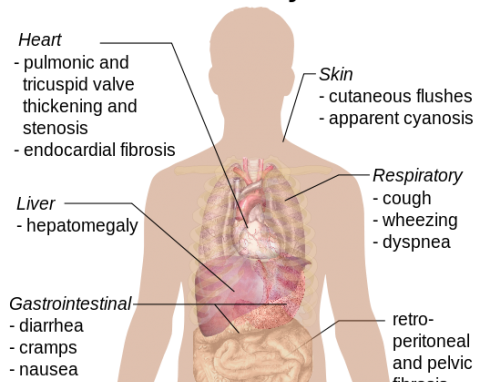 Tumeurs carcinoïdes
La tumeur carcinoïde est définie comme maligne prolifération de cellules neuroendocrines bien différenciées (cellules qui génèrent des composés bioactifs). Les tumeurs contiennent des granules de sé
Tumeurs carcinoïdes
La tumeur carcinoïde est définie comme maligne prolifération de cellules neuroendocrines bien différenciées (cellules qui génèrent des composés bioactifs). Les tumeurs contiennent des granules de sé