Contexte
Nous avons lancé une étude prospective pour identifier les altérations de la transcription associés à la résistance à la chimiothérapie acquise à partir d'échantillons avant et après biopsie du même patient et de découvrir les voies moléculaires potentiels impliqués dans échec du traitement pour aider à guider les alternatives thérapeutiques.
Méthodologie /principales constatations
A, à haut débit étude de profilage transcriptionnel prospective a été réalisée en utilisant des échantillons de biopsie endoscopiques à partir de 123 patients atteints de cancer métastatique de l'estomac avant le cisplatine et fluorouracile (CF) combinaison chimiothérapie. 22 patients ayant initialement répondu au CF ont été re-biopsiées après qu'ils ont développé une résistance à la CF. Une signature de résistance à la chimiothérapie acquise a été identifié par l'analyse des profils d'expression génique de la pré- et post-CF appariés traités échantillons.
La signature de la résistance acquise a été en mesure de séparer une cohorte séparée de 101 patients atteints de cancer gastrique nouvellement diagnostiqué en fonction du temps jusqu'à la progression après CF. classification hiérarchique en utilisant une signature de résistance acquise 633 gènes (la sélection des fonctionnalités à P Conclusion /importance Cette signature peut offrir de nouvelles perspectives sur l'identification de nouvelles cibles et des thérapeutiques nécessaires pour surmonter la résistance du cancer gastrique acquise pour FC Citation: Kim HK, Choi IJ, Kim CG, Kim HS, Oshima A, Michalowski A, et al. (2011) A Gene Expression Signature des acquis chimiorésistance à Mixtes cisplatine et fluorouracile chimiothérapie dans le cancer gastrique patients. PLoS ONE 6 (2): e16694. doi: 10.1371 /journal.pone.0016694 Editeur: Alfons Navarro, Université de Barcelone, Espagne reçues: 10 Septembre 2010; Accepté le 24 Décembre 2010; Publié le 18 Février, 2011 Ceci est un article en accès libre distribué sous les termes de la déclaration Creative Commons Public Domain qui stipule que, une fois placé dans le domaine public, ce travail peut être librement reproduit, distribué, transmis, modifié, construit sur, ou autrement utilisé par quiconque à des fins licites financement:. Ce travail a été soutenu en partie par le national Institutes of Health Programme intra-muros, Centre de recherche sur le cancer, l'Institut national du cancer; par la Korean National Cancer Center Grant 0910570 et par le Programme Centre de recherche Converging par le Ministère de l'éducation, de la science et de la technologie (2010K001121). Les bailleurs de fonds ont joué aucun rôle dans la conception de l'étude, la collecte et l'analyse des données, la décision de publier, ou de la préparation du manuscrit Intérêts concurrents:.. Les auteurs ont déclaré aucun conflit d'intérêts existent Introduction Comprendre comment les tumeurs évoluent à un niveau moléculaire pour surmonter les effets cytotoxiques de la chimiothérapie est une étape critique dans le développement d'approches thérapeutiques qui empêcheront ou surmonter chimiorésistance. Cependant, en raison des difficultés à obtenir des biopsies série tumorales de patients à différents stades de la thérapie, l'identification des altérations moléculaires qui se produisent sous forme de tumeurs deviennent résistantes à la thérapie a été un problème épineux. La collection de série d'échantillons de tumeurs solides provenant des mêmes patients a été extrêmement difficile dans le contexte clinique, mais le cancer gastrique offre une occasion unique à cet effet, car il est souvent d'abord sensibles à la chimiothérapie et endoscopies répétées peut être effectué pour surveiller la réponse de la tumeur au la chimiothérapie. Dans cette étude, des échantillons de biopsie endoscopiques ont été prélevés chez des patients atteints de cancer gastrique. Nous avons identifié une signature d'expression génique pour la chimiorésistance acquise au cisplatine et fluorouracile (CF) combinaison de chimiothérapie, en comparant les échantillons prélevés avant CF thérapie avec des échantillons prélevés sur les mêmes patients à la résistance de temps à CF développé basé sur la progression clinique objective. En utilisant cette approche, nous avons pu identifier les candidats moléculaires qui peuvent éventuellement conduire au développement de nouvelles thérapies ciblées pour le cancer gastrique. Surtout, nous avons également constaté que la signature de la chimiorésistance acquise pourrait déterminer si les patients atteints de cancer gastrique nouvellement diagnostiqués auraient une thérapie réponse CF courte ou plus soutenue. Depuis la signature de la résistance acquise est déjà fortement représentée chez les non-répondeurs et qu'il semble peu probable que les nombreuses expression changements qui se produisent au niveau mondial évoluerait dans une période relativement courte de temps, nos résultats semblent soutenir le modèle de sélection clonale classique pour la progression tumorale et chimiorésistance acquise [1]. L'identification des biomarqueurs qui distinguent les patients cancéreux qui sera ou ne sera pas bénéficier de la chimiothérapie cytotoxique permettra d'améliorer grandement la gestion clinique. Bien que des études utilisant à haut débit transcription profilage des échantillons de biopsie prétraitement ont tenté d'identifier ces prédicteurs, la performance de ces prédicteurs ont été mitigés [2]. En partie, cela peut être dû à la difficulté d'identifier les signatures de gènes robustes dans les tumeurs des populations avec une grande variation génétique. Nos données suggèrent que l'expression-profilage des échantillons de post-traitement pourrait être une approche alternative possible. Certaines études ont suggéré que les tumeurs qui se développent chimiorésistance peuvent acquérir certaines propriétés inhérentes aux cellules souches, et que le traitement de chimiothérapie conduit à un enrichissement concomitant du cancer des cellules souches in vitro Matériel et méthodes exercice patient et Ceci est la partie d'un essai prospectif approuvé par le Institutional Review Board (IRB) de l'Hôpital national Cancer Center à Goyang, en Corée (NCCNHS01-003). Tous les participants ont signé un formulaire de consentement éclairé IRB approuvé. L'admissibilité à l'enrôlement dans l'étude comprenait les paramètres suivants: 1) âge ≥18 ans; 2) histologiquement confirmés adénocarcinome gastrique; 3) cliniquement documenté métastases à distance; 4) pas malignes antérieurs ou concomitants autres que le cancer de l'estomac; 5) sans antécédents de chimiothérapie, soit adjuvant ou palliatif; et 6) une fonction adéquate de tous les organes principaux. Les patients qui ont été perdus de vue avant la fin de 6 cycles de chimiothérapie, à l'exception de la maladie progressive documentée, ont été exclus de l'analyse. Notre étude prospective avait 2 objectifs. Le premier objectif, qui est l'objet d'un autre article [4], était de développer un prédicteur génomique pour la réponse à la chimiothérapie initiale en corrélant le profilage de l'expression des données d'échantillons de prétraitement avec des résultats cliniques ( résistance intrinsèque étude Le deuxième objectif de notre étude prospective, qui est poursuivi par des analyses présentées dans ce document, était d'identifier une expression génique signature de la chimiorésistance acquise en comparant des échantillons des répondeurs cliniques pré- et post-traitement ( la résistance acquise étude Il y avait 38 patients atteints de PR entre 96 ensemble de formation (de résistance intrinsèque étude Deux pièces de des échantillons de tissus de la muqueuse gastrique grossièrement à la normale ont également été recueillies auprès de antrum de 21 volontaires sains (tableau S1). identification d'une signature de résistance acquise à CF biopsies endoscopiques ont été effectuées pour obtenir le tissu frais . Cinq à dix morceaux de tissus tumoraux frais ont été obtenus à partir de la partie non nécrotique de la tumeur à l'aide de grandes pinces à biopsie godet d'un diamètre de 7,3 mm (Olympus FB-24K-1, Olympus, Tokyo, Japon). Ensuite, les tissus frais obtenus ont été congelés dans de l'azote liquide dans les 15 minutes de la première récolte de biopsie. Des échantillons de tissus contenant des cellules tumorales d'au moins 50% ont été traités pour l'ARN comme décrit précédemment [7]. Un microgramme d'ARN total a été amplifié et hybride à une cartouche matrice HG U133A, selon le protocole du fabricant (Affymetrix, Santa Clara, CA). Toutes les données d'expression des microréseaux est disponible à l'expression génique Omnibus (GEO) Base de données (numéro d'accession GSE14210, http://www.ncbi.nlm.nih.gov/geo) [ACTUELLEMENT, ACCESS COMMENTATEURS SEULEMENT: http: //www.ncbi .nlm.nih.gov /geo /query /acc.cgi token = rtgnlocqoqeiwtw &? acc = GSE14210]. données de biopuces d'expression de gènes ont été normalisées par Robust multipuce moyenne (RMA) en utilisant R2.6. Pré et post-FC données d'expression de 22 répondeurs rebiopsied ont été normalisées indépendamment à partir des données d'expression d'un groupe distinct de 101 patients non-rebiopsied. les données de biopuces ont été analysées en utilisant ArrayTools BRB (version 3.6, National Cancer Institute, http://linus.nci.nih.gov/BRB-ArrayTools.html) [8]. changements d'expression des gènes qui distinguent la première l'état de la transcription des tumeurs à partir des profils d'expression génique lorsque les tumeurs sont devenues chimiorésistance ont été déterminées pour les 22 patients présentant une réponse initiale documenté (PR) pour le traitement des FC. les données de puces à ADN appariées a été comparée entre les échantillons obtenus avant le traitement des FC et des échantillons recueillis après la résistance à la thérapie développée. Ces données ont été analysées en utilisant l'algorithme de comparaison de classe de BRB-ArrayTools (modèle de variance aléatoire), qui calcule un appariés t Temps de progression a été tracée en utilisant la méthode de Kaplan-Meier. Un test log-rank a été utilisé pour déterminer les différences entre les courbes de survie. Le test de Wald a été utilisé pour évaluer la signification statistique du rapport de risque Cox. Des analyses de régression multivariables ont été effectuées en utilisant un modèle de risque proportionnel de Cox. Toutes ces analyses ont été effectuées en utilisant SPSS (version 15.0, SPSS, Inc., Chicago, IL). L'analyse multivariée par régression logistique ordinale a été réalisée à l'aide de SAS (version 9.1.3, SAS, Cary, Caroline du Nord), pour évaluer l'association entre l'indice prédictif de 72 gènes et réponse radiographique. facteur de transcription analyse analyses des facteurs de transcription ont été effectués pour rechercher l'enrichissement de cibles de facteurs de transcription dans les gènes comprenant la signature de la résistance acquise (BRB-ArrayTools). Tous les gènes interrogés dans cet algorithme d'analyse ont été catalogués à facteur de transcription catégories sensibles basées sur expérimentalement vérifié la réactivité du facteur de transcription. informations de curation dans la base de données transcriptionnelle Regulatory Element (TRED) Facteur de transcription contraignant [9] a été utilisé pour éliminer les cibles sans aucune vérification expérimentale. L'analyse des données de puces à ADN public des patients atteints de cancer gastrique traités chirurgicalement données microarray accessibles au public pour les patients gastriques chirurgicalement traités générés par le Stanford génomique fonctionnelle Facility ont également été obtenu à partir de la base de données NCBI GEO (GSE4007) et comprenait environ 30.300 gènes communs à ces ensembles de données. Les données de puces à ADN ont été générées et normalisé comme décrit dans Leung et al L'ensemble des gènes outil de comparaison des analyses ensembles de gènes définis par l'utilisateur pour l'expression différentielle entre les classes prédéfinies d'un ensemble de données source. Pour chaque jeu de données source, P L'ARN total a été isolé à partir endoscopique congelé des échantillons de biopsie de la muqueuse antrale recueillies auprès de 21 volontaires en bonne santé et analysés par puce à ADN comme décrit précédemment. Afin d'identifier la signature spécifique du cancer gastrique, nous avons comparé les données d'expression des 21 échantillons normaux avec 101 échantillons provenant de patients avant échantillons de chimiothérapie (à l'exclusion de 22 patients rebiopsied utilisés pour développer la signature de la résistance acquise) en utilisant la comparaison de la classe des algorithmes de BRB- ArrayTools. Sur les 101 patients, 41 patients avaient le type histologique intestinal de Lauren de tumeurs primaires et 60 avaient le type diffus. Les tumeurs de type mixte ont été classés avec le type diffus. Une signature de différenciation a été identifiée en comparant les données d'expression génique à partir des 41 échantillons de type intestinal avec des échantillons de type 60 diffuse en utilisant la comparaison de la classe des algorithmes de BRB-ArrayTools. pour générer un gène ensemble défini par l'utilisateur pour analyse notre comparaison des gènes, nous avons adopté plusieurs listes de gènes de l'œuvre publiée de Ben-Porath et al Parmi les 468 gènes surexprimés à l'état de chimiorésistance ( P Résultats de identification d'une signature de résistance acquise à CF Vingt-deux patients qui ont démontré une réponse clinique (PR) des FC ont été la thérapie biopsié avant le début du traitement, puis suit la progression de la maladie après la chimiothérapie. Pré et post-échantillons des FC ne sont pas significativement différents dans le pourcentage et les mesures de contrôle de la qualité des données de puces à ADN (tableau 1 et le tableau S2) cellule tumorale. intervalle médian entre les 2 biopsies était de 8,7 mois (intervalle interquartile, 06.04 à 12.06). Depuis la permutation P analyse facteur de comparaison ensemble des gènes de transcription indiqué que la signature de la résistance acquise est enrichi avec des cibles de multiples facteurs de transcription, y compris un ensemble de gènes cibles de MYC (TRED MYC_T00140) [9] (tableau S4). Ceci est cohérent avec les données de puces à ADN dans la littérature que la plupart des gènes sensibles à Myc surexpression sont impliquées dans la synthèse macromoléculaire, le renouvellement des protéines et le métabolisme, y compris les 30 gènes de la protéine ribosomique [26]. Nous avons voulu déterminer si l'expression de la signature de la résistance acquise dans les tumeurs du cancer de l'estomac au moment du diagnostic initial était prédictif de la réponse au traitement des FC. Expression de la signature de la résistance acquise dans un groupe séparé de 101 patients atteints de cancer gastrique non-rebiopsied a été déterminée et liée à l'évolution clinique des patients selon laquelle un grand pôle hiérarchique les patients ont été regroupés. Chez les patients sans lésions initialement mesurables par l'imagerie diagnostique, le temps jusqu'à progression (TTP) a été mesurée à partir de l'initiation du traitement CF à l'époque où un changement de traitement a été nécessaire en raison de la progression de la maladie sans équivoque. classification hiérarchique des échantillons de pré-traitement 101 a été réalisée en utilisant la signature de la résistance acquise 2446 du gène. Résultat mesuré par le TTP est significativement différente entre les patients dans chacun des deux groupes primaires. Les patients du groupe avec une expression accrue des gènes surexprimés dans l'état de chimiorésistance avaient un TTP significativement plus courte que les patients ayant une plus faible expression de ces gènes (Log-rank P Nous avons également souhaité examiner plus si ces signatures de résistance acquises étaient prédictifs de la réponse des FC ou représentent une signature pronostique général qui pourrait prédire la survie de 88 patients atteints de cancer gastrique qui ont été traités par la chirurgie seule et non par la chimiothérapie 10. Aucun des deux signatures de résistance acquises (2.446 ou 633 gènes) était prédictive de la survie chez les patients atteints de cancer gastrique traités chirurgicalement en utilisant la même méthode de classification hiérarchique comme ci-dessus (Log-rank P Ces signatures de résistance acquises ont ensuite été comparés avec la signature de la résistance aux médicaments intrinsèque d'un groupe distinct de 101 patients non-rebiopsied, en utilisant une analyse de comparaison ensemble des gènes de BRB-ArrayTools [12] . En bref, cet algorithme calcule un P l'utilisation similaire de comparaison ensemble des gènes analyses, les signatures de résistance acquises ont ensuite été comparés avec " gastrique spécifique du cancer signature Étant donné que réseaux de régulation complexes dans les cellules souches peuvent être mieux détectées par analyse de l'expression de nombreux gènes, nous avons effectué plusieurs gènes mis en comparaison des analyses comparant nos signatures de résistance acquises avec les signatures de cellules ES publiées tel que rapporté par Ben-Porath et al Notre signature de résistance acquise, contrairement à la
< 0,01), séparés les échantillons de patients 101 de prétraitement en deux groupes avec significativement différents temps jusqu'à progression (2,5 vs 4,7 mois). Cette signature de 633 gènes inclus la régulation positive de AKT1
, eIF4B
et rpS6
(mTOR), les gènes de réparation de l'ADN et le métabolisme des médicaments, et a été enrichi pour les gènes surexprimé dans embryonnaires signatures de cellules souches. Une signature de résistance acquise 72 gène (un sous-ensemble de la signature 633 du gène a également identifié dans ES ensembles de gènes liés aux cellules) était un facteur prédictif indépendant pour le temps jusqu'à progression (ajusté P
= 0,011) et la survie (ajustée P
= 0,034) de ces 101 patients.
[3]. Nous démontrons en outre que la signature de la résistance acquise est enrichie pour les gènes précédemment identifiés dans souches embryonnaires (ES) signatures d'expression cellulaire, ce qui suggère en outre que le cancer gastrique, chimiorésistance provient de la sélection des cellules pré-existantes avec des caractéristiques particulières de cellules souches.
suivi
). Taille de l'échantillon de l'essai a été prévu sur la base de ce premier objectif. Pour l'ensemble de la formation, on estime que 91 événements pour être requis à α = 0,001, β = 0,05, τ (écart-type de l'intensité du journal) = 0,75, et δ (rapport de risque associé au changement d'une unité d'intensité du journal) = 2. par conséquent, 96 échantillons de prétraitement ont été recueillies à partir de Août 2001 à Janvier 2005, l'ensemble de la formation (pour le résistance intrinsèque étude
). Un deuxième groupe de 27 patients éligibles a été inscrit comme la cohorte de validation de tableau entre Février 2005 et Avril 2006, qui comprend 22 patients traités par CF, et 5 patients traités par cisplatine plus capécitabine orale (un fluorouracile pro-médicament considéré comme équivalent à fluorouracil; CX ). la thérapie CX a été démontrée pour être thérapeutiquement équivalent au régime de CF pour le cancer gastrique métastatique [5].
). Après une biopsie endoscopique initiale, tous les patients de l'étude ont été prospectivement traités et suivis. Les patients ont été traités avec le cisplatine (60 mg /m 2, D1) en combinaison avec soit fluorouracile (1 g /m 2 pendant 5 jours; n = 118) ou la capécitabine (Xeloda, Roche; 1,250 mg /m 2 fois par jour pendant 2 semaines; n = 5) 5 toutes les 3 semaines. doses de chimiothérapie ont été réduites en fonction de toxicités et l'état de la performance du patient. systèmes de modification de dose spécifiques pour le cycle de traitement subséquent étaient à la discrétion de l'oncologue traitant. Le programme de traitement pour fluorouracile pourrait être raccourci à la discrétion de l'oncologue de 5 à 3 jours pour les patients âgés (≥70 ans) ou les patients ayant un faible indice de performance (Eastern Cooperative Oncology Group (ECOG) de statut de performance ≥2). spirale abdominale tomodensitométrie (TDM) ont été effectuées pour tous les patients tous les 3 cycles de chimiothérapie ( i.e.
, 9 semaines). Une réponse objective a été documentée pour les patients avec une maladie mesurable selon l'Organisation mondiale de la santé (OMS) critères [6]. Une réponse partielle (RP) a été défini comme une baisse de plus de 50% de la somme des produits des 2 plus grands diamètres perpendiculaires de lésions mesurables pendant au moins 4 semaines, mais une confirmation CT n'a pas été systématiquement effectué 4 semaines après la documentation initiale de PR.
). Les patients atteints de PR ont subi une biopsie de suivi à la progression de la maladie de temps a été observée ( i.e.
, Maladie progressive selon les critères de l'OMS), appelé le «état de chimiorésistance". des échantillons de biopsie adéquats de tumeurs dans un état de chimiorésistance étaient disponibles auprès de 22 patients atteints de PR (57,9%). Etat de chimiorésistance des échantillons de biopsie des 16 autres patients (42,1%) ne pouvaient pas être profilées en raison de l'insuffisance ou l'autre ARN quantité /qualité ou le refus de patients. Il n'y avait aucune différence d'âge, le sexe, le type histologique, le temps jusqu'à progression (TTP), et la survie globale entre les 22 patients re-biopsie et les 16 autres patients qui avaient PR, mais ne sont pas re-biopsiées. Des échantillons ont été prélevés au moins 2 semaines après la dernière dose de fluorouracile et
avant la chimiothérapie de deuxième ligne a été lancé, afin de minimiser les effets des médicaments aigus sur le profil d'expression.
-test pour chaque gène en utilisant les log-intensités RMA-résumées pour Affymetrix tableaux de U133A. Les gènes exprimés de manière différentielle entre ces 22 échantillons appariés définis la signature de la résistance acquise. A la sélection de fonction P
Seuils -value de 0,05 et 0,01, une valeur de permutation de P a été calculé, ce qui est la proportion de permutations aléatoires qui identifient un nombre similaire de gènes importants que l'on trouve lorsque l'on compare les étiquettes de classe véritables.
[10]. effets Batch dans l'expression génique ont été enlevés avec sonde sage moyenne de centrage et les données manquantes ont été imputées à la méthode moyenne du plus proche voisin [11]. Les tableaux des clones d'ADNc ont été annotés en utilisant SOURCE (Base de données de Stanford Microarray) et le Entrez- GeneID a été utilisé comme l'identificateur de mappage pour le réseau Affymetrix HG-U133A.
Comparaison de jeu de Gene analyses
-value est
calculée pour chaque gène de corréler le niveau d'expression vs. Temps de survie en utilisant un modèle des risques proportionnels (ou pour l'expression différentielle entre pré-défini les classes, en fonction de la nature du phénotype), générant une liste de gène d'un projet évalué BRB-ArrayTools donné. Pour un ensemble de N
gènes, la statistique de LS est défini comme étant le logarithme naturel négatif moyenne de la P
-values des tests univariés monogéniques appropriés [12]. Une statistique sommaire est calculée qui résume ces Les valeurs P
sur le gène ensemble défini par l'utilisateur; la statistique récapitulative est log moyenne ( P
) pour le résumé de LS de la façon dont les P
valeurs diffèrent d'une distribution uniforme pour LS [12]. La statistique sommaire est liée à la distribution des statistiques sommaires pour les échantillons aléatoires de N de gènes, échantillonnées à partir de ceux qui sont représentés sur le tableau. Ici N
est le nombre de gènes dans le gène ensemble défini par l'utilisateur. 100.000 ensembles de gènes aléatoires ont été échantillonnées pour calculer cette distribution. Le LS P
valeur est la proportion des ensembles aléatoires de N de gènes avec des statistiques sommaires moyennes plus petites que les LS résumés calculés pour les données réelles. Cette approche est utilisée pour une variété de types de corrélations entre les taux d'expression de gènes et le phénotype. La nature du phénotype (par exemple, le temps de survie ou des indicateurs binaires) détermine la manière dont les spécifique du gène P
valeurs sont calculées. Un LS P
valeur inférieure à 0,005 est considéré comme significatif.
Identification d'une signature spécifique de cancer gastrique et une différenciation de cancer gastrique signature
Génération de signatures de cellules ES à partir de données publiées
[13], dans lequel plusieurs ensembles de gènes associés à l'identité des cellules ES ont été compilées pour les analyses de comparaison ensemble des gènes. Un " ES expression mis
" a déjà été défini par Ben-Porath et al
[13] que les gènes sur-exprimés dans les cellules ES humaines dans au moins 5 des 20 études de profilage [14 ]. Cette ES set d'expression a ensuite été modifié [13] de sorte que les gènes de la «prolifération» Gene Ontology et le pôle de la prolifération du cancer du sein [13], [15] ont été exclus et désignés comme ES Situé sans gènes de prolifération
. Listes de gènes cibles pour MYC [16], SOX2 [17], OCT4 [17], NANOG [17], SUZ12 [18], l'EED [18] et H3K27 [18], qui sont des facteurs de transcription dans les cellules souches, ont également été adoptés de Ben-Porath [13]. Ces gènes ont été identifiés à l'origine par des études de réseau chromatine immunoprécipitation [16] - [18]. Pour les analyses de notre comparaison ensemble de gènes, ENTREZ ID [13] de gènes cibles ont été cartographiés pour sonder les ID fixés sur la matrice HG-U133A (www.NetAffx.com).
Identification d'un indice prédictif 72 gène
< 0,01), 72 gènes uniques étaient membres d'au moins un des 4 publié ES ensembles de gènes liés à cellules
( " ES définir sans gènes de prolifération
" [13], [15], le MYC transcription ensemble du gène cible de facteur expérimentalement validé (TRED MYC_T00140) [9], et les gènes cibles de MYC et SOX2 identifiés par une étude du tableau chromatine immunoprécipitation [16], [17]). Un prédicteur génomique (appelé «indice prédictif 72-gène") a été construit en calculant la combinaison linéaire pondérée des valeurs de signal de log de ces 72 gènes uniques qui se chevauchent entre la signature de la résistance acquise et " ES ensembles de gènes liés aux cellules
". Le univariée t
-statistics pour comparer les classes (acquis chimiorésistance vs. États prétraitement
) ont été utilisés comme les poids. BRB-ArrayTools (la prédiction de classe) a été utilisé pour calculer le t
-value de chaque gène. Le pouvoir prédictif de l'indice prédictif de 72 gènes a été testé pour le temps de la progression et de la survie à l'aide du modèle de Cox des risques proportionnels.
valeurs étaient toujours inférieures à 0,05 à P
Seuils pour la sélection des fonctionnalités de 0,01 et 0,05 (permutation P
valeurs, 0,012 et 0,006, respectivement), cela démontre que l'expression du gène est significativement différente entre la chimiorésistance et les états de prétraitement. Les gènes exprimés de manière différentielle entre l'état de 22 tumeurs qui se sont révélées d'abord sensibles à la chimiothérapie FC et les tumeurs des mêmes patients après avoir évolué dans un état chimiorésistance acquis prétraitement ont été identifiés comme les "signatures de résistance acquis». 2.446 gènes ont été identifiés dans la signature de la résistance acquise avec une sélection de caractéristique de P
< 0,05, alors que 633 gènes ont été identifiés en utilisant une sélection de caractéristique de P
< 0,01. La catégorie fonctionnelle les plus représentées dans la signature de la résistance acquise était (tableau S3; Ingenuity Pathway Analysis [www.ingenuity.com])
synthèse de protéine, qui comprend AKT1
, ARNm de sous-unité ribosomique ( rpS6, RPL13, RPL14, RPL15, RPL18, RPL29, RPL3, RPL30, RPL4, RPS11, RPS19, RPS9
), et les facteurs d'initiation de traduction eucaryotes ( eIF4B EIF3D, EIF3E, eIF3f, EIF3H
). /modules de signalisation Akt mTOR et Ras-MAPK sont deux voies les plus étudiées qui présentent un effet primordial sur la régulation de la traduction [19]. Compte tenu de la surexpression simultanée de ces éléments clés de cette voie ( AKT1
( P
= 0,0012), eIF4B
( P
= 0,0089), et rpS6
( P
= 0,0009)), la voie PI3K /Akt /mTOR voie de transduction du signal est présumé être activé dans l'état de résistance acquise (Figure S1). AKT1 a été liée à in vitro
résistance au cisplatine [20] - [22]. l'inhibition de mTOR a aussi été connu pour inverser in vitro
acquis une résistance à la thérapie endocrinienne et les inhibiteurs de l'EGFR de cancers du sein et du poumon, respectivement [23], [24]. Depuis ERBB2
est également régulée positivement dans la signature de la résistance acquise ( P
= 0,0065), ERBB2
peuvent jouer un rôle dans la régulation positive de Synthèse de protéine
gènes concernant la PI, par l'activation de la voie mTOR [25].
La signature de la résistance acquise ségrège patients en fonction du temps de progression de la maladie après un traitement CF, mais n'est pas pronostique chez les patients atteints de cancer gastrique traités uniquement par la chirurgie
value = 0,033) (figure 1A). Afin d'évaluer davantage l'association de ces 2.446 gènes avec TTP de 101 patients, nous avons également effectué une analyse de prédiction du risque de survie de BRB-ArrayTools, dans lequel l'ensemble du processus de validation croisée 10 fois a été répété en utilisant 2.446 gènes et un log- classement statistique pour TTP entre 2 prédit des groupes à risque a été obtenu pour chaque jeu de données aléatoire avec des données TTP mélangées parmi 101 patients [8], [27]. La permutation P valeur
pour tester l'hypothèse nulle selon laquelle il n'y a pas de relation entre 2.446 gènes et TTP, qui est la zone de queue de cette distribution nulle au-delà de la valeur du log-rank obtenue pour les données réelles, a été estimé à 0,06 , ce qui suggère une signification limite de l'association. Les patients du groupe avec une expression accrue de 468 gènes régulés à la hausse dans l'état de chimiorésistance à P
< 0,01 a également eu un TTP significativement plus courte que les patients ayant une plus faible expression de ces gènes (Log-rank P
value = 0,012) (figure 1B). Ces résultats suggèrent que la signature de la résistance acquise reflète le profil moléculaire réel des clones chimiorésistantes, et non pas des effets non spécifiques de la drogue.
valeurs, 0,84 et 0,41, respectivement ). Ainsi, la signature de la résistance acquise est prédictive de la réponse du patient à la CF et non pas seulement le pronostic des patients atteints de cancer de l'estomac en général.
Les signatures des actions de résistance acquis de nombreuses caractéristiques avec la signature de résistance intrinsèque, mais pas avec un cancer- gastrique signature spécifique ou une différenciation de cancer gastrique signature
-value pour chacun des 2.446 gènes de corréler le niveau d'expression vs. Es TTP de ces 101 patients en utilisant un modèle des risques proportionnels. Ensuite, il calcule signifie logarithme négatif naturel des P
-values des tests univariés monogéniques (LS statistique de cet ensemble de 2.446 gènes) et la proportion des ensembles aléatoires de 2.446 gènes avec des statistiques sommaires moyenne plus petite que la LS résumés calculés pour les données réelles (LS P
valeur). La même analyse a été répétée pour 633 gènes sélectionnés à P
< 0,01. Conformément aux résultats de la classification hiérarchique des analyses, les signatures de résistance acquises ont été jugées hautement enrichi dans la "signature de résistance intrinsèque" d'un groupe distinct de 101 patients atteints de mucoviscidose traité. LS ré-échantillonnage P
valeurs étaient < 10 -5 pour les deux ensembles de gènes définis par l'utilisateur sélectionnées avec différents seuils pour définir la signature de la résistance acquise ( ie
, pour 2446 et 633 gènes). Les gènes qui se chevauchent entre les signatures de résistance acquises et intrinsèques sont énumérés dans le tableau 2. La figure 2C b
affiche graphiquement que 468 gènes surexprimés à l'état de chimiorésistance de 22 patients rebiopsied ( P
< 0,01) show la surexpression concordante chez les patients non-rebiopsied avec TTP plus courte, tandis que 165 gènes régulés à la baisse à l'état de chimiorésistance montrent la surexpression concordante chez les patients avec plus TTP.
" et "gastrique différenciation du cancer signature
" de ces 101 patients. Pour comparer la signature de la résistance acquise avec " gastrique spécifique du cancer signature
", la statistique de LS de 2.446 gènes dans la signature de la résistance acquise a été estimée en calculant un logarithme naturel négatif moyenne de la P
-values des gènes tests univariés simples pour l'expression différentielle de chacun des 2.446 gènes entre 101 patients atteints de cancer gastrique et 21 volontaires en bonne santé. Pas de chevauchement significatif dans l'expression du gène a été observée en comparant la signature de la résistance acquise à un " gastrique spécifique du cancer signature
" (LS P
valeur = 0,96; Tableau S5). De même, il n'y avait pas de chevauchement important entre la signature de la résistance acquise 2446-gène et un «différenciation de cancer gastrique signature
" que nous avons identifié à travers la comparaison de l'expression génique entre le intestinal- de Lauren (n = 41) vs.
diffuse de type (n = 60) des tumeurs, soit (LS P
valeur = 0,024; Tableau S5). Ces résultats suggèrent en outre que la signature de la résistance acquise représente un ensemble de gènes surexprimés en association avec la chimiorésistance, et non le cancer en général.
La résistance des actions de signature acquises caractéristiques avec les signatures de cellules souches
13 (tableau S6). Nous avons supposé que la comparaison de la signature de la résistance acquise avec les signatures de cellules ES serait instructif, car il a été suggéré que les cellules progénitrices du cancer possèdent souches traits analogues à des cellules [28].
 Une étude génétiquement modifiée des bactéries intestinales réduit le risque de cancer colorectal chez la souris
Une étude génétiquement modifiée des bactéries intestinales réduit le risque de cancer colorectal chez la souris
 Allergies liées à une consommation plus élevée de malbouffe,
Allergies liées à une consommation plus élevée de malbouffe,
 L'utilisation d'antibiotiques à long terme chez les prématurés favorise les bactéries intestinales résistantes aux médicaments
L'utilisation d'antibiotiques à long terme chez les prématurés favorise les bactéries intestinales résistantes aux médicaments
 Pourquoi ai-je besoin d'une coloscopie?
Pourquoi ai-je besoin d'une coloscopie?
 Diagnostic des infections virales à l'aide de technologies micro et nanométriques
Diagnostic des infections virales à l'aide de technologies micro et nanométriques
 Faire face à la maladie cœliaque
Faire face à la maladie cœliaque
 Le microbiome du pénis est un réservoir pour les bactéries associées à la vaginose bactérienne
Parmi les infections sexuellement transmissibles (IST), La vaginose bactérienne affecte plus de 20 pour cent des femmes à travers le monde. La condition découle de la prolifération de certaines bactér
Le microbiome du pénis est un réservoir pour les bactéries associées à la vaginose bactérienne
Parmi les infections sexuellement transmissibles (IST), La vaginose bactérienne affecte plus de 20 pour cent des femmes à travers le monde. La condition découle de la prolifération de certaines bactér
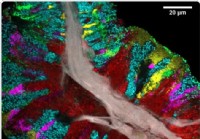 Carte détaillée du microbiome de la langue humaine
Une nouvelle étude publiée dans la revue Rapports de cellule en mars 2020 rapporte lutilisation de techniques dimagerie spectrale avancées et rapides pour développer une carte détaillée des communau
Carte détaillée du microbiome de la langue humaine
Une nouvelle étude publiée dans la revue Rapports de cellule en mars 2020 rapporte lutilisation de techniques dimagerie spectrale avancées et rapides pour développer une carte détaillée des communau
 Symposium scientifique à LABVOLUTION se concentre sur les questions clés des sciences de la vie
Le Symposium Scientifique de LABVOLUTION se concentrera sur les questions clés des sciences de la vie. Par conséquent, une forte demande de visiteurs est attendue. Le Symposium Scientifiq
Symposium scientifique à LABVOLUTION se concentre sur les questions clés des sciences de la vie
Le Symposium Scientifique de LABVOLUTION se concentrera sur les questions clés des sciences de la vie. Par conséquent, une forte demande de visiteurs est attendue. Le Symposium Scientifiq