Abstract
Genomic copy number afwijkingen (CNA's) bij maagkanker zijn reeds uitvoerig gekenmerkt door een reeks vergelijkende genomische hybridisatie (array CGH) analyse. Het optreden van genomische CNAs in het proces van submucosale invasie en metastase in lymfeklieren vroege maagkanker vrij onduidelijk. In deze studie, om dit probleem aan te pakken, we verzamelde een totaal van 59 tumor monsters van 27 patiënten met submucosale-invasieve maagkanker (SMGC), hun genomische profielen door array CGH geanalyseerd en vergeleken tussen gepaarde monsters van mucosale (MU) en submucosale (SM) invasie (23 paren) en SM invasie en lymfeknoop (LN) metastase (9 paren). Aanvankelijk veronderstelden we dat de aanschaf van specifiek CNA (s) is belangrijk voor deze processen. Echter, zagen we geen significant verschil in het aantal genomische CNAs tussen gepaarde MU en SM en tussen gepaarde SM en LN. Bovendien waren we niet in staat om CNA's die specifiek verband houden met SM invasie of LN metastase te vinden. Onder de 23 onderzochte gevallen, 15 had een aantal vergelijkbaar patroon van genomische profilering tussen SM en MU. Interessant is dat 13 van de 15 gevallen bleek ook enkele verschillen in genomisch profielen. Deze resultaten suggereren dat de meeste SMGCS bestaan uit heterogene subpopulaties afgeleid van dezelfde kloon. Vergelijking van de genomische CNA's tussen SMGCS met en zonder LN metastase bleek dat winst van 11q13, 11q14, 11q22, 14q32 en de amplificatie van 17q21 kwamen vaker voor bij metastatische SMGCS, wat suggereert dat deze CNA's gerelateerd zijn aan LN metastase van de vroege maagkanker. Samenvattend zijn onze gegevens suggereren dat de verzameling van genetisch onderscheiden subklonen plaats van de aanschaf specifieke CNA bij MU, is volwaardige componenten van submucosale invasie en dat subklonen die winst van 11q13, 11q14, 11q22, 14q32 of amplificatie van 17q21 verwerven waarschijnlijk metastatische geworden
Visum:. Kuroda A, Tsukamoto Y, Nguyen LT, Noguchi T, Takeuchi I, Uchida M, et al. (2011) Genomic Profilering van Submucosale-invasieve Maag Kanker door Array-Based Comparative Genomic Hybridization. PLoS ONE 6 (7): e22313. doi: 10.1371 /journal.pone.0022313
Editor: Giuseppe Novelli, Tor Vergata Universiteit van Rome, Italië |
Ontvangen: 25 februari 2011; Aanvaard: 19 juni 2011; Gepubliceerd: 21 juli 2011
Copyright: © 2011 Kuroda et al. Dit is een open-access artikel gedistribueerd onder de voorwaarden van de Creative Commons Attribution License, die onbeperkt gebruik, distributie en reproductie maakt in elk medium, op voorwaarde dat de oorspronkelijke auteur en de bron worden gecrediteerd
Financiering:. Dit onderzoek werd mede ondersteund door het Ministerie van Onderwijs, Wetenschap, Sport en Cultuur van Japan, en Subsidies-in-Aid for Young Scientists (B), nr 20790286 (http://www.mext.go.jp), en de onderzoek Fonds aan het oordeel van de voorzitter, Oita University (http://www.oita-u.ac.jp/english/index.html). Geen extra externe financiering ontvangen voor deze studie. De financiers hadden geen rol in de studie design, het verzamelen van gegevens en analyse, besluit te publiceren, of de voorbereiding van het manuscript
Competing belangen:.. De auteurs hebben verklaard dat er geen tegenstrijdige belangen bestaan
Introductie
Maagkanker blijft een van de meest dodelijke ziekten, ondanks de gestaag dalende trend wereldwijd. Overall, is sterfte door maagkanker schatting 700.000 gevallen per jaar (10,4% van alle sterfte aan kanker) zijn, als 2de na longkanker [1]. Klinisch resultaat is beter wanneer de tumorcellen worden beperkt tot de mucosa. Echter, wanneer de tumorcellen door de muscularis mucosa passeren, de klinische uitkomst erger, omdat het risico van lymfeknoop metastasen, die een van de belangrijkste prognostische factoren bij maagkanker, verhoogt tot 18% of meer, vergeleken met minder dan 4% wanneer de tumorcellen beperkt blijven tot de mucosa [2], [3]. Daarom is een beter begrip van de bij het proces van submucosale invasie mechanismen vereist.
Er wordt momenteel erkend dat meerdere stappen accumulatie van genetische afwijkingen is verantwoordelijk voor het ontstaan en de progressie van verschillende kankers [4]. In feite is gerapporteerd dat het totale aantal genomische afwijkingen toeneemt tot tumorprogressie in verschillende typen tumoren [5]. We vonden ook dat de frequenties van de winsten op 20q, 20p12, 1q42, 3q27 en 13q34 en verliezen aan 4q34-qter, 4p15, 9p21, 16q22, 18q21 en 3p14, die vaak had ontdekt bij maagkanker, kwamen vaker voor bij AGC dan in EGC [6]. Inmiddels is recent gerapporteerd dat in de loop van tumorprogressie, een tumorcel vertrek evolueert naar verscheidene genetisch onderscheiden subpopulaties door het verwerven van een groot aantal genomische afwijkingen. De verkregen tumormassa, dat bestaat uit genetisch heterogene subpopulaties, wordt beschouwd als resistent tegen verschillende omgevingsselectie druk worden [7], [8], [9], [10].
Array-gebaseerde vergelijkende genomische hybridisatie (array CGH) geeft informatie over de genomische kopij afwijkingen (CNA's) over het gehele genoom [11]. Bovendien CGH ook voor de studie van intratumorale genomisch heterogeniteit [12], [13], [14], [15]. Hoewel verschillende groepen gebruikt array CGH regio's herbergen oncogene of tumor-onderdrukkende genen bij maagkanker [6], [16], [17], [18], [19], [20], [21], [22 identificeren ], [23], [24], [25], CNA's met betrekking tot invasie en de vroege fase van lymfeknoop metastasen submucosale nog niet vastgesteld. Aangezien de meeste eerdere studies van CNA bij maagkanker slechts één monster per tumor geanalyseerd zijn details van de heterogeniteit van de genomische profielen binnen één maagkanker grotendeels onduidelijk gebleven.
In deze studie hebben we het betrokkenheid van genomisch CNAs in het proces van submucosale invasie en metastase in lymfeklieren vroege maagkanker. Hiertoe verzamelden we tumormonsters van verschillende delen van dezelfde tumor afzonderlijk hun genomische profiel van array CGH geanalyseerd en vergeleken de genomische profielen tussen gepaarde monsters van mucosale (MU) en submucosale (SM) porties en SM gedeelte en lymfe knooppunt (LN) metastase. Verder kan door het vergelijken van de CNA tussen uitgezaaide en niet-uitgezaaide submucosale-invasieve maagkanker (SMGC) gaf aan welke kandidaat CNA's met betrekking tot LN metastase van vroege maagkanker.
ethiek Verklaring
Deze studie werd goedgekeurd door de ethische commissie van Oita University Hospital (goedkeuring nr P-05-04). Informed schriftelijke toestemming werd verkregen van alle patiënten en /of hun families.
Zevenentwintig SMGCS werden chirurgisch weggesneden bij Oita University Hospital. Weefsel secties werden gesneden uit met formaline gefixeerd paraffine-ingebed weefsel en gekleurd met hematoxyline-eosine (HE) voor histologische analyse en toluidine blauw (Wako, Osaka, Japan) voor extractie van genomisch DNA (Figuur 1A). Met behulp van laser-capture microdissectie, we verzamelde 1-3 monsters uit de MU, SM en /of gemetastaseerde LN deel van dezelfde SMGC weefsel afzonderlijk. Daardoor konden we in totaal 59 monsters verkregen uit 27 patiënten (tabel 1). Alle monsters die een deel van tumorcellen van meer dan 70% van het totaal. Genomisch DNA werd geëxtraheerd volgens de standaard proteinase K digestie methode, gevolgd door fenol /chloroform extractie. Niet-neoplastische maag weefsel van dezelfde patiënt werd als een normale controle.
array CGH-analyse werd uitgevoerd met 44 K CGH oligonucleotide arrays (Agilent Technologies Inc. , Palo Alto, CA). Labeling en hybridisatie werden uitgevoerd volgens het protocol geleverd door Agilent Technologies Inc. kort 0,85-2 ug tumor DNA en evenveel controle DNA werd geknipt met AluI en RsaI (Promega, Madison, WI, USA) gedurende 24 uur bij 37 ° C. De gedigereerde tumor en de controle DNA werden gemerkt met Cy5-dUTP en Cy3-dUTP respectievelijk met een Genomic DNA Labeling Kit Plus (Agilent), gezuiverd met Microcon YM-30 filter (Millipore, Billerica, MA, USA), en geconcentreerd 80,5 pl. Gelijke hoeveelheden tumor en controle DNA's werden vervolgens samengevoegd en gemengd met menselijk Cot-1 DNA, opgelost in hybridisatiebuffer (Agilent Oligo aCGH Hybridization Kit, Agilent Technologies), gedenatureerd en gehybridiseerd aan de array-CGH bij 65 ° C gedurende 24 uur. Glasplaatjes werden gewassen en vervolgens gescand in overeenstemming met de instructies van de fabrikant.
Microarray beelden werden geanalyseerd met behulp van kenmerken v.9.5.3.1 (Agilent Technologies) met lineaire normalisatie (protocol CGH-v4_95_Feb07), en de resulterende gegevens werden ingevoerd in DNA Analytics v.4.0.81 (Agilent Technologies). Na normalisatie van ruwe gegevens, werd de log2ratio van Cy5 (tumor) tot Cy3 (controle) berekend. Afwijkende gebieden werden bepaald door de ADM-2 algoritme op een drempel van 8,0. Om winsten en verliezen op te sporen, zetten we de waarden van de parameters voor aberratie filters als: minimum aantal sondes in regio 2, minimum absolute gemiddelde log2ratio voor regio 0.26, maximum aantal afwijkende regio 10.000, en het percentage penetrantie per functie 0. Ook aan detecteren amplificaties en deleties, zetten we de waarden van de parameters voor aberratie filters als: minimum aantal sondes in regio 2, minimum absolute gemiddelde log2ratio voor regio 1.0, maximum aantal afwijkende regio 10.000, en het percentage penetrantie per functie 0. gegevens gegenereerd door probes toegewezen aan de X- en Y-chromosomen werden geëlimineerd. Genoomposities probes en afwijkende gebieden werden gebaseerd op de UCSC maart 2006 humane referentiesequentie (hg18) (NCBI bouwen 36 referentiesequentie). Alle gegevens zijn MIAME compliant (http://www.mged.org/Workgroups/MIAME/miame.html) en de ruwe gegevens zijn in de MIAME-compliant GEO-database (http gestort: //www.ncbi.nlm.nih .gov /geo /, toegangsnummer GSE26800). Een overzicht van de experimentele opzet wordt getoond in Figuur 1B. Ter vergelijking van gepaarde CNAs tussen MU en SM porties, selecteerden we 23 gevallen van het totaal van 27 (Figuur 1B, a en b) omdat de genomische profielen van beide delen in deze gevallen waren succesvol geanalyseerd. Ook voor de vergelijking van CNA's tussen gepaarde SM en LN porties, hebben we gekozen voor 9 van de 12 gevallen met een LN deel (Figuur 1B, c en d). Verder vergeleken we de frequenties van CNAs tussen gevallen met en zonder LN metastase (Figuur 1B, e en f).
Immunohistochemie werd uitgevoerd zoals eerder beschreven [21] met anti- EGFR (1:100; Dako, Glostrup, Denemarken), anti-CTTN (1:200; Abcam, Cambridge, MA, USA) en anti-ERBB2 (1:800; Cell Signaling Technology, Berverly, MA, USA) antilichamen.
Statistische analyse
Gekoppelde t-test en Fisher's exact test werden gebruikt. Verschillen op P Resultaten Genomic clonaliteit en heterogeniteit in mucosale en submucosale delen van SMGC Om de onderzoeken betrokkenheid van genomisch CNAs in het proces van submucosale invasie werden eerst het aantal gepaarde CNAs tussen MU en SM monsters van 23 SMGCS (Figuur 2A). Elf van de 23 gevallen toonde een toegenomen aantal CNAs in het SM gedeelte ten opzichte van het MU gedeelte 11 vertoonden een verminderd aantal en de resterende geval geen verschil zien (Figuur 2A). Dientengevolge was er geen statistisch significant verschil in het aantal gepaarde CNAs tussen MU en SM gedeelten (Figuur 2A, niet significant gepaarde t-test). Bovendien identificeren CNA specifiek aan submucosale invasie, vergeleken we de gemiddelde frequentie van CNAs in de MU deel met die in de gekoppelde SM gedeelte (figuur 2B), maar konden recht komen. De onderzoeken verschil tussen MU en CNAs SM uit dezelfde tumor, vergeleken we de genomische profielen van gepaarde MU en SM deelneemt. Een representatief geval is getoond in figuur 2C, D, E en F. De gepaarde MU en SM monsters deelden een vergelijkbaar patroon van genomische aberratie in chromosoom 9p (figuur 2D). Er waren echter duidelijke genomische afwijkingen in chromosomen 7p en 11 in dezelfde zaak, zoals in figuur 2E en F. Amplificatie van 7p12 pas in MU waargenomen, maar niet in SM (figuur 2E), en winst van chromosoom 11 werd waargenomen alleen SM, maar niet in MU (figuur 2F). Deze resultaten suggereren dat tumorcellen in de MU en SM van het onderhavige geval klonaal verwante, maar uit genetisch heterogene subpopulaties. vervolgens te bepalen of de tumorcellen toont amplificatie van 7p12 en die tonen winst van 11q13 van bij 4 waren echt beperkt tot de MU en SM, respectievelijk, analyseerden we coupes van geval 4 door immunohistochemie met antilichamen tegen EGFR, die alleen in de MU gedeelte (figuur 2E) werd versterkt, en CTTN, die alleen in de SM werd opgedaan gedeelte (Figuur 2F). Zoals getoond in Figuur 3, werd een positieve immunoreactiviteit voor EGFR beperkt tot het MU gedeelte (Figuur 3D, E en F), terwijl alleen het SM gedeelte vertoonden sterke immunoreactiviteit voor CTTN (figuur 3G, H en I). Deze resultaten suggereren dat, in geval 4, de tumorcellen met 7p versterking in MU kon niet de SM binnengevallen, terwijl die met chromosoom 11 gain kan het SM zijn binnengedrongen. Vervolgens analyseerden we genomische clonaliteit en heterogeniteit in de MU en SM van andere zaken. Onder de andere 22 gevallen 14 vertoonden een vergelijkbaar patroon van genomische aberratie in de MU en SM (figuur S1 (6 gevallen) en S2 (8 gevallen)), wat suggereert dat de kankercellen in de MU en SM van deze gevallen waren klonaal verwante . Interessant is dat 12 van de 14 gevallen toonde een significant verschil in de genomische profiel patronen tussen MU en SM (figuur S1 (6 gevallen) en S2 (6 gevallen)), wat suggereert dat deze gevallen werden ook uit genetisch heterogene subpopulaties. Genomisch klonaliteit en ongelijkmatig primaire (SM) en metastatische (LN) porties SMGC Vervolgens de betrokkenheid van CNAs bezig lymfekliermetastase van vroege maagkanker onderzoeken, vergeleken we het aantal van CNAs tussen gepaarde primaire (SM) en metastatische (LN) porties 9 SMGCS (Figuur 4A). Drie van de 9 gevallen toonde een toegenomen aantal CNAs in de LN gedeelte, terwijl de resterende 6 gevallen een daling (figuur 4A). Dientengevolge was er geen significant verschil in het aantal CNAs tussen de gepaarde SM en LN delen (figuur 4A, niet significant gepaarde t-test). Teneinde voorts CNA specifiek aan LN metastase te identificeren, hebben we de gemiddelde frequentie van CNA in SM met die in de gekoppelde LN gedeelte (figuur 4B), maar konden recht komen. Om het verschil te onderzoeken CNA's tussen SM en LN van dezelfde tumor, vergeleken we de genomische profielen van gepaarde SM en LN monsters in elk geval. Een representatief geval is getoond in figuur 4C, D en E. De gepaarde SM en LN monsters deelden een vergelijkbaar patroon van genomische aberratie in chromosoom 8 (Figuur 4D), hetgeen suggereert dat beide gedeelten zijn afgeleid van dezelfde kloon. Echter, winst van chromosoom 14 werd alleen waargenomen in SM, maar niet in LN (figuur 4E). Deze resultaten suggereerden dat de tumorcellen in de SM-Nul gedeelten van het onderhavige geval klonaal verwante, maar uit genetisch heterogene subpopulaties. Ook geanalyseerd genomische klonaliteit en ongelijkmatig SM en LN delen uit andere gevallen. Onder de overige 8 gevallen, 5 vertoonden een vergelijkbaar patroon van genomische aberratie in zowel SM en LN (figuur S3), wat suggereert dat de gepaarde SM en LN gedeelten van deze gevallen waren klonaal verwante. Verder 4 van de 5 gevallen toonde een significant verschil in de genomische profiel patronen tussen SM en LN (figuur S3), wat suggereert dat deze gevallen werden ook uit genetisch heterogene subpopulaties. aangezien geen statistisch significante verschillen in de frequenties van CNAs tussen gepaarde SM en LN gedeelten (Figuur 4B) werden gedetecteerd veronderstelden we dat subpopulaties uitvoering metastase gerelateerde CNA net zo goed in de SM worden als LN deel van metastatische SMGC. Daarom hebben we naast vergeleken de frequenties van CNA's in de SM gedeelte van uitgezaaide SMGCS (12 gevallen) met die van niet-uitgezaaide SMGCS (15 gevallen), en vond dat winsten op 11q13, 11q14, 11q22 en 14q32 vaker werden ontdekt in metastatische SMGCS dan bij niet-metastatische SMGCS (Figuur 5A en Tabel 2). We vergeleken ook de frequenties van high-level kopie-aantal afwijkingen, zoals versterking en verwijdering tussen de twee groepen, en vond dat amplificatie van 17q21 werd vaker waargenomen bij uitgezaaide SMGCS dan bij niet-uitgezaaide SMGCS (tabel 3 en tabel S1) . Deze resultaten suggereerden dat winst op 11q13, 11q14, 11q22, 14q32 en 17q21 amplificatie bij zijn betrokken bij de LN metastase van SMGCS. De kleinste gemene regio amplificatie op 17q21 die 5 genen in tabel 3 opgesomde Sinds ERBB2 een bekende oncogen [26], [27], [28], werd opgenomen in de lijst, voerden we immunohistochemische analyse van ErbB2 overexpressie in alle 27 gevallen. Zoals getoond in figuur 5B, gevallen 17q21 amplificatie vertoonden sterke kleuring ErbB2 in SM, terwijl een geval zonder versterking niet. Voorts werd overexpressie van ErbB2 significant geassocieerd met 17q21 amplificatie (Tabel 4), wat suggereert dat ERBB2 amplificatie en overexpressie kan worden betrokken bij de LN metastase van een deel van SMGCS. Het is wijd aanvaard dat een tumor ontstaat uit een enkele cel. Echter, hoe het zich ontwikkelt tot een vergevorderd stadium wordt nog steeds gedebatteerd. Vroege studies van colorectale en pancreaskanker geleid tot een begrip dat de ontwikkeling en progressie van deze kankers zijn geassocieerd met accumulatie van chromosomale afwijkingen, aangeduid als het model multistep tumorigenese [29], [30]. Zo worden genomische afwijkingen van de APC, KRAS, SMAD4 TP53 en genen betrokken bij de adenoom-carcinoom-sequentie in het colon [29]. Dergelijke studies gericht op slechts een deel van tumor-gerelateerde genen en verwaarloosd de rol van de meeste andere genen. Bovendien was dit model niet de betekenis van intratumorale genomische heterogeniteit voor tumorontwikkeling en progressie evalueren. Inmiddels hebben recente studies hebben geleid tot de oprichting van een ander model, aangeduid als de klonale evolutie model [7], [9], [10]. In dit model, een enkele kloon ontwikkelt tot een aantal verschillende subpopulaties door de accumulatie van verschillende genetische afwijkingen. De overheersende populatie worden vervangen door afzonderlijke subpopulaties binnen een tumormassa door het effect van milieu- selectiedruk en /of het stadium van tumorprogressie. Bijgevolg kunnen verscheidene genetisch heterogene celpopulaties binnen eenzelfde enkele tumormassa. Bewijs van intratumorale genetische heterogeniteit gekoppeld aan klonale ontwikkeling is verkregen voor verschillende vaste tumoren, met inbegrip van prostaatkanker [14], barrettoesofagus [31], ovariale kanker [32], [33], cervicale kanker [34], borstkanker [15], [35], neuroblastoma [36], pancreaskanker [13], [37], en colorectale kanker [38]. Interessant is dat in een studie van dodelijke metastatische prostaatkanker, geen CNA specifiek op de plaats van metastase gevonden [14]. Op dezelfde manier, in een studie van high-grade sereus ovariumcarcinoom, was er geen bewijs voor een verband tussen de verwerving van cisplatine weerstand en specifieke CNA's [39]. Deze resultaten suggereren dat de meerstaps tumorigenese model, waarin bepaalde afwijkingen spelen een belangrijke rol bij tumorontwikkeling en progressie, niet altijd de wijze waarop tumoren verwerven hun kwaadaardig karakter ervan. In de onderhavige studie hebben we de hypothese dat aanvankelijk aanschaf van specifiek CNA (s) van belang voor submucosale invasie kunnen zijn. Maar we waren niet in staat om CNA's die in SM vaker waren dan in de gekoppelde MU monster te vinden. Daarnaast zagen we ook geen significant verschil wat betreft het aantal CNAs in de gekoppelde MU en SM porties. Echter, we vonden dat de meeste SMGCS bestonden uit klonaal verwante, maar genetisch verschillende subpopulaties, wat suggereert dat klonale evolutie kan optreden tijdens de progressie van maagkanker. Samengevat, hoewel het aantal gevallen beperkt onderzochte onze bevindingen suggereerden dat de verzameling van genetisch verschillende subpopulaties plaats van aanschaf van specifiek CNAs in de MU deel belangrijk voor de werkwijze van submucosale invasie kan zijn. Op basis van deze bevindingen, stellen we een hypothetisch model voor de werkwijze SM invasie en metastase LN van vroege maagkanker (figuur 6). Om deze hypothese te bevestigen, zullen verdere studies met grotere steekproeven vereist. Onze gegevens geven dat SMGCS bestaan uit genetisch heterogene subpopulaties zijn belangrijk in de context van maagkanker onderzoek en behandeling aangezien tumor heterogeniteit maakt de ontwikkeling van doeltreffende geneesmiddelen moeilijk. Aangezien genomische CNA's hebben invloed op genexpressieprofielen bij verschillende kankersoorten [16], [21], [40], [41], [42], [43], is het mogelijk dat elk van de genetisch onderscheiden subpopulaties binnen één tumor kunnen verschillen in zowel biologisch gedrag en de reactie op drugs, met inbegrip van moleculaire richtende middelen tegen kanker. Cooke et al. dat verduidelijking van verschillende genetische subpopulaties voorgesteld binnen een tumor zou effectieve therapie mogelijk onder toepassing van een specifiek middel gericht op een gemeenschappelijke genomische aberratie of gecombineerde middelen gericht op unieke genomische afwijkingen in elk van de verschillende subpopulaties [39]. Deze strategie kan ook voor de behandeling van maagkanker. Van de 23 gevallen hebben geanalyseerd, 15 toonde een klonale relatie tussen MU en SM porties. Verder 13 van deze 15 gevallen bleek ook verschillen in CNA tussen de twee regio's, wat suggereert dat klonale evolutie vaak voorkomt in de vroege fase van maagkanker. De relatie tussen de gepaarde MU en SM monsters in de overige 8 gevallen zonder gemeenschappelijke CNA's onduidelijk. Twee mogelijke verklaringen voor dit kan worden voorgesteld. Een daarvan is dat tumoren in de gekoppelde gedeelten, die niet gebruikelijk CNA had ontwikkeld onafhankelijk. De andere is dat de gekoppelde gedeelten gedeelde andere vormen van genetische afwijkingen, zoals mutaties en translocaties, die door array CGH kan worden gedetecteerd. In het laatste geval zou next-generation sequencing bruikbaar voor de analyse van dergelijke relaties zijn. In deze studie, winsten op 11q13, 11q14, 11q22 en 14q32, en versterking op 17q21, kwamen vaker voor bij het SM gedeelte van uitgezaaide SMGCS dan in die van niet-uitgezaaide SMGCS. Interessant is dat winsten op 11q13 en 14q32 naar verluidt betrokken bij levermetastasen van darmkanker [38]. Derhalve suggereren deze gegevens dat versterking bij 11q13 en 14q32 betrokken kan zijn bij de metastase van maagdarmkanker. Chromosoom 17q21 herbergt een krachtige oncogen, ERBB2. Associatie van ErbB2-expressie met de klinische en pathologische kenmerken van maagkanker is onderzocht in verscheidene studies [44], [45], [46], [47], [48], [49]. De invloed van ErbB2 overexpressie LN metastase verschillen tussen deze studies [44], [46], [47]. In deze studie, ondanks het beperkte aantal SMGCS onderzochte al die met ERBB2 amplificatie en overexpressie vertoonden lymfeklier. Nader onderzoek met behulp van een groter aantal SMGCS vereist zal zijn om de betekenis van deze tendens te evalueren. Dankwoord Wij danken Misuzu Taguchi, Yoko Miyanari en Tsuyoshi Iwao voor hun uitstekende technische ondersteuning
. ≪ 0,05 werden beschouwd als statistisch significant
Vergelijking van genomische profielen tussen metastatische en niet-metastatische SMGC
Discussie
Ondersteunende informatie
Figuur S1.
Cases toont zowel de gemeenschappelijke en de verschillende genomische afwijkingen tussen de MU en SM porties. De linker panelen tonen voorkomende patronen van genomische afwijkingen in MU SM en voor elk geval. Het midden en rechts panelen tonen verschillende patronen van genomische aberratie tussen de twee delen telkens
doi:. 10.1371 /journal.pone.0022313.s001
(TIF)
Figuur S2.
Cases toont zowel de gemeenschappelijke en de verschillende genomische afwijkingen tussen de MU en SM porties. Gemeenschappelijke en verschillende patronen van genomische aberratie tussen MU en SM voor elk geval worden getoond
doi:. 10.1371 /journal.pone.0022313.s002
(TIF)
Figuur S3.
Cases toont zowel de gemeenschappelijke en de verschillende genomische afwijkingen tussen de SM en LN porties. De linker panelen tonen gemeenschappelijke patronen van genomische aberratie tussen SM en LN voor elk geval. Het midden en rechts panelen tonen verschillende patronen van genomische aberratie tussen de twee delen telkens
doi:. 10.1371 /journal.pone.0022313.s003
(TIF)
Tabel S1.
Terugkerende amplificaties en deleties in SMGCS.
doi:. 10.1371 /journal.pone.0022313.s004
(DOC)
 Longmicrobioom voorspelt ernst van de ziekte van COVID-19
Longmicrobioom voorspelt ernst van de ziekte van COVID-19
 Goed nieuws voor IBS-patiënten, aangezien onderzoekers "darm jeuk" identificeren
Goed nieuws voor IBS-patiënten, aangezien onderzoekers "darm jeuk" identificeren
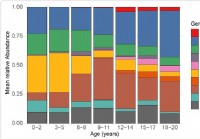 Verandering van het microbioom van de bovenste luchtwegen bij kinderen in verband met gevoeligheid voor SARS-CoV-2
Verandering van het microbioom van de bovenste luchtwegen bij kinderen in verband met gevoeligheid voor SARS-CoV-2
 Onderzoekers hopen dat een bloedtest die fibromyalgie nauwkeurig diagnosticeert, binnen vijf jaar beschikbaar kan zijn
Onderzoekers hopen dat een bloedtest die fibromyalgie nauwkeurig diagnosticeert, binnen vijf jaar beschikbaar kan zijn
 Wetenschapper bewijst rol van microbioom bij obesitas
Wetenschapper bewijst rol van microbioom bij obesitas
 Nieuwe studie kan dodelijke infecties bij baby's helpen voorkomen
Nieuwe studie kan dodelijke infecties bij baby's helpen voorkomen
 Onderzoek toont aan dat probiotica angst en depressie kunnen helpen bestrijden
Eerder onderzoek heeft psychische problemen en ontwikkelingsstoornissen gekoppeld aan de darmgezondheid. Nutsvoorzieningen, een team van Britse wetenschappers heeft aangetoond dat voedingsmiddelen die
Onderzoek toont aan dat probiotica angst en depressie kunnen helpen bestrijden
Eerder onderzoek heeft psychische problemen en ontwikkelingsstoornissen gekoppeld aan de darmgezondheid. Nutsvoorzieningen, een team van Britse wetenschappers heeft aangetoond dat voedingsmiddelen die
 Wat u eet, kan de manier veranderen waarop antibiotica uw darmen beïnvloeden
Een nieuwe studie door onderzoekers van de Brown University in Rhode Island heeft aangetoond dat voeding van invloed kan zijn op hoe het darmmicrobioom wordt beïnvloed door behandeling met antibiotica
Wat u eet, kan de manier veranderen waarop antibiotica uw darmen beïnvloeden
Een nieuwe studie door onderzoekers van de Brown University in Rhode Island heeft aangetoond dat voeding van invloed kan zijn op hoe het darmmicrobioom wordt beïnvloed door behandeling met antibiotica
 Hoe u uw immuunsysteem kunt versterken om het coronavirus te bestrijden
Terwijl de wereld worstelt met de pandemie van het coronavirus, steeds meer mensen proberen zich met alle mogelijke middelen tegen het virus te beschermen. Er zijn momenteel geen vaccins tegen het ern
Hoe u uw immuunsysteem kunt versterken om het coronavirus te bestrijden
Terwijl de wereld worstelt met de pandemie van het coronavirus, steeds meer mensen proberen zich met alle mogelijke middelen tegen het virus te beschermen. Er zijn momenteel geen vaccins tegen het ern